Lichtgestuurde fermentaties voor microbiële chemische en eiwitproductie
Summary
Optogenetische controle van microbieel metabolisme biedt flexibele dynamische controle over fermentatieprocessen. Het protocol hier laat zien hoe blauw licht-gereguleerde fermentaties voor chemische en eiwitproductie op verschillende volumetrische schalen kunnen worden opgezet.
Abstract
Microbiële celfabrieken bieden een duurzaam alternatief voor het produceren van chemicaliën en recombinante eiwitten uit hernieuwbare grondstoffen. Het overbelasten van een micro-organisme met genetische modificaties kan echter de fitheid en productiviteit van de gastheer verminderen. Dit probleem kan worden opgelost door dynamische controle te gebruiken: induceerbare expressie van enzymen en routes, meestal met behulp van chemische of op voedingsstoffen gebaseerde additieven, om cellulaire groei en productie in evenwicht te brengen. Optogenetica biedt een niet-invasieve, zeer afstembare en omkeerbare methode voor het dynamisch reguleren van genexpressie. Hier beschrijven we hoe lichtgecontroleerde fermentaties van gemanipuleerde Escherichia coli pt Saccharomyces cerevisiae kunnen worden opgezet voor de productie van chemicaliën of recombinante eiwitten. We bespreken hoe licht op geselecteerde tijden en doseringen kan worden toegepast om microbiële groei en productie te ontkoppelen voor verbeterde fermentatiecontrole en productiviteit, evenals de belangrijkste optimalisatie-overwegingen voor de beste resultaten. Daarnaast beschrijven we hoe lichtregelaars kunnen worden geïmplementeerd voor bioreactorexperimenten op laboratoriumschaal. Deze protocollen vergemakkelijken de toepassing van optogenetische controles in gemanipuleerde micro-organismen voor verbeterde fermentatieprestaties.
Introduction
Optogenetica, de controle van biologische processen met lichtgevoelige eiwitten, biedt een nieuwe strategie om microbiële fermentaties dynamisch te beheersen voor chemische en eiwitproductie1,2. De belasting van gemanipuleerde metabole routes en de toxiciteit van sommige tussenproducten en producten schaadt vaak de celgroei3. Dergelijke spanningen kunnen leiden tot een slechte accumulatie van biomassa en verminderde productiviteit3. Deze uitdaging kan worden aangepakt door fermentaties tijdelijk te verdelen in een groei- en productiefase, die metabolische middelen besteden aan respectievelijk biomassaaccumulatie of productsynthese4. We hebben onlangs aangetoond dat de overgang van groei naar productie in deze tweefasige gisting kan worden geïnduceerd met veranderingen in verlichtingsomstandigheden5,6,7. De hoge tonijnbaarheid, omkeerbaarheid en orthogonaliteit van lichtinputs8 bieden unieke voordelen voor lichtgecontroleerde fermentaties die moeilijk of onmogelijk te repliceren zijn met chemische inductoren die worden gebruikt bij dynamische controle van conventionele tweefasige fermentaties4,9,10,11.
Het blauw-licht responsieve EL222-eiwit afgeleid van Erythrobacter litoralis is gebruikt om verschillende optogenetische circuits te ontwikkelen voor metabole engineering in Saccharomyces cerevisiae5,7,12,13. EL222 bevat een licht-zuurstof-spanningssensor (LOV) domein dat een conformatieverschuiving ondergaat bij activering van blauw licht (465 nm), waardoor het kan binden aan zijn verwante DNA-sequentie (C120) 13. Het fuseren van EL222 met het virale VP16-activeringsdomein (VP16-EL222) resulteert in een blauw-licht responsieve transcriptiefactor die reversibel genexpressie in S. cerevisiae7 en andere organismen14 van de synthetische promotor PC120 kan activeren. Verschillende circuits op basis van EL222 zijn ontwikkeld en gebruikt voor chemische productie in S. cerevisiae, zoals het basis lichtgeactiveerde OptoEXP-systeem7, waarin het gen van belang direct tot expressie komt uit PC120 (figuur 1A). Bezorgdheid over lichtpenetratie bij de hoge celdichtheden die typisch zijn in de productiefase van fermentaties motiveerde ons echter om omgekeerde circuits te ontwikkelen die in het donker worden geïnduceerd, zoals de OptoINVRT- en OptoQ-INVRT-circuits (figuur 1B) 5,7,13. Deze systemen maken gebruik van de galactose (GAL) of quinic acid (Q) regulons van respectievelijk S. cerevisiae pt N. crassa, en regelen hun overeenkomstige repressoren (GAL80 en QS) met VP16-EL222, om genexpressie in het licht te onderdrukken en sterk te induceren in het donker. De combinatie van OptoEXP- en OptoINVRT-circuits resulteert in bidirectionele controle van genexpressie, waardoor tweefasige fermentaties mogelijk zijn waarbij de groeifase wordt geïnduceerd met blauw licht en de productiefase met duisternis (figuur 2A)5,7.
Het gebruik van licht in plaats van duisternis om genexpressie tijdens de productiefase te induceren, zou de mogelijkheden van optogenetische controles aanzienlijk uitbreiden, maar zou ook het overwinnen van de lichtpenetratiebeperkingen van de hoge celdichtheden vereisen die typisch worden aangetroffen in deze fase van fermentatie. Hiertoe hebben we circuits ontwikkeld, bekend als OptoAMP en OptoQ-AMP, die de transcriptionele respons op stimulatie van blauw licht versterken. Deze circuits gebruiken wild-type of overgevoelige mutanten van VP16-EL222 om de productie van de transcriptionele activatoren Gal4p of QF2 van respectievelijk de GAL- of Q-regulons te regelen, waardoor een verhoogde gevoeligheid en sterkere genexpressie met licht12,13 wordt bereikt (figuur 1C). OptoAMP-circuits kunnen volledige en homogene lichtinductie bereiken in 5 L bioreactoren met een optische dichtheid (gemeten bij 600 nm; OD600) waarden van ten minste 40 met slechts ~ 0,35% verlichting (5% lichtdosis op slechts ~ 7% van het bulkoppervlak). Dit toont een hogere mate van gevoeligheid in vergelijking met OptoEXP, die bijna 100% verlichting vereist12. Het vermogen om effectief genexpressie te induceren met licht bij hoge celdichtheden opent nieuwe mogelijkheden voor dynamische controle van fermentaties. Dit omvat operationele fermentaties in meer dan twee temporele fasen, zoals driefasige fermentaties, waarin groei-, inductie- en productiefasen worden vastgesteld met unieke lichtschema’s om de chemische productie te optimaliseren (figuur 2B)12.
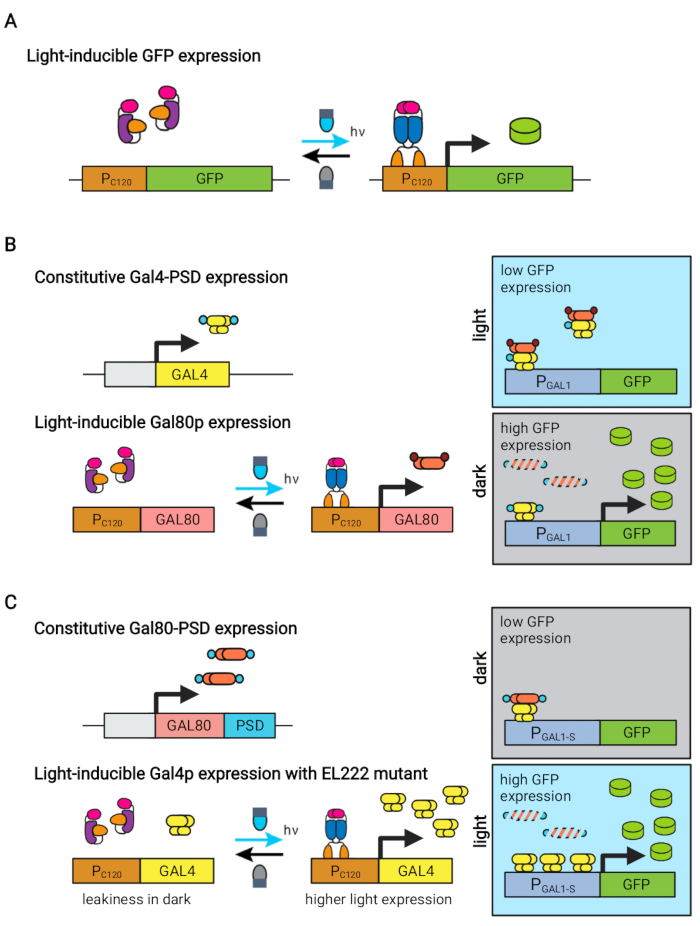
Figuur 1: Optogenetische circuits voor dynamische controle van S. cerevisiae. De OptoEXP-, OptoINVRT- en OptoAMP-circuits zijn gebaseerd op het lichtgevoelige VP16-EL222-systeem. (A) In het OptoEXP-circuit veroorzaakt blootstelling aan blauw licht een conformatieverandering en dimerisatie van VP16-EL222, die een DNA-bindend domein blootlegt en transcriptie van PC120 mogelijk maakt. De figuur is aangepast van Zhao et al.7. (B) OptoINVRT-circuits maken gebruik van de GAL (afgebeelde) of Q-regulons om expressie in het donker te induceren. In GAL-gebaseerde circuits worden VP16-EL222 en GAL4 constitutief uitgedrukt, terwijl PC120-aandrijvingen de expressie van de GAL80-onderdrukker (in Q-gebaseerde circuits worden GAL4 pt GAL80 vervangen door respectievelijk QF2 en QS, en een synthetische QUAS-bevattende promotor wordt gebruikt in plaats van een GAL-promotor). Bij licht voorkomt Gal80p activering van het interessante gen van PGAL1. In het donker wordt GAL80 niet tot expressie gebracht en snel gedegradeerd door het samen te smelten tot een constitutief degron-domein (klein bruin domein), wat activering van PGAL1 door Gal4p mogelijk maakt. De figuur is aangepast van Zhao et al.5. (C) OptoAMP-circuits gebruiken ook VP16-EL222 om de GAL (afgebeeld) of Q-regulons te regelen. In deze circuits wordt de GAL80-repressor (of QS) constitutief uitgedrukt en versmolten tot een fotogevoelig degron (klein blauw domein) voor strakke repressie in het donker. PC120 en een overgevoelige VP16-EL222 mutante controle expressie van GAL4 (of QF2) met licht, dat PGAL1 (of een QUAS-bevattende promotor) sterk activeert in het licht. GAL-afgeleide circuits kunnen gebruik maken van gemanipuleerde vormen van PGAL1, zoals PGAL1-M of PGAL1-S, die een verhoogde activiteit hebben, evenals wild-type promotors gecontroleerd door de GAL regulon (PGAL1, PGAL10, PGAL2, PGAL7). De figuur is aangepast van Zhao et al.12. Klik hier om een grotere versie van deze figuur te bekijken.
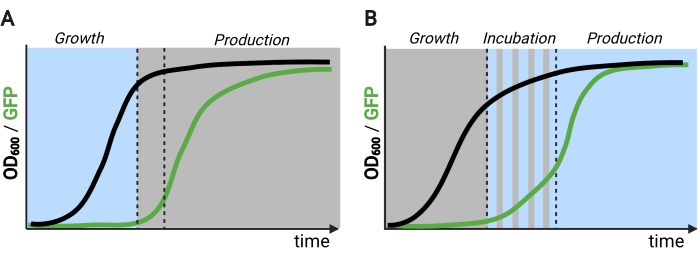
Figuur 2: Twee- en driefasige fermentaties door de tijd heen. (A) Tweefasige fermentaties met omgekeerde circuits bestaan uit een lichtgedreven groeifase en een donkere productiefase. In de groeifase hoopt biomassa zich op naarmate de productieroute onderdrukt blijft. Bij het bereiken van de gewenste OD600 worden cellen naar het donker verplaatst om zich metabolisch aan te passen voordat ze opnieuw worden besteed aan verse media voor de productiefase. (B) In een driefasig proces worden de groei-, incubatie- en productiefasen gedefinieerd door unieke lichtschema’s, die kunnen bestaan uit een donkere groeiperiode, gepulseerde incubatie en volledig verlichte productiefase. Figuur gemaakt met Biorender. Klik hier om een grotere versie van deze figuur te bekijken.
Optogenetische circuits zijn ook ontwikkeld voor dynamische controle van chemische en eiwitproductie in E. coli. OptoLAC-circuits regelen de bacteriële LacI-repressor met behulp van het lichtgevoelige pDawn-circuit, dat is gebaseerd op het YF1 / FixJ-tweecomponentensysteem6 (figuur 3). Net als OptoINVRT5 zijn OptoLAC-circuits ontworpen om genexpressie in het licht te onderdrukken en in het donker te induceren. Expressieniveaus met behulp van OptoLAC-circuits kunnen overeenkomen met of hoger zijn dan die bereikt met standaard isopropyl-β-d-1-thiogalactopyranoside (IPTG) inductie, waardoor de sterkte van chemische inductie behouden blijft en tegelijkertijd verbeterde tonijnbaarheid en omkeerbaarheid wordt geboden6. Daarom maken OptoLAC-circuits effectieve optogenetische controle mogelijk voor metabole engineering in E. coli.
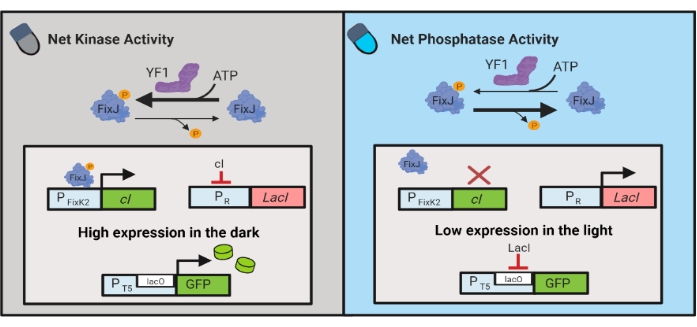
Figuur 3: OptoLAC-circuits voor dynamische controle van E. coli. De OptoLAC-circuits passen het pDawn-systeem en het lac operon aan om activering in het donker en onderdrukking in het licht te bereiken. In het donker fosforyleert YF1 FixJ, dat vervolgens de PFixK2-promotor activeert om de cI-repressor tot expressie te brengen. De cI-repressor voorkomt expressie van de lacI-repressor van de PR-promotor , waardoor transcriptie van het betreffende gen van een lacO-bevattende promotor mogelijk is. Omgekeerd vermindert blauw licht de netto kinaseactiviteit van YF1, waardoor FixJ-fosforylering en dus cI-expressie wordt omgedraaid, wat de expressie van lacI onderdrukt en expressie van de lacO-bevattende promotor voorkomt. Het cijfer is aangepast van Lalwani et al.6. Klik hier om een grotere versie van deze figuur te bekijken.
We beschrijven hier de basisprotocollen voor lichtgecontroleerde fermentaties van S. cerevisiae pt E. coli voor chemische of eiwitproductie. Voor zowel gist als bacteriën richten we ons eerst op fermentaties met een lichtgedreven groeifase en een door duisternis geïnduceerde productiefase mogelijk gemaakt door OptoINVRT- en OptoLAC-circuits. Vervolgens beschrijven we een protocol voor een driefasige (groei, inductie, productie) lichtgestuurde fermentatie mogelijk gemaakt door OptoAMP-circuits. Verder beschrijven we hoe we optogenetisch gecontroleerde fermentaties kunnen opschalen van microplaten naar bioreactoren op laboratoriumschaal. Met dit protocol willen we een complete en gemakkelijk reproduceerbare gids bieden voor het uitvoeren van lichtgestuurde fermentaties voor chemische of eiwitproductie.
Protocol
Representative Results
Discussion
Dynamische controle wordt al lang toegepast om de opbrengsten voor metabole engineering en recombinante eiwitproductie te verbeteren4. Verschuivingen in enzymatische expressie worden meestal geïmplementeerd met behulp van chemische inductoren zoals IPTG21, galactose22 en tetracycline23, maar zijn ook gemedieerd met behulp van procesomstandigheden zoals temperatuur en pH. Optogenetische controle van genexpressie elimineert de …
Declarações
The authors have nothing to disclose.
Acknowledgements
Dit onderzoek werd ondersteund door het Amerikaanse ministerie van Energie, Office of Science, Office of Biological and Environmental Research Award Number DE-SC0019363, de NSF CAREER Award CBET-1751840, The Pew Charitable Trusts en de Camille Dreyfus Teacher-Scholar Award.
Materials
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
Referências
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. Bioengenharia. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).

