미생물 화학 및 단백질 생산을 위한 광 조절 발효
Summary
미생물 대사의 광유전학적 조절은 발효 과정에 대한 유연한 동적 제어를 제공합니다. 여기의 프로토콜은 다양한 부피 규모에서 화학 및 단백질 생산을위한 청색광 조절 발효를 설정하는 방법을 보여줍니다.
Abstract
미생물 세포 공장은 재생 가능한 공급 원료에서 화학 물질 및 재조합 단백질을 생산하기위한 지속 가능한 대안을 제공합니다. 그러나 유전자 변형으로 미생물에게 과도한 부담을 주면 숙주 적합성과 생산성이 떨어질 수 있습니다. 이 문제는 동적 조절을 사용하여 극복 할 수 있습니다 : 효소 및 경로의 유도 가능한 발현, 일반적으로 화학 물질 또는 영양소 기반 첨가제를 사용하여 세포 성장과 생산의 균형을 유지합니다. 광유전학은 유전자 발현을 동적으로 조절하는 비침습적이고, 고도로 조정가능하며, 가역적인 방법을 제공한다. 여기에서는 화학 물질 또는 재조합 단백질 생산을 위해 조작 된 에스케리치아 콜리 및 사카로 마이스 세레 비시아 (Saccharomyces cerevisiae )의 광 조절 발효를 설정하는 방법을 설명합니다. 우리는 발효 제어 및 생산성 향상을 위해 미생물 성장과 생산을 분리하기 위해 선택된 시간과 복용량에 빛을 적용하는 방법과 최상의 결과를 위한 주요 최적화 고려 사항에 대해 논의합니다. 또한, 우리는 실험실 규모의 생물 반응기 실험을위한 조명 제어를 구현하는 방법을 설명합니다. 이러한 프로토콜은 향상된 발효 성능을 위해 조작된 미생물에서 광유전학적 제어의 채택을 용이하게 합니다.
Introduction
광유전학은 광반응 단백질로 생물학적 과정을 제어하며, 화학 및 단백질 생산을 위한 미생물 발효를 동적으로 제어하는 새로운 전략을 제공합니다1,2. 조작된 대사 경로의 부담과 일부 중간체 및 제품의 독성은 종종 세포 성장을 손상시킵니다3. 이러한 스트레스는 바이오매스 축적을 저하시키고 생산성을 저하시킬 수 있습니다3. 이 과제는 발효를 일시적으로 성장 및 생산 단계로 나눠서 해결할 수 있으며, 이는 각각 바이오 매스 축적 또는 제품 합성에 대사 자원을 바칩니다4. 우리는 최근에이 두 단계 발효에서 성장에서 생산으로의 전환이 조명 조건의 변화에 따라 유도 될 수 있음을 보여주었습니다5,6,7. 광 입력의 높은 튜닝성, 가역성 및 직교성8은 기존의 2상 발효의 동적 제어에 사용되는 화학 유도제와 복제하기가 어렵거나 불가능한 광 제어 발효에 고유한 이점을 제공합니다4,9,10,11.
Erythrobacter litoralis에서 유래 한 청색광 반응 EL222 단백질은 Saccharomyces cerevisiae5,7,12,13에서 대사 공학을위한 여러 광유전학 회로를 개발하는 데 사용되었습니다. EL222는 청색광 활성화(465nm)시 형태적 이동을 겪는 광산소-전압 센서(LOV) 도메인을 함유하고 있으며, 이를 통해 동족 DNA 서열(C120)13에 결합할 수 있습니다. EL222를 바이러스 VP16 활성화 도메인 (VP16-EL222)에 융합시키는 것은 합성 프로모터 PC120으로부터 S. cerevisiae7 및 다른 유기체14에서 유전자 발현을 가역적으로 활성화시킬 수 있는 청색광 반응성 전사 인자를 초래한다. EL222에 기초한 몇몇 회로가 개발되어 PC120으로부터 관심있는 유전자가 직접 발현되는 기본 광활성화 OptoEXP 시스템7과 같은 S. cerevisiae의 화학 생산에 사용되었다(도 1A). 그러나 발효의 생산 단계에서 일반적으로 발생하는 높은 세포 밀도에서의 광 침투에 대한 우려는 OptoINVRT 및 OptoQ-INVRT 회로와 같은 어둠 속에서 유도되는 반전 회로를 개발하도록 동기를 부여했습니다 (그림 1B) 5,7,13. 이들 시스템은 각각 S. cerevisiae 및 N. crassa로부터의 갈락토오스 (GAL) 또는 퀸산 (Q) 조절을 이용하여, VP16-EL222로 그들의 상응하는 억제기 (GAL80 및 QS)를 제어하여, 빛에서 유전자 발현을 억제하고 어둠 속에서 강하게 유도한다. OptoEXP와 OptoINVRT 회로를 결합하면 유전자 발현의 양방향 제어가 가능하여 성장 단계가 청색광으로 유도되는 2 상 발효가 가능하고 어둠이있는 생산 단계 (그림 2A)5,7.
생산 단계에서 유전자 발현을 유도하기 위해 어둠 대신 빛을 사용하는 것은 광유전학적 조절의 능력을 크게 확장시킬 것이지만, 또한 발효의 이 단계에서 전형적으로 직면하는 높은 세포 밀도의 광 침투 한계를 극복할 필요가 있을 것이다. 이를 위해 우리는 청색광 자극에 대한 전사 반응을 증폭시키는 OptoAMP 및 OptoQ-AMP로 알려진 회로를 개발했습니다. 이들 회로는 VP16-EL222의 야생형 또는 과민성 돌연변이체를 사용하여 각각 GAL 또는 Q 레귤론의 전사 활성화제 Gal4p 또는 QF2의 생산을 조절하여, light12,13으로 향상된 감도와 더 강한 유전자 발현을 달성한다(도 1C). OptoAMP 회로는 광학 밀도(600nm에서 측정; OD600) 값은 조명의 ~0.35%만을 갖는 적어도 40의 값(벌크 표면의 ~7%에서만 5% 광량). 이는 100%에 가까운 조명이 필요한 OptoEXP에 비해 더 높은 수준의 감도를 보여줍니다12. 높은 세포 밀도에서 빛으로 유전자 발현을 효과적으로 유도하는 능력은 발효의 역동적 인 조절을위한 새로운 기회를 열어줍니다. 여기에는 화학 생산을 최적화하기 위해 고유한 조명 일정으로 성장, 유도 및 생산 단계가 설정되는 삼상 발효와 같은 두 개 이상의 시간적 단계에서 발효를 운영하는 것이 포함됩니다(그림 2B)12.
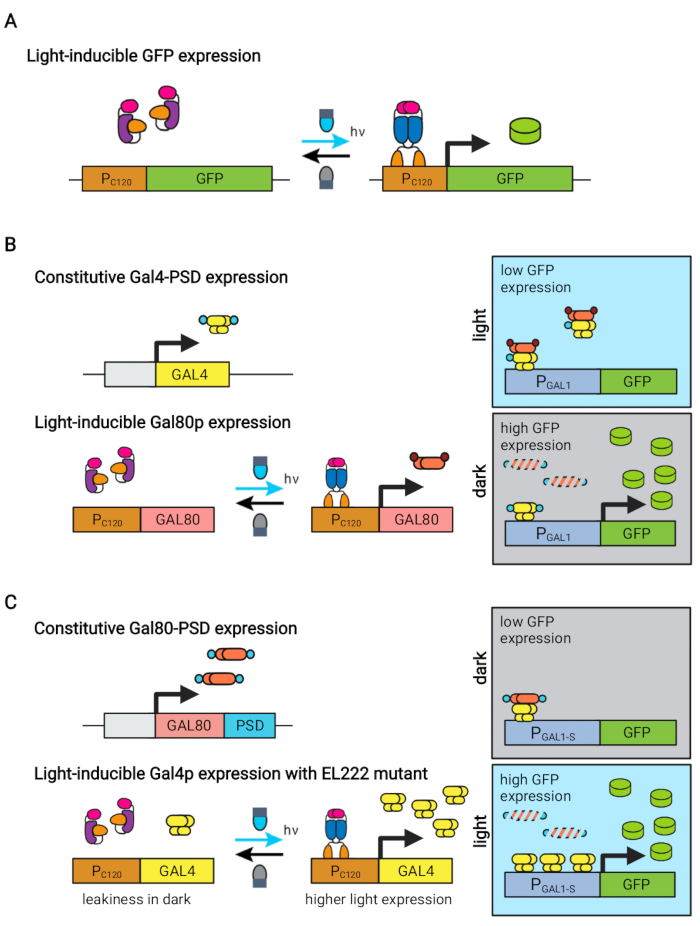
그림 1 : S. cerevisiae의 동적 제어를위한 광유전학 회로. OptoEXP, OptoINVRT 및 OptoAMP 회로는 빛에 민감한 VP16-EL222 시스템을 기반으로 합니다. (A) OptoEXP 회로에서, 청색광에 노출되면 VP16-EL222의 형태적 변화 및 이량체화가 일어나며, 이는 DNA 결합 도메인을 노출시키고 PC120으로부터의 전사를 허용한다. 그림은 Zhao et al.7에서 수정되었습니다. (b) OptoINVRT 회로는 어둠 속에서 발현을 유도하기 위해 GAL (도시) 또는 Q 레귤론을 이용한다. GAL 기반 회로에서 VP16-EL222 및 GAL4는 구성적으로 발현되는 반면, PC120은 GAL80 리프레서의 발현을 구동합니다 (Q 기반 회로에서는 GAL4 및 GAL80이 각각 QF2 및 QS 로 대체되고 GAL 프로모터 대신 합성 QUAS 함유 프로모터가 사용됩니다). 빛에서, Gal80p는 PGAL1로부터 관심있는 유전자의 활성화를 방지한다. 어둠 속에서, GAL80은 발현되지 않고 Gal4p에 의한 PGAL1의 활성화를 허용하는 구성적 데그론 도메인 (작은 갈색 도메인)에 융합함으로써 빠르게 분해된다. 그림은 Zhao et al.5에서 수정되었습니다. (C) OptoAMP 회로는 또한 VP16-EL222를 사용하여 GAL (그림) 또는 Q 레귤론을 제어합니다. 이 회로에서 GAL80 리프레서 (또는 QS)는 구성적으로 표현되고 감광성 데그론 (작은 파란색 도메인)에 융합되어 어둠 속에서 엄격한 억압을 보장합니다. PC120 및 과민성 VP16-EL222 돌연변이체는 빛과 함께 GAL4 (또는 QF2)의 발현을 조절하며, 이는 광에서 PGAL1 (또는 QUAS 함유 프로모터)을 강하게 활성화시킨다. GAL에서 유래한 회로는 활성이 증가한 PGAL1-M 또는 PGAL1-S와 같은 조작된 형태의 PGAL1뿐만 아니라 GAL 레귤론(PGAL1, PGAL10, PGAL2, PGAL7)에 의해 조절되는 야생형 프로모터를 사용할 수 있다. 그림은 Zhao et al.12에서 수정되었습니다.이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
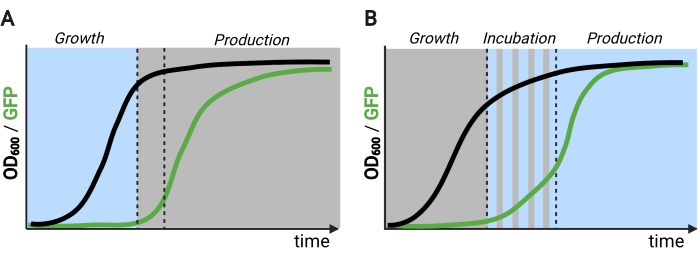
그림 2: 시간에 따른 2상 및 삼상 발효. (A) 거꾸로 된 회로로 작동하는 2상 발효는 광구동 성장 단계와 어두운 생산 단계로 구성됩니다. 성장 단계에서 바이오 매스는 생산 경로가 억압 된 상태로 유지됨에 따라 축적됩니다. 원하는 OD600에 도달하면, 세포는 생산 단계를 위해 신선한 배지에 재현탁되기 전에 대사적으로 조정하기 위해 어둠으로 이동된다. (B) 삼상 공정에서 성장, 배양 및 생산 단계는 어두운 성장 기간, 펄스 인큐베이션 및 완전히 조명 된 생산 단계로 구성 될 수있는 고유 한 광 일정에 의해 정의됩니다. 바이오렌더로 만든 그림입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
광유전학 회로는 또한 대장균에서 화학 및 단백질 생산의 동적 제어를 위해 개발되었다. OptoLAC 회로는 YF1/FixJ 두 구성 요소 시스템6을 기반으로 하는 광 반응 pDawn 회로를 사용하여 박테리아 LacI 리프레저를 제어합니다(그림 3). OptoINVRT5와 유사하게, OptoLAC 회로는 빛에서 유전자 발현을 억제하고 어둠 속에서 유도하도록 설계되었습니다. OptoLAC 회로를 사용하는 발현 수준은 표준 이소프로필 β-d-1-티오갈락토피라노사이드(IPTG) 유도로 달성된 수준과 일치하거나 초과할 수 있으므로 화학적 유도의 강도를 유지하면서 향상된 튜닝성 및 가역성을 제공합니다6. 따라서, OptoLAC 회로는 대장균에서 대사 공학을 위한 효과적인 광유전학적 조절을 가능하게 한다.
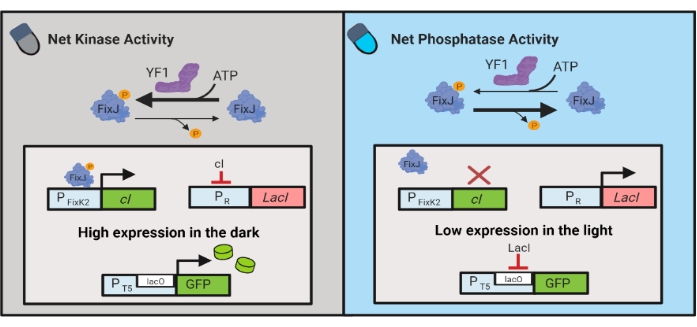
그림 3: 대장균의 동적 제어를 위한 OptoLAC 회로. OptoLAC 회로는 pDawn 시스템과 lac 오페론을 조정하여 어둠 속에서의 활성화와 빛에서의 억압을 달성합니다. 어둠 속에서, YF1은 FixJ를 인산화시키고, 이는 pFixK2 프로모터를 활성화시켜 cI 리프레서를 발현시킨다. cI 리프레서는 PR 프로모터로부터의 lacI 리프레서의 발현을 방지하며, 이는 lacO 함유 프로모터로부터 관심있는 유전자의 전사를 허용한다. 반대로, 청색광은 YF1 순 키나제 활성을 감소시키고, FixJ 인산화 및 따라서 cI 발현을 역전시키고, 이는 lacI의 발현을 억제하고 lacO 함유 프로모터로부터의 발현을 방지한다. 이 그림은 Lalwani et al.6에서 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
우리는 화학 또는 단백질 생산을위한 S. cerevisiae 및 대장균 의 광 조절 발효를위한 기본 프로토콜을 여기에서 설명합니다. 효모와 박테리아 모두를 위해, 우리는 먼저 OptoINVRT 및 OptoLAC 회로에 의해 활성화 된 빛 구동 성장 단계와 어둠으로 인한 생산 단계를 가진 발효에 중점을 둡니다. 이어서, 우리는 OptoAMP 회로에 의해 활성화된 삼상(성장, 유도, 생산) 광 제어 발효를 위한 프로토콜을 설명한다. 또한, 우리는 광유전학적으로 제어된 발효물을 마이크로플레이트에서 실험실 규모의 생물반응기로 확장하는 방법을 설명합니다. 이 프로토콜을 통해 우리는 화학 또는 단백질 생산을위한 광 제어 발효를 수행하기위한 완전하고 쉽게 재현 할 수있는 가이드를 제공하는 것을 목표로합니다.
Protocol
Representative Results
Discussion
동적 조절은 대사 공학 및 재조합 단백질 생산을 위한 수율을 향상시키기 위해 오랫동안 적용되어 왔다4. 효소 발현의 이동은 가장 일반적으로 IPTG21, 갈락토오스22 및 테트라사이클린23과 같은 화학 유도제를 사용하여 구현되지만 온도 및 pH와 같은 공정 조건을 사용하여 매개되었습니다. 유전자 발현의 광유전학적 조절은 발?…
Declarações
The authors have nothing to disclose.
Acknowledgements
이 연구는 미국 에너지부, 과학 사무소, 생물 및 환경 연구 사무소 상 번호 DE-SC0019363, NSF CAREER Award CBET-1751840, 퓨 자선 신탁 및 카밀 드레퓌스 교사 – 학자 상이 지원했습니다.
Materials
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
Referências
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. Bioengenharia. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).

