En masspektrometribaserad metod för att identifiera fosfoproteinfosfataser och deras interagerare
Summary
Här presenterar vi ett protokoll för anrikning av endogena fosfoproteinfosfataser och deras interagerande proteiner från celler och vävnader och deras identifiering och kvantifiering genom masspektrometribaserad proteomik.
Abstract
De flesta cellulära processer regleras av dynamisk proteinfosforylering. Mer än tre fjärdedelar av proteinerna fosforyleras och fosfoproteinfosfataser (PPP) samordnar över 90% av all cellulär serin/ treonin defosforylering. Avreglering av proteinfosforylering har varit inblandad i patofysiologin för olika sjukdomar, inklusive cancer och neurodegeneration. Trots sin utbredda aktivitet är de molekylära mekanismer som styr växtskyddsmedel och de som kontrolleras av växtskyddsmedel dåligt karakteriserade. Här beskrivs ett proteomiskt tillvägagångssätt som kallas fosfatasinhibitorpärlor och masspektrometri (PIB-MS) för att identifiera och kvantifiera PPP, deras posttranslationella modifieringar och deras interagerare på så lite som 12 timmar med hjälp av någon cellinje eller vävnad. PIB-MS använder en icke-selektiv PPP-hämmare, mikrocystin-LR (MCLR), immobiliserad på sepharospärlor för att fånga och berika endogena PPP och deras associerade proteiner (kallad PPPome). Denna metod kräver inte exogent uttryck av märkta versioner av växtskyddsmedel eller användning av specifika antikroppar. PIB-MS erbjuder ett innovativt sätt att studera de evolutionärt bevarade PPP:erna och utöka vår nuvarande förståelse av defosforyleringssignalering.
Introduction
Proteinfosforylering styr de flesta cellulära processer, inklusive men inte begränsat till svaret på DNA-skador, tillväxtfaktorsignalering och passagen genom mitos 1,2,3. I däggdjursceller fosforyleras majoriteten av proteinerna vid en eller flera serin-, treonin- eller tyrosinrester vid någon tidpunkt, med fosfoseriner och fosfotreoniner som utgör cirka 98% av alla fosforyleringsställen 2,3. Medan kinaser har studerats omfattande inom cellulär signalering, är PPP: s roll i regleringen av dynamiska cellulära processer fortfarande framväxande.
Fosforyleringsdynamiken styrs av det dynamiska samspelet mellan kinaser och fosfataser. I däggdjursceller finns det mer än 400 proteinkinaser som katalyserar serin / treoninfosforylering. Över 90% av dessa platser defosforyleras av fosfoproteinfosfataser (PPP), en liten familj av enzymer som består av PP1, PP2A, PP2B, PP4-7, PPT och PPZ 2,3. PP1 och PP2A är ansvariga för majoriteten av fosfoserin och fosfotreonin defosforylering inom en cell 2,3,4. Den anmärkningsvärda skillnaden i antal mellan kinaser och fosfataser och bristen på specificitet hos PPP-katalytiska subenheter in vitro ledde till tron att kinaser är den viktigaste determinanten för fosforylering 2,3. Emellertid, flera studier har visat fosfataser för att fastställa substratspecificitet genom bildandet av multimera holoenzymer 5,6,7,8,9. Till exempel är PP1 en heterodimer som består av en katalytisk underenhet och vid en given tidpunkt en av de mer än 150 reglerande underenheterna 6,7,8. Omvänt är PP2A en heterotrimer som bildas av en byggnadsställning (A), en reglerande (B) och en katalytisk (C) underenhet 2,3,9. Det finns fyra distinkta familjer av PP2A-regulatoriska underenheter (B55, B56, PR72 och striatin), var och en med flera gener, skarvvarianter och lokaliseringsmönster 2,3,9. Offentlig-privata partnerskapens multimeriska karaktär fyller gapet i antalet kinaser och PPP-katalytiska underenheter. Det skapar dock analytiska utmaningar för att studera PPP-signalering. För att omfattande analysera PPP-signalering är det viktigt att undersöka de olika holoenzymerna i en cell eller vävnad. Stora framsteg har gjorts för att studera det mänskliga släktomet genom användning av kinashämmare pärlor, kallade multiplexhämmare pärlor eller kinobeads, en kemisk proteomisk strategi där kinashämmare immobiliseras på pärlor och masspektrometri används för att identifiera berikade kinaser och deras interagerare 10,11,12,13.
Vi har etablerat ett liknande tillvägagångssätt för att studera PPP-biologi. Denna teknik involverar affinitetsfångst av PPP-katalytiska underenheter med användning av pärlor med en immobiliserad, icke-selektiv PPP-hämmare som kallas mikrocystin-LR (MCLR) som kallas fosfatashämmarepärlor (PIBs)14,15. Till skillnad från andra metoder som kräver endogen märkning eller uttryck av exogena PPP-underenheter som kan förändra proteinaktivitet eller lokalisering, möjliggör PIB-MS anrikning av endogena PPP-katalytiska underenheter, deras associerade reglerande och byggnadsställningar och interagerande proteiner (kallat PPPome) från celler och vävnader vid en given tidpunkt eller under specifika behandlingsförhållanden. MCLR hämmar PP1, PP2A, PP4-6, PPT och PPZ vid nanomolära koncentrationer, vilket gör PIBs mycket effektiva vid anrikning för PPPome16. Denna metod kan skalas för användning på vilket utgångsmaterial som helst från celler till kliniska prover. Här beskriver vi i detalj användningen av PIB och masspektrometri (PIB-MS) för att effektivt fånga, identifiera och kvantifiera det endogena PPPome och dess modifieringstillstånd.
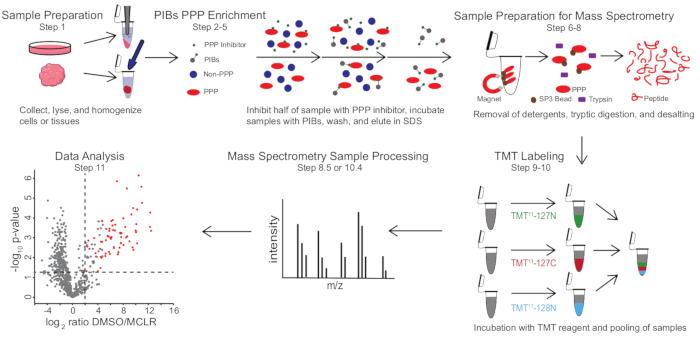
Bild 1: Visuell sammanfattning av PIB-MS-protokollet. I ett PIB-MS-experiment kan prover erhållas i olika former, från celler till tumörer. Provet samlas in, lyseras och homogeniseras före PPP-anrikning. För att berika för PPP inkuberas lysaten med PIBs med eller utan en PPP-hämmare, såsom MCLR. PDB tvättas sedan och växtskyddsmedel elueras under denatureringsförhållanden. Proverna bereds för masspektrometrianalys genom avlägsnande av tvättmedel genom SP3-proteinanrikning, tryptisk matsmältning och avsaltning. Prover kan sedan valfritt TMT-märkas före masspektrometrianalys. Klicka här för att se en större version av denna siffra.
PIB-MS involverar lys och förtydligande av celler eller vävnader, inkubation av lysaten med PIBs, eluering och analys av eluatet via western blotting eller masspektrometribaserade metoder (Figur 1). Tillägget av fri MCLR kan användas som en kontroll för att skilja specifika PIB-bindemedel från icke-specifika interagerare. För de flesta applikationer kan ett etikettfritt tillvägagångssätt användas för att direkt identifiera proteiner i eluater. I de fall där större precision i kvantifieringen eller identifieringen av arter med låg förekomst behövs, kan ytterligare bearbetning med tandem-masstagg (TMT) -märkning användas för att öka täckningen och minska inmatningen.
Protocol
Representative Results
Discussion
PIB-MS är en kemisk proteomikmetod som används för att kvantitativt profilera PPPome från olika provkällor i en enda analys. Mycket arbete har gjorts med hjälp av kinashämmare pärlor för att studera kinomet och hur det förändras i cancer och andra sjukdomstillstånd 10,11,12,13. Ändå ligger studien av PPPome bakom. Vi förväntar oss att detta tillvägagångssätt kan fylla detta g…
Declarações
The authors have nothing to disclose.
Acknowledgements
A.N.K. erkänner stöd från NIH R33 CA225458 och R35 GM119455. Vi tackar Kettenbach- och Gerber-laboratorierna för deras hjälpsamma diskussion.
Materials
| Acetonitrile (ACN) | Honeywell | AH015-4 | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Anhydrous Acetonitrile | Sigma-Aldrich | 271004-100ML | CAUTION: ACN is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Benchtop centrifuge | Eppendorf | model no. 5424 | |
| Beta-glycerophosphoric acid, disodium salt pentahydrate | Acros Organics | 410991000 | |
| Centrifuge | Eppendorf | model no. 5810 R 15 amp version | |
| Distilled water | |||
| DMSO | Fisher Scientific | BP231-100 | |
| Dounce tissue grinder | Fisherbrand Pellet Pestles | 12-141-363 | |
| Empore solid phase extraction disk, C18 | CDS Analytical | 76333-132 | |
| Eppendorf tubes, 1.5 mL | Eppendorf | 22363204 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Eppendorf tubes, 2 mL | Eppendorf | 22363352 | CRITICAL: Other tubes may leach polymer into sample, contaminating the analysis. |
| Extraction plate manifold | Waters | WAT097944 | |
| Falcon tubes, 50 mL | VWR | 21008 | |
| Generic blunt end needle and plunger | |||
| Generic magnetic separation rack | |||
| HEPES | Sigma-Aldrich | H3375 | |
| Hydrogen chloride (HCl) | VWR Chemicals BDH | BDH3028 | CAUTION: HCl is corrosive; wear gloves and work in a chemical fume hood. |
| Hydroxylamine solution 50% (wt/vol) | Sigma-Aldrich | 467804 | |
| Incubator, 65 °C | VWR | model no. 1380FM | |
| Koptec Pure Ethanol, 200 Proof | Decon Labs | V1001 | |
| Methanol for HPLC (MeOH) | Sigma-Aldrich | 34860-4L-R | CAUTION: MeOH is flammable and toxic; wear gloves, and work in a chemical fume hood. |
| Microcystin LR (MCLR) | Cayman Chemical | 10007188 | CAUTION: MCLR is toxic; wear gloves when handling and avoid skin contact. |
| PBS, 1× without calcium and magnesium, pH 7.4 ± 0.1 | Corning | 21-040-CV | |
| pH test strips, such as MilliporeSigma MColorpHast pH test strips and indicator papers | Fisher Scientific | M1095310001 | |
| PIBs | For protocol for the generation of PIBs, see Moorhead et al., 2007. | ||
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23225 | |
| Pipette tips, 10 μL | Eppendorf | 22491504 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 1000 μL | Eppendorf | 22491555 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| Pipette tips, 200 μL | Eppendorf | 22491539 | CRITICAL: Other tips may leach polymer into samples, contaminating the analysis. |
| plastic syringe, 10 mL | BD | 309604 | |
| Protease inhibitor cocktail III | Research Products International | P50700-1 | |
| Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer, Oribtrap Fusion, Orbitrap Fusion Lumos, or Orbitrap Eclipse Tribrid Mass Spectrometer | Thermo Scientific | ||
| Refrigerated benchtop centrifuge | Eppendorf | model no. 5424 R | |
| Rotator (Labquake Shaker Rotisserie) | Thermo Scientific | 13-687-12Q | 8 rpm rotation |
| Sample collection plate, 96- well, 1 mL | Waters | WAT058957 | |
| SDS | Fisher Scientific | BP1311-1 | |
| Sequencing grade modified trypsin | Promega | V511C | |
| Sodium azide | EMD Chemicals | SX0299-1 | CAUTION: Sodium azide is explosive and toxic; wear gloves, work in a chemical fume hood and avoid contact with metals. |
| Sodium chloride (NaCl) | Fisher Chemical | S27110 | |
| Sonicator (Branson digital sonifier) | model no. SFX 250 | ||
| SPE C18 desalting plate | Waters | 186001828BA | |
| SpeedBeads magnetic carboxylate modified particles (SP3 beads) | Cytiva | 6.51521E+13 | |
| Thermomixer | Eppendorf | model no. 5350 | |
| TMT10plex Isobaric Label Reagent Set plus TMT11-131C Label Reagent, 3 × 0.8 mg per tag | ThermoFisher | A37725 | |
| Trifluoroacetic acid (TFA) | Honeywell | T6508-25ML | CAUTION: TFA is corrosive and will irritate skin on contact. Wear gloves and eye protection, and work in a chemical fume hood. |
| Tris Base | Research Products International | T60040 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Vacuum centrifuge and vapor trap | Thermo Scientific | model nos. SpeedVac SPD120 and RVT5105 | |
| Vortexer (Vortex-Genie 2) | Scientific Industries | ||
| Water LC-MS | Honeywell | LC365-4 |
Referências
- Nilsson, J. Protein phosphatases in the regulation of mitosis. Journal of Cell Biology. 218 (2), 395-409 (2019).
- Brautigan, D. L. Protein Ser/Thr phosphatases–the ugly ducklings of cell signalling. The FEBS Journal. 280 (2), 324-345 (2013).
- Brautigan, D. L., Shenolikar, S. Protein serine/threonine phosphatases: keys to unlocking regulators and substrates. Annual Review of Biochemistry. 87, 921-964 (2018).
- Janssens, V., Goris, J. Protein phosphatase 2A: A highly regulated family of serine/threonine phosphatases implicated in cell growth and signalling. Biochemical Journal. 353, 417-439 (2001).
- Virshup, D. M., Shenolikar, S. From promiscuity to precision: protein phosphatases get a makeover. Molecular Cell. 33 (5), 537-545 (2009).
- Bollen, M., Peti, W., Ragusa, M. J., Beullens, M. The extended PP1 toolkit: designed to create specificity. Trends in Biochemical Sciences. 35 (8), 450-458 (2010).
- Qian, J., Winkler, C., Bollen, M. 4D-networking by mitotic phosphatases. Current Opinion in Cell Biology. 25 (6), 697-703 (2013).
- Heroes, E., et al. The PP1 binding code: a molecular-lego strategy that governs specificity. The FEBS Journal. 280 (2), 584-595 (2013).
- Eichhorn, P. J., Creyghton, M. P., Bernards, R. Protein phosphatase 2A regulatory subunits and cancer. Biochimica et Biophysica Acta. 1795 (1), 1-15 (2009).
- Bantscheff, M., et al. Quantitative chemical proteomics reveals mechanisms of action of clinical ABL kinase inhibitors. Nature Biotechnology. 25 (9), 1035-1044 (2007).
- Klaeger, S., et al. Chemical proteomics reveals ferrochelatase as a common off-target of kinase inhibitors. ACS Chemical Biology. 11 (5), 1245-1254 (2016).
- Duncan, J. S., et al. Dynamic reprogramming of the kinome in response to targeted MEK inhibition in triple-negative breast cancer. Cell. 149 (2), 307-321 (2012).
- Cooper, M. J., et al. Application of multiplexed kinase inhibitor beads to study kinome adaptations in drug-resistant leukemia. PLoS ONE. 8 (6), 66755 (2013).
- Lyons, S. P., et al. A quantitative chemical proteomic strategy for profiling phosphoprotein phosphatases from yeast to humans. Molecular and Cellular Proteomics. 17 (12), 2448-2461 (2018).
- Nasa, I., et al. Quantitative kinase and phosphatase profiling reveal that CDK1 phosphorylates PP2Ac to promote mitotic entry. Science Signaling. 13 (648), (2020).
- Swingle, M., Ni, L., Honkanen, R. E. Small-molecule inhibitors of ser/thr protein phosphatases: specificity, use and common forms of abuse. Methods in Molecular Biology. 365, 23-38 (2007).
- Moorhead, G. B. G., Haystead, T. A. J., MacKintosh, C. Synthesis and use of the protein phosphatase affinity matrices microcystin-sepharose and microcystin-biotin-sepharose. Methods in Molecular Biology. 365, 39-45 (2007).
- Brauer, B. L., Wiredu, K., Mitchell, S., Moorhead, G. B., Gerber, S. A., Kettenbach, A. N. Affinity-based profiling of endogenous phosphoprotein phosphatases by mass spectrometry. Nature Protocols. 16 (10), 4919-4943 (2021).
- Hughes, C. S., Moggridge, S., Müller, T., Sorensen, P. H., Morin, G. B., Krijgsveld, J. Single-pot, solid-phase-enhanced sample preparation for proteomics experiments. Nature Protocols. 14 (1), 68-85 (2019).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Zecha, J., et al. TMT labeling for the masses: A robust and cost-efficient, in-solution labeling approach. Molecular and Cellular Proteomics. 18 (7), 1468-1478 (2019).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Eng, J. K., Jahan, T. A., Hoopmann, M. R. Comet: an open-source MS/MS sequence database search tool. Proteomics. 13 (1), 22-24 (2013).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Yu, S. H., Ferretti, D., Schessner, J. P., Rudolph, J. D., Borner, G. H. H., Cox, J. Expanding the Perseus software for omics data analysis With custom plugins. Current Protocols in Bioinformatics. 71 (1), 1-29 (2020).

