High-Throughput screening van microbiële isolaten met impact op caenorhabditis elegans gezondheid
Summary
Darmmicroben kunnen de gezondheid van hun gastheer positief of negatief beïnvloeden via specifieke of geconserveerde mechanismen. Caenorhabditis elegans is een handig platform om te screenen op dergelijke microben. Het huidige protocol beschrijft high-throughput screening van 48 bacteriële isolaten op impact op nematodenstressresistentie, gebruikt als proxy voor de gezondheid van wormen.
Abstract
Met zijn kleine formaat, korte levensduur en gemakkelijke genetica biedt Caenorhabditis elegans een handig platform om de impact van microbiële isolaten op de fysiologie van de gastheer te bestuderen. Het fluoresceert ook in blauw tijdens het sterven, wat een handig middel is om de dood te lokaliseren. Deze eigenschap is gebruikt om high-throughput labelvrije C. elegans survival assays (LFASS) te ontwikkelen. Deze omvatten time-lapse fluorescentieregistratie van wormpopulaties in multiwell-platen, waaruit populatiemediane tijd van overlijden kan worden afgeleid. De huidige studie hanteert de LFASS-benadering om meerdere microbiële isolaten tegelijk te screenen op de effecten op de gevoeligheid van C. elegans voor ernstige hitte en oxidatieve stress. Een dergelijke microbiële screeningspijplijn, die met name kan worden gebruikt om probiotica vooraf te screenen, met behulp van ernstige stressresistentie als een proxy voor de gezondheid van de gastheer, wordt hier gerapporteerd. Het protocol beschrijft hoe zowel C. elegans darmmicrobiota-isolaatcollecties als synchrone wormpopulaties in multiwell-arrays kunnen groeien voordat ze worden gecombineerd voor de assays. Het gegeven voorbeeld omvat het testen van 47 bacteriële isolaten en één controlestam op twee wormstammen, in twee stresstests parallel. De aanpakpijplijn is echter gemakkelijk schaalbaar en toepasbaar op de screening van vele andere modaliteiten. Het biedt dus een veelzijdige opstelling om snel een multiparametrisch landschap van biologische en biochemische omstandigheden te onderzoeken die van invloed zijn op de gezondheid van C. elegans .
Introduction
Het menselijk lichaam herbergt naar schatting 10-100 biljoen levende microbiële cellen (bacteriën, archaea-schimmels), die voornamelijk worden aangetroffen in de darm-, huid- en mucosale omgevingen1. In een gezonde toestand bieden deze voordelen voor hun gastheer, waaronder vitamineproductie, rijping van het immuunsysteem, stimulatie van aangeboren en adaptieve immuunresponsen op pathogenen, regulatie van vetmetabolisme, modulatie van stressreacties en meer, met een impact op groei en ontwikkeling, het begin van de ziekte en veroudering 2,3,4,5 . De darmmicrobiota evolueert ook aanzienlijk gedurende het hele leven. De meest drastische evolutie vindt plaats tijdens de kindertijd en de vroege kindertijd6, maar significante veranderingen treden ook op met de leeftijd, waaronder een afname van de overvloed aan Bifidobacterium en een toename van Clostridium, Lactobacillus, Enterobacteriaceae pt Enterococcus species7. Levensstijl kan de microbiële samenstelling van de darm verder veranderen, wat leidt tot dysbiose (verlies van nuttige bacteriën, overgroei van opportunistische bacteriën), wat resulteert in verschillende pathologieën zoals inflammatoire darmaandoeningen, diabetes en obesitas5, maar ook bijdragen aan de ziekte van Alzheimer en Parkinson 8,9,10,11.
Dit besef heeft kritisch bijgedragen aan het verfijnen van het concept van de darm-hersenas (GBA), waar interacties tussen darmfysiologie (nu inclusief de microben erin) en het zenuwstelsel worden beschouwd als de belangrijkste regulator van dierlijk metabolisme en fysiologische functies12. De precieze rol van microbiota in darm-hersensignalering en de bijbehorende werkingsmechanismen zijn echter nog lang niet volledig begrepen13. Met darmmicrobiota als een belangrijke determinant van gezond ouder worden, is hoe bacteriën het verouderingsproces moduleren een onderwerp geworden van intensief onderzoek en controverse 6,14,15.
Met de demonstratie dat de rondworm Caenorhabditis elegans een bonafide darmmicrobiota herbergt die wordt gedomineerd – zoals bij andere soorten – door Bacteroidetes, Firmicutes pt Actinobacteria 16,17,18,19,20, is de snelle opkomst ervan als een experimenteel platform om gastheer-darm commensale interacties te bestuderen 21,22,23,24 ,25,26 heeft ons onderzoeksarsenaal aanzienlijk uitgebreidmet 26,27,28,29. In het bijzonder kunnen high-throughput experimentele benaderingen die beschikbaar zijn voor C. elegans om gen-dieet, gen-medicijn, gen-pathogeen, enz. interacties te bestuderen, worden aangepast om snel te onderzoeken hoe bacteriële isolaten en cocktails de gezondheid en veroudering van C. elegans beïnvloeden.
Het huidige protocol beschrijft een experimentele pijplijn om in één keer arrays van bacteriële isolaten of mengsels in multiwell-platen te screenen op effecten op C. elegans stressresistentie als een proxy voor gezondheid, die kan worden gebruikt om probiotica te identificeren. Het beschrijft hoe grote wormpopulaties kunnen groeien en bacteriële arrays in 96- en 384-well plaatformaten kunnen worden verwerkt voordat wormen worden verwerkt voor geautomatiseerde stressbestendigheidsanalyse met behulp van een fluorescentieplaatlezer (figuur 1). De aanpak is gebaseerd op labelvrije geautomatiseerde overlevingstests (LFASS)30 die gebruik maken van het fenomeen van doodsfluorescentie31, waarbij stervende wormen een uitbarsting van blauwe fluorescentie produceren die kan worden gebruikt om het tijdstip van overlijden te bepalen. Blauwe fluorescentie wordt uitgestoten door glucosylesters van anthranilic acid opgeslagen in C. elegans darmkorrels (een soort lysosoom-gerelateerd organel), die barsten wanneer een necrotische cascade wordt geactiveerd in de worm darm bij overlijden31.
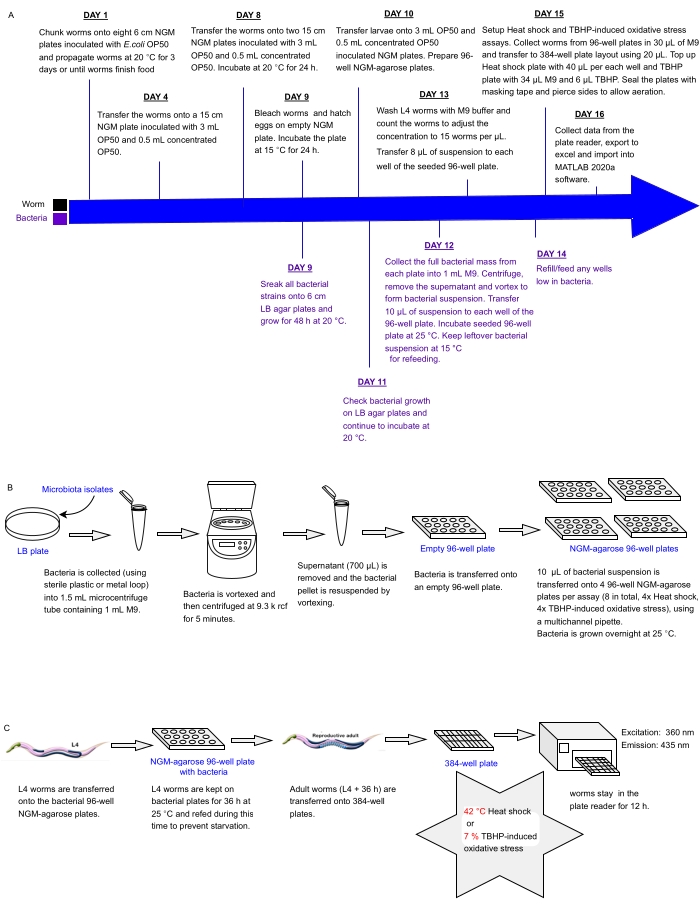
Figuur 1: Experimentele workflow voor high-throughput screening van bacteriële isolaten met impact op C. elegans resistentie tegen stress. (A) Tijdlijn voor worm- en bacterieonderhoud en testopstelling. (B) 96-well bacteriële plaat array setup en handling. (C) 384-well wormplaat opstelling. Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
C. elegans biedt vele voordelen voor het snel screenen van meerdere experimentele parameters tegelijk, vanwege het kleine formaat, de transparantie, de snelle ontwikkeling, de korte levensduur, de goedkoopheid en het gebruiksgemak. Het aanzienlijk eenvoudigere genoom, het lichaamsplan, het zenuwstelsel, de darm en het microbioom, maar toch complex en vergelijkbaar genoeg met mensen, maken het een krachtig preklinisch model, waar mechanistisch inzicht kan worden verkregen tijdens het testen op bioactieve werkzaam…
Declarações
The authors have nothing to disclose.
Acknowledgements
We bedanken de CGC Minnesota (Madison, VS, NIH – P40 OD010440) voor het leveren van wormstammen en OP50 en Pr. Hinrich Schulenburg (CAU, Kiel, Duitsland) voor het leveren van alle microbiële isolaten uit het milieu die hier worden afgebeeld. Dit werk werd gefinancierd door een UKRI-BBSRC-subsidie aan AB (BB/S017127/1). JM wordt gefinancierd door een Lancaster University FHM PhD-beurs.
Materials
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
Referências
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood – Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977 (2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer’s disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98 (2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson’s disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921 (2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095 (2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323 (2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio – The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364 (2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545 (2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849 (2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998 (2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613 (2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer’s disease: a systematic review. Nutrients. 14 (1), 20 (2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson’s disease. Ageing Research Reviews. 69, 101347 (2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -. E. H., Jung, Y., Lee, S. -. J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190 (2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652 (2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583 (2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398 (2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448 (2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35 (2016).

