カエノラブディティス・エレガンスの健康に影響を与える微生物分離物のハイスループットスクリーニング
Summary
腸内微生物は、特定のまたは保存されたメカニズム を介して 宿主の健康にプラスまたはマイナスの影響を与える可能性があります。 カエノラブディティスエレガンスは 、そのような微生物をスクリーニングするための便利なプラットフォームです。本プロトコルは、線虫の健康状態の代理として使用される、線虫ストレス耐性への影響についての48の細菌分離株のハイスループットスクリーニングを記載している。
Abstract
Caenorhabditis elegansは、サイズが小さく、寿命が短く、遺伝学が容易なため、微生物分離株が宿主生理機能に与える影響を研究するための便利なプラットフォームを提供します。また、死ぬと青色に蛍光を発し、死を特定する便利な手段を提供します。この特性を利用して、ハイスループットラベルフリーのC.エレガンス生存アッセイ(LFASS)を開発しました。これらには、マルチウェルプレートにセットされた線虫集団のタイムラプス蛍光記録が含まれ、そこから集団死亡時間の中央値を導き出すことができます。本研究では、LFASSアプローチを採用して、複数の微生物分離株を一度にスクリーニングし、 C.エレガンス 重度の熱および酸化ストレスに対する感受性。このような微生物スクリーニングパイプラインは、特にプロバイオティクスをプレスクリーニングするために使用でき、宿主の健康の代理として重度のストレス耐性を使用することがここで報告されています。このプロトコルでは、C. elegans腸内細菌叢分離コレクションと同期線虫集団の両方をマルチウェルアレイで増殖させてから、アッセイのためにそれらを組み合わせる方法を説明しています。提供された例は、2つのストレスアッセイを並行して行う2つのワーム株に対する47の細菌分離株と1つの対照株のテストをカバーしています。ただし、アプローチパイプラインは容易に拡張可能であり、他の多くのモダリティのスクリーニングに適用できます。したがって、C.エレガンスの健康に影響を与える生物学的および生化学的条件のマルチパラメトリックランドスケープを迅速に調査するための汎用性の高いセットアップを提供します。
Introduction
人体には、主に腸、皮膚、粘膜環境に見られる推定10〜100兆個の生きた微生物細胞(細菌、古細菌)が生息しています1。健康な状態では、これらはビタミン産生、免疫系の成熟、病原体に対する自然免疫応答および適応免疫応答の刺激、脂肪代謝の調節、ストレス応答の調節など、宿主に利益をもたらし、成長と発達、疾患の発症、および老化に影響を与えます2,3,4,5.腸内細菌叢も生涯を通じてかなり進化します。最も劇的な進化は乳児期と幼児期に起こりますが6、ビフィズス菌の存在量の減少やクロストリジウム、ラクトバチルス、腸内細菌科、 エンテロコッカス種の増加など、年齢とともに大きな変化も起こります7。ライフスタイルは腸内微生物の組成をさらに変化させ、腸内細菌叢症(有益な細菌の喪失、日和見細菌の異常増殖)を引き起こし、炎症性腸疾患、糖尿病、肥満などのさまざまな病状を引き起こす可能性があります5だけでなく、アルツハイマー病やパーキンソン病にも寄与します8,9,10,11。
この認識は、腸生理学(現在はその中の微生物を含む)と神経系の間の相互作用が動物の代謝と生理学的機能の主要な調節因子と考えられている腸脳軸(GBA)の概念の洗練に大きく貢献しています12。しかし、腸脳シグナル伝達における微生物叢の正確な役割とそれに関連する作用機序は完全には理解されていません13。腸内細菌叢が健康的な老化の重要な決定要因であるため、細菌が老化プロセスをどのように調節するかは、激しい研究と論争の対象となっています6,14,15。
回虫Caenorhabditis elegansが、他の種と同様に、バクテロイデス、ファーミキューテス、および放線菌によって優勢な正真正銘の腸内細菌叢を宿主としていることの実証により16,17,18,19,20、宿主と腸の共生相互作用を研究するための実験プラットフォームとしての急速な台頭21,22,23,24、25,26は、調査兵器26,27,28,29を大幅に拡大しました。特に、C.エレガンスが遺伝子-食事、遺伝子-薬物、遺伝子-病原体などの相互作用を研究するために利用できるハイスループット実験アプローチを適応させて、細菌分離株とカクテルがC.エレガンスの健康と老化にどのように影響するかを迅速に調査することができます。
本プロトコルは、健康の代理として C.エレガンスの ストレス耐性への影響についてマルチウェルプレートにセットされた細菌分離株または混合物のアレイを一度にスクリーニングするための実験パイプラインを説明し、プロバイオティクスを同定するために使用することができる。蛍光プレートリーダーを使用した自動ストレス耐性分析のためにワームを処理する前に、大規模なワーム集団を増殖させ、96ウェルおよび384ウェルプレートフォーマットで細菌アレイを処理する方法について詳しく説明します(図1)。このアプローチは、死に至る線虫が死の時間を特定するために使用できる青色蛍光のバーストを生成する死の蛍光31 の現象を利用するラベルフリー自動生存アッセイ(LFASS)30に基づいています。青色蛍光は、 C.エレガンス 腸顆粒(リソソーム関連オルガネラの一種)に貯蔵されたアントラニル酸のグルコシルエステルによって放出され、死亡時に線虫腸内で壊死カスケードがトリガーされると破裂します31。
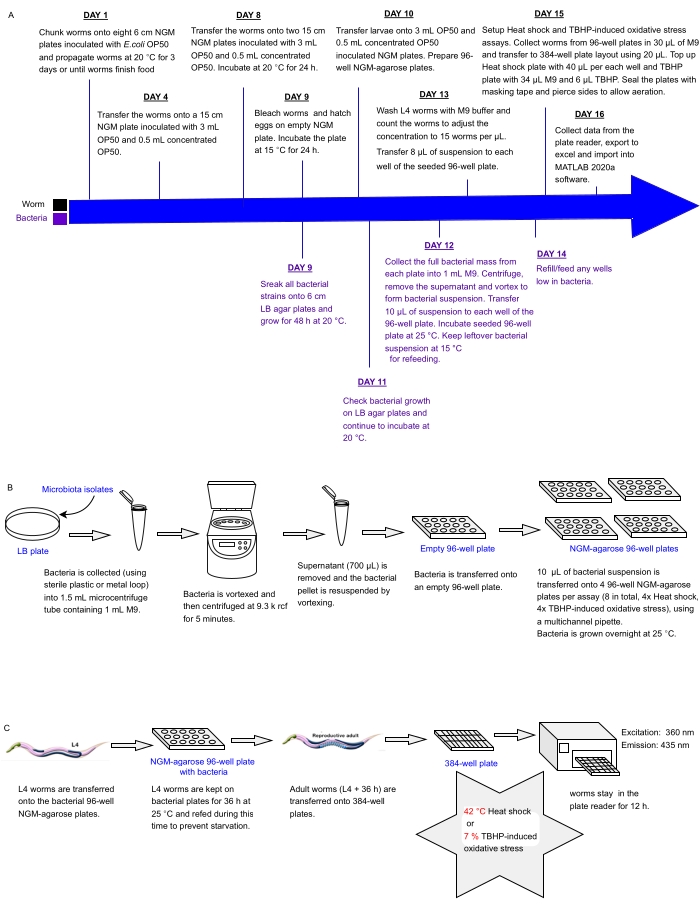
図1: ストレスに対するC.エレガンスの 耐性に影響を与える細菌分離株のハイスループットスクリーニングのための実験ワークフロー 。 (A)ワームとバクテリアのメンテナンスとアッセイのセットアップのタイムライン。(B)96ウェル細菌プレートアレイのセットアップと取り扱い。(C)384ウェルウォームプレートのセットアップ。 この図の拡大版を表示するには、ここをクリックしてください。
Protocol
Representative Results
Discussion
C. elegansは、その小型、透明性、迅速な開発、短寿命、安価、取り扱いの容易さにより、一度に複数の実験パラメータを迅速にスクリーニングするための多くの利点を提供します。そのかなり単純なゲノム、ボディプラン、神経系、腸、およびマイクロバイオームでありながら、複雑で人間と十分に類似しているため、生物活性の有効性または毒性をテストしながらメカニズムの洞察を…
Declarações
The authors have nothing to disclose.
Acknowledgements
我々は、寄生虫株を提供してくれたCGCミネソタ州(米国マディソン、NIH – P40 OD010440)と、ここに描かれているすべての環境微生物分離株を提供してくれたOP50およびPr. Hinrich Schulenburg(CAU、キール、ドイツ)に感謝する。この研究は、ABへのUKRI-BBSRC助成金(BB / S017127 / 1)によって資金提供されました。JMは、ランカスター大学FHM博士号奨学金によって資金提供されています。
Materials
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
Referências
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood – Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977 (2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer’s disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98 (2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson’s disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921 (2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095 (2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323 (2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio – The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38 (2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364 (2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545 (2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849 (2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998 (2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613 (2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019 (2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer’s disease: a systematic review. Nutrients. 14 (1), 20 (2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson’s disease. Ageing Research Reviews. 69, 101347 (2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -. E. H., Jung, Y., Lee, S. -. J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190 (2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652 (2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583 (2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398 (2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448 (2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35 (2016).

