सोयाबीन सहजीवी नोड्यूल से पॉलीसोम शुद्धिकरण
Summary
यह प्रोटोकॉल बरकरार सोयाबीन नोड्यूल से यूकेरियोटिक पॉलीसोम शुद्धिकरण के लिए एक विधि का वर्णन करता है। अनुक्रमण के बाद, जीन अभिव्यक्ति विश्लेषण के लिए मानक पाइपलाइनों का उपयोग ट्रांसक्रिप्टोम और ट्रांसलेटोम स्तरों पर अलग-अलग व्यक्त जीन की पहचान करने के लिए किया जा सकता है।
Abstract
इस प्रोटोकॉल का उद्देश्य सोयाबीन (ग्लाइसिन अधिकतम) सहजीवी नोड्यूल के यूकेरियोटिक अनुवाद का अध्ययन करने के लिए एक रणनीति प्रदान करना है। यह पत्र पौधे-व्युत्पन्न पॉलीराइबोसोम और उनके संबंधित एमआरएनए को आरएनए-अनुक्रमण का उपयोग करके विश्लेषण करने के लिए अनुकूलित तरीकों का वर्णन करता है। सबसे पहले, साइटोप्लाज्मिक लाइसेट्स पूरे, जमे हुए सोयाबीन नोड्यूल से पॉलीसोम- और आरएनए-संरक्षण स्थितियों में होमोजेनाइजेशन के माध्यम से प्राप्त किए जाते हैं। फिर, लाइसेट्स को कम गति वाले सेंट्रीफ्यूजेशन द्वारा साफ किया जाता है, और सतह पर तैरनेवाला का 15% कुल आरएनए (कुल) अलगाव के लिए उपयोग किया जाता है। शेष साफ़ लाइसेट का उपयोग दो-परत सुक्रोज कुशन (12% और 33.5%) के माध्यम से अल्ट्रासेंट्रीफ्यूजेशन द्वारा पॉलीसोम को अलग करने के लिए किया जाता है। पॉलीसोम से जुड़े एमआरएनए (पीएआर) को पुन: निलंबन के बाद पॉलीसोमल छर्रों से शुद्ध किया जाता है। आरएनए-सेक के लिए अनुक्रमण पुस्तकालयों के गुणवत्ता मानकों को पूरा करने के लिए अत्यधिक संवेदनशील केशिका वैद्युतकणसंचलन द्वारा कुल और पीएआर दोनों का मूल्यांकन किया जाता है। डाउनस्ट्रीम एप्लिकेशन के उदाहरण के रूप में, अनुक्रमण के बाद, जीन अभिव्यक्ति विश्लेषण के लिए मानक पाइपलाइनों का उपयोग ट्रांसक्रिप्टोम और ट्रांसलेटोम स्तरों पर अलग-अलग व्यक्त जीन प्राप्त करने के लिए किया जा सकता है। संक्षेप में, यह विधि, आरएनए-सेक के साथ संयोजन में, सहजीवी नोड्यूल जैसे जटिल ऊतक में यूकेरियोटिक एमआरएनए के ट्रांसलेशनल विनियमन के अध्ययन की अनुमति देती है।
Introduction
फलीदार पौधे, जैसे सोयाबीन (ग्लाइसिन अधिकतम), राइज़ोबिया नामक विशिष्ट मिट्टी के बैक्टीरिया के साथ सहजीवन स्थापित कर सकते हैं। यह पारस्परिक संबंध पौधे की जड़ों पर उपन्यास अंगों, सहजीवी नोड्यूल के गठन को प्राप्त करता है। नोड्यूल बैक्टीरिया की मेजबानी करने वाले पौधे के अंग होते हैं और इसमें मेजबान कोशिकाएं होती हैं जिनके साइटोप्लाज्म को राइज़ोबिया के एक विशेष रूप के साथ उपनिवेशित किया जाता है जिसे बैक्टीरॉइड कहा जाता है। ये बैक्टीरॉइड वायुमंडलीय नाइट्रोजन (एन2) को अमोनिया में कम करने के लिए उत्प्रेरित करते हैं, जिसे कार्बोहाइड्रेट 1,2 के बदले में पौधे में स्थानांतरित किया जाता है।
यद्यपि यह नाइट्रोजन-फिक्सिंग सहजीवन सबसे अच्छी तरह से अध्ययन किए गए पौधे-सूक्ष्मजीव सहजीवी में से एक है, कई पहलुओं को बेहतर ढंग से समझा जाना बाकी है, जैसे कि विभिन्न अजैविक तनाव स्थितियों के अधीन पौधे अपने सहजीवी साथी के साथ अपनी बातचीत को कैसे संशोधित करते हैं और यह नोड्यूल चयापचय को कैसे प्रभावित करता है। नोड्यूल ट्रांसलेटोम (यानी, मैसेंजर आरएनए [एमआरएनए] के सबसेट का सक्रिय रूप से अनुवाद किया गया) का विश्लेषण करके इन प्रक्रियाओं को बेहतर ढंग से समझा जा सकता है। पॉलीराइबोसोम या पॉलीसोम एमआरएनए से जुड़े कई राइबोसोम के परिसर हैं, जिनका उपयोग आमतौर पर अनुवाद का अध्ययन करने के लिए किया जाताहै 3. पॉलीसोम प्रोफाइलिंग विधि में पॉलीसोम से जुड़े एमआरएनए का विश्लेषण होता है और इसका उपयोग जीन अभिव्यक्ति को नियंत्रित करने वाले पोस्टट्रांसक्रिप्शनल तंत्रका अध्ययन करने के लिए सफलतापूर्वक किया जाता है जो विभिन्न जैविक प्रक्रियाओं 4,5 में होता है।
ऐतिहासिक रूप से, जीनोम अभिव्यक्ति विश्लेषण ने मुख्य रूप से एमआरएनए बहुतायत 6,7,8,9 का निर्धारण करने पर ध्यान केंद्रित किया है। हालांकि, जीन अभिव्यक्ति के पोस्टट्रांसक्रिप्शनल विनियमन के विभिन्न चरणों के कारण प्रतिलेख और प्रोटीन के स्तर के बीच सहसंबंध की कमी है, विशेष रूप से अनुवाद 10,11,12। इसके अलावा, ट्रांसक्रिप्टोम के स्तर पर परिवर्तनों और अनुवाद13 के स्तर पर होने वाले परिवर्तनों के बीच कोई निर्भरता नहीं देखी गई है। एमआरएनए के सेट का प्रत्यक्ष विश्लेषण जिसका अनुवाद किया जा रहा है, सेल जीन अभिव्यक्ति (जिसका समापन बिंदु प्रोटीन बहुतायत है) के अधिक सटीक और पूर्ण माप की अनुमति देता है, जब केवल एमआरएनए स्तरों का विश्लेषण 14,15,16 किया जाता है।
यह प्रोटोकॉल बताता है कि पौधे-व्युत्पन्न पॉलीसोम को दो-परत सुक्रोज कुशन (चित्रा 1) के माध्यम से अंतर सेंट्रीफ्यूजेशन द्वारा बरकरार सोयाबीन नोड्यूल से कैसे शुद्ध किया जाता है। हालांकि, चूंकि बैक्टीरॉइड-व्युत्पन्न राइबोसोम नोड्यूल में भी मौजूद होते हैं, इसलिए राइबोसोम और आरएनए प्रजातियों का मिश्रण शुद्ध होता है, भले ही यूकेरियोटिक वाले मुख्य अंश (90% -95%) का प्रतिनिधित्व करते हैं। बाद के आरएनए अलगाव, मात्रा का ठहराव, और गुणवत्ता नियंत्रण भी वर्णित हैं (चित्रा 1)। यह प्रोटोकॉल, आरएनए-सेक के साथ संयोजन में, सहजीवी नोड्यूल जैसे जटिल ऊतक में यूकेरियोटिक एमआरएनए के ट्रांसलेशनल विनियमन पर प्रयोगात्मक परिणाम प्रदान करना चाहिए।
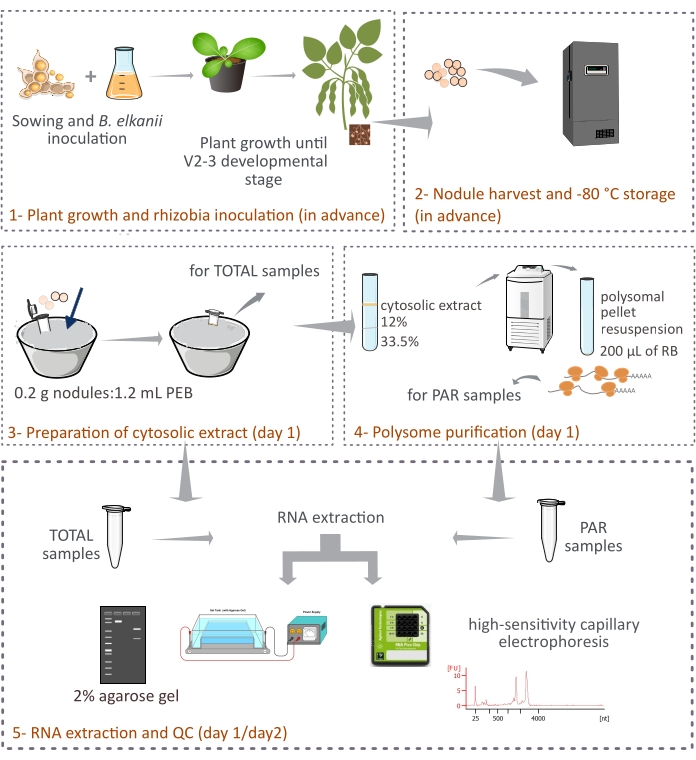
चित्रा 1: सहजीवी नोड्यूल से यूकेरियोटिक पॉलीसोम शुद्धिकरण के लिए प्रस्तावित पद्धति का योजनाबद्ध अवलोकन। योजना (1) पौधे की वृद्धि और (2) नोड्यूल फसल से (3) साइटोसोलिक अर्क की तैयारी, (3) कुल नमूने और (4) पीएआर नमूने प्राप्त करने, और (5) आरएनए निष्कर्षण और गुणवत्ता नियंत्रण से प्रोटोकॉल में पीछा किए गए चरणों का अवलोकन देती है। संक्षिप्त नाम: पीईबी = पॉलीसोम निष्कर्षण बफर; आरबी = पुन: निलंबन बफर; कुल = कुल आरएनए; पीएआर = पॉलीसोम से जुड़े एमआरएनए। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें।
Protocol
Representative Results
Discussion
ट्रांसलेशनल स्तर पर जीन अभिव्यक्ति विनियमन का अध्ययन विभिन्न जैविक प्रक्रियाओं को बेहतर ढंग से समझने के लिए महत्वपूर्ण है क्योंकि सेल जीन अभिव्यक्ति का समापन बिंदु प्रोटीन बहुतायत13,14<sup …
Declarações
The authors have nothing to disclose.
Acknowledgements
इस शोध को सीएसआईसी आई + डी 2020 अनुदान संख्या 282, एफवीएफ 2017 अनुदान संख्या 210 और पीईडीईसीआईबीए (मारिया मार्था सैंज) द्वारा वित्त पोषित किया गया था।
Materials
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
Referências
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer’s disease model mice. Journal of Alzheimer’s Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).

