Purificazione dei polisomi dai noduli simbiotici di soia
Summary
Questo protocollo descrive un metodo per la purificazione dei polisomi eucariotici da noduli di soia intatti. Dopo il sequenziamento, le pipeline standard per l’analisi dell’espressione genica possono essere utilizzate per identificare geni differenzialmente espressi a livello di trascrittoma e translatoma.
Abstract
Lo scopo di questo protocollo è quello di fornire una strategia per lo studio del translatoma eucariotico del nodulo simbiotico di soia (Glycine max). Questo articolo descrive metodi ottimizzati per isolare i poliribosomi di origine vegetale e i loro mRNA associati da analizzare utilizzando il sequenziamento dell’RNA. In primo luogo, i lisati citoplasmatici sono ottenuti attraverso l’omogeneizzazione in condizioni di conservazione di polisomi e RNA da noduli di soia interi e congelati. Quindi, i lisati vengono eliminati mediante centrifugazione a bassa velocità e il 15% del surnatante viene utilizzato per l’isolamento totale dell’RNA (TOTAL). Il restante lisato eliminato viene utilizzato per isolare i polisomi mediante ultracentrifugazione attraverso un cuscino di saccarosio a due strati (12% e 33,5%). L’mRNA associato ai polisomi (PAR) viene purificato dai pellet polisomiali dopo la risospensione. Sia TOTAL che PAR sono valutati mediante elettroforesi capillare altamente sensibile per soddisfare gli standard di qualità delle librerie di sequenziamento per RNA-seq. Come esempio di applicazione a valle, dopo il sequenziamento, è possibile utilizzare pipeline standard per l’analisi dell’espressione genica per ottenere geni differenzialmente espressi a livello di trascrittoma e translatoma. In sintesi, questo metodo, in combinazione con RNA-seq, permette lo studio della regolazione traslazionale degli mRNA eucariotici in un tessuto complesso come il nodulo simbiotico.
Introduction
Le leguminose, come la soia (Glycine max), possono stabilire simbiosi con specifici batteri del suolo chiamati rizobia. Questa relazione mutualistica provoca la formazione di nuovi organi, i noduli simbiotici, sulle radici delle piante. I noduli sono gli organi vegetali che ospitano i batteri e sono costituiti da cellule ospiti il cui citoplasma è colonizzato con una forma specializzata di rizobia chiamata batterioidi. Questi batterioidi catalizzano la riduzione dell’azoto atmosferico (N 2) in ammoniaca, che viene trasferita alla pianta in cambio di carboidrati 1,2.
Sebbene questa simbiosi azotofissatrice sia una delle simbiosi pianta-microbo più studiate, molti aspetti rimangono da comprendere meglio, come il modo in cui le piante sottoposte a diverse condizioni di stress abiotico modulano la loro interazione con il loro partner simbiotico e come questo influisce sul metabolismo dei noduli. Questi processi potrebbero essere meglio compresi analizzando il translatoma dei noduli (cioè, il sottoinsieme di RNA messaggeri [mRNA] attivamente tradotti). I poliribosomi o polisomi sono complessi di ribosomi multipli associati all’mRNA, comunemente usati per studiare la traduzione3. Il metodo di profilazione dei polisomi consiste nell’analisi degli mRNA associati ai polisomi ed è stato utilizzato con successo per studiare i meccanismi posttrascrizionali che controllano l’espressione genica che avviene in diversi processi biologici 4,5.
Storicamente, l’analisi dell’espressione del genoma si è concentrata principalmente sulla determinazione dell’abbondanza di mRNA 6,7,8,9. Tuttavia, vi è una mancanza di correlazione tra i livelli di trascrizione e proteina a causa delle diverse fasi di regolazione posttrascrizionale dell’espressione genica, in particolare la traduzione10,11,12. Inoltre, non è stata osservata alcuna dipendenza tra i cambiamenti a livello del trascrittoma e quelli che si verificano a livello del translatoma13. L’analisi diretta dell’insieme di mRNA che vengono tradotti consente una misura più accurata e completa dell’espressione genica cellulare (il cui endpoint è l’abbondanza proteica) rispetto a quella ottenuta quando vengono analizzati solo i livelli di mRNA14,15,16.
Questo protocollo descrive come i polisomi di origine vegetale vengono purificati dai noduli di soia intatti mediante centrifugazione differenziale attraverso un cuscino di saccarosio a due strati (Figura 1). Tuttavia, poiché nei noduli sono presenti anche ribosomi di derivazione batterioide, viene purificato un mix di ribosomi e specie di RNA, anche se quelli eucariotici rappresentano la frazione principale (90%-95%). Vengono inoltre descritti il successivo isolamento, quantificazione e controllo di qualità dell’RNA (Figura 1). Questo protocollo, in combinazione con RNA-seq, dovrebbe fornire risultati sperimentali sulla regolazione traslazionale degli mRNA eucariotici in un tessuto complesso come il nodulo simbiotico.
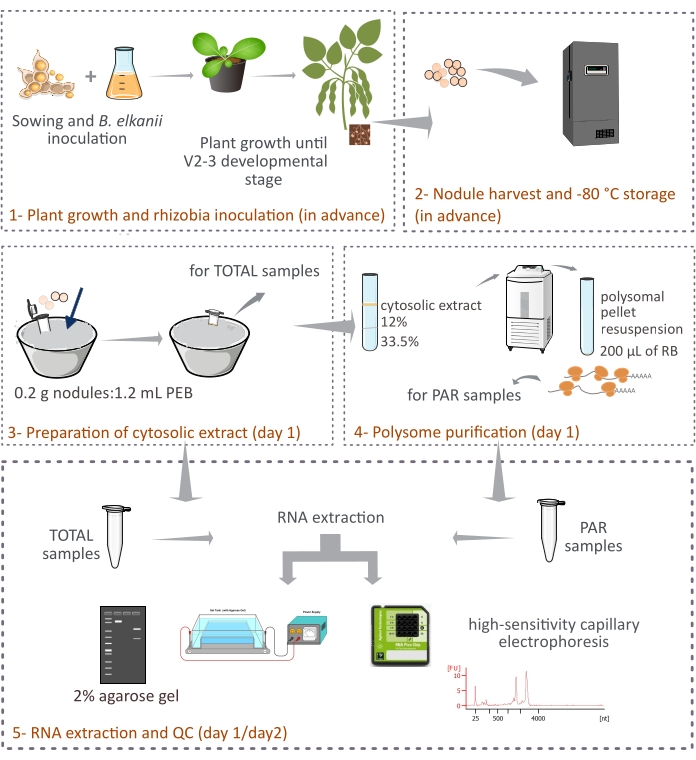
Figura 1: Panoramica schematica della metodologia proposta per la purificazione dei polisomi eucariotici da noduli simbiotici. Lo schema fornisce una panoramica delle fasi seguite nel protocollo da (1) crescita delle piante e (2) raccolta di noduli a (3) preparazione degli estratti citosolici, (3) ottenimento di campioni TOTAL e (4) campioni PAR e (5) estrazione dell’RNA e controllo di qualità. Abbreviazioni: PEB = tampone di estrazione dei polisomi; RB = buffer di risospensione; TOTALE = RNA totale; PAR = mRNA associato ai polisomi. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Protocol
Representative Results
Discussion
Studiare la regolazione dell’espressione genica a livello traslazionale è fondamentale per comprendere meglio i diversi processi biologici poiché l’endpoint dell’espressione genica cellulare è l’abbondanza proteica13,14. Questo può essere valutato analizzando il translatoma del tessuto o dell’organismo di interesse per il quale la frazione polisomiale deve essere purificata e i suoi mRNA associati analizzati 3,4,34,35,36.<su…
Declarações
The authors have nothing to disclose.
Acknowledgements
Questa ricerca è stata finanziata dalla sovvenzione CSIC I + D 2020 n. 282, dalla sovvenzione FVF 2017 n. 210 e PEDECIBA (María Martha Sainz).
Materials
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
Referências
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer’s disease model mice. Journal of Alzheimer’s Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).

