Purificação polissóbica de nódulos simbióticos de soja
Summary
Este protocolo descreve um método de purificação de pórsome eucariótico a partir de nódulos de soja intactos. Após o sequenciamento, os dutos padrão para análise de expressão genética podem ser usados para identificar genes expressos diferencialmente nos níveis de transcriptome e translatome.
Abstract
O objetivo deste protocolo é fornecer uma estratégia para estudar o traduzindo eucariótico do nódulo simbiótico de soja (Glycine max). Este artigo descreve métodos otimizados para isolar poliribos derivados de plantas e seus mRNAs associados a serem analisados usando sequenciamento de RNA. Em primeiro lugar, os licoplasatos citoplasmados são obtidos através da homogeneização em condições de preservação de poliso e RNA de nódulos inteiros e congelados de soja. Em seguida, os lysates são limpos por centrifugação de baixa velocidade, e 15% do supernante é usado para o isolamento total do RNA (TOTAL). O restante do lise limpo é usado para isolar pórsomeos por ultracentrifugação através de uma almofada de sacarose de duas camadas (12% e 33,5%). O mRNA (PAR) associado ao polissomo é purificado a partir de pelotas polissonais após a ressuspensão. Tanto total quanto PAR são avaliados por eletroforese capilar altamente sensível para atender aos padrões de qualidade das bibliotecas de sequenciamento para RNA-seq. Como exemplo de uma aplicação a jusante, após o sequenciamento, os pipelines padrão para análise de expressão genética podem ser usados para obter genes expressos diferencialmente nos níveis de transcriptome e tradução. Em resumo, este método, em combinação com o RNA-seq, permite o estudo da regulação translacional de mRNAs eucarióticas em um tecido complexo como o nódulo simbiótico.
Introduction
Plantas leguminous, como a soja (Glycine max), podem estabelecer simbiose com bactérias específicas do solo chamadas rizobia. Essa relação mutualista provoca a formação de novos órgãos, os nódulos simbióticos, nas raízes das plantas. Os nódulos são os órgãos vegetais que hospedam as bactérias e consistem em células hospedeiras cujo citoplasma é colonizado com uma forma especializada de rizobia chamada bacteroides. Esses bacteroides catalisam a redução do nitrogênio atmosférico (N2) em amônia, que é transferida para a planta em troca de carboidratos 1,2.
Embora essa simbiose fixador de nitrogênio seja uma das simbioses vegetais mais bem estudadas, muitos aspectos continuam sendo melhor compreendidos, como como as plantas submetidas a diferentes condições de estresse abiótico modulam sua interação com seu parceiro simbiótico e como isso afeta o metabolismo do nódulo. Esses processos poderiam ser melhor compreendidos analisando o translatome de nódulo (ou seja, o subconjunto de RNAs mensageiros [mRNAs] traduzido ativamente). Poliribosomes ou polissomos são complexos de ribossomos múltiplos associados ao mRNA, comumente usado para estudar a tradução3. O método de perfil polisome consiste na análise dos mRNAs associados aos polissomos e tem sido utilizado com sucesso para estudar os mecanismos pós-transcriionais que controlam a expressão genética que ocorre em diversos processos biológicos 4,5.
Historicamente, a análise da expressão do genoma tem focado principalmente na determinação da abundância de mRNA 6,7,8,9. No entanto, há falta de correlação entre a transcrição e os níveis de proteína devido aos diferentes estágios de regulação pós-escrita da expressão genética, particularmente a tradução 10,11,12. Além disso, não foi observada dependência entre as alterações no nível do transcriptome e aquelas que ocorrem no nível do traduzome13. A análise direta do conjunto de mRNAs que estão sendo traduzidos permite uma medição mais precisa e completa da expressão genética celular (cujo ponto final é a abundância de proteínas) do que a obtida quando apenas os níveis de mRNA são analisados 14,15,16.
Este protocolo descreve como os polimentos derivados das plantas são purificados de nódulos de soja intactos por centrifugação diferencial através de uma almofada de sacarose de duas camadas (Figura 1). No entanto, uma vez que os ribossomos derivados de bacteroides também estão presentes nos nódulos, uma mistura de ribossomos e espécies de RNA são purificadas, embora as eucarióticas representem a fração principal (90%-95%). O subsequente isolamento do RNA, quantificação e controle de qualidade também são descritos (Figura 1). Este protocolo, em combinação com o RNA-seq, deve fornecer resultados experimentais sobre a regulação translacional de mRNAs eucarióticas em um tecido complexo como o nódulo simbiótico.
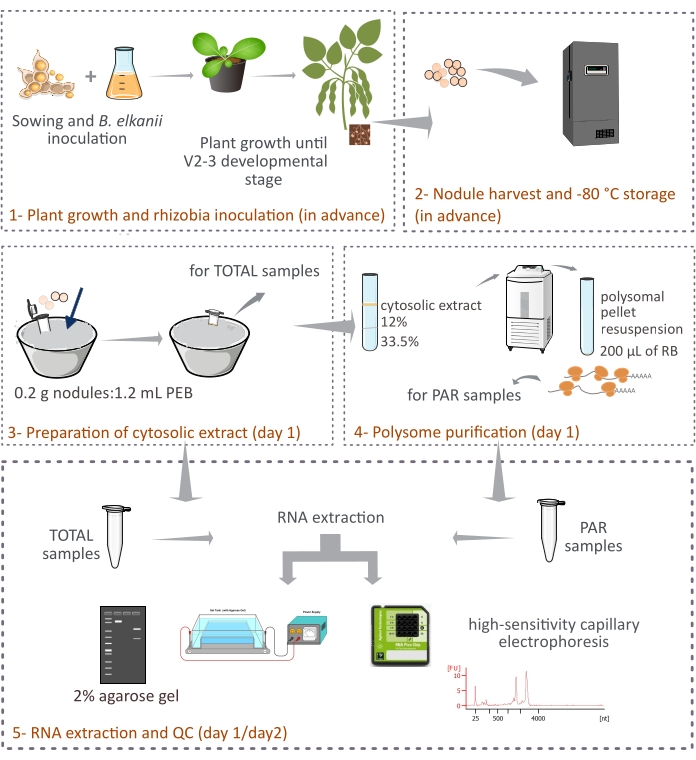
Figura 1: Visão geral esquemática da metodologia proposta para purificação de pórsomee eucariótico a partir de nódulos simbióticos. O esquema fornece uma visão geral das etapas seguidas no protocolo de (1) crescimento vegetal e (2) colheita de nódulos até (3) preparação dos extratos citolíticos, (3) obtenção de amostras TOTAIS e (4) amostras PAR e (5) extração e controle de qualidade do RNA. Abreviaturas: PEB = tampão de extração políduo; RB = tampão de resuspensão; TOTAL = RNA total; PAR = mRNA associado ao polissomo. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
Estudar a regulação da expressão genética no nível translacional é fundamental para compreender melhor diferentes processos biológicos, uma vez que o ponto final da expressão genética celular é a abundância deproteínas 13,14. Isso pode ser avaliado analisando o translatório do tecido ou organismo de interesse para o qual a fração polissômica deve ser purificada e seus mRNAs associados analisaram 3,4,34,35,36.<sup…
Declarações
The authors have nothing to disclose.
Acknowledgements
Esta pesquisa foi financiada pela bolsa CSIC I+D 2020 nº 282, FVF 2017 grant No. 210 e PEDECIBA (María Martha Sainz).
Materials
| Plant growth and rhizobia inoculation | |||
| Orbital shaker | Daihan Scientific | Model SHO-1D | |
| YEM-medium | Amresco | J850 (yeast extract) 0122 (mannitol) | |
| Water deficit treatment | |||
| KNO3 | Merck | 221295 | |
| Porometer | Decagon Device | Model SC-1 | |
| Scalpel | |||
| Preparation of cytosolic extracts | |||
| Brij L23 | Sigma-Aldrich | P1254 | |
| Centrifuge | Sigma | Model 2K15 | |
| Chloranphenicol | Sigma-Aldrich | C0378 | |
| Cycloheximide | Sigma-Aldrich | C7698 | |
| DOC | Sigma-Aldrich | 30970 | |
| DTT | Sigma-Aldrich | D9779 | |
| EGTA | Sigma-Aldrich | E3889 | |
| Igepal CA 360 | Sigma-Aldrich | I8896 | |
| KCl | Merck | 1.04936 | |
| MgCl2 | Sigma-Aldrich | M8266 | |
| Plastic tissue grinder | Fisher Scientific | 12649595 | |
| PMSF | Sigma-Aldrich | P7626 | |
| PTE | Sigma-Aldrich | P2393 | |
| Tris | Invitrogen | 15504-020 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Weighing dish | Deltalab | 1911103 | |
| Preparation of sucrose cushions | |||
| Sucrose | Invitrogen | 15503022 | |
| SW 40 Ti rotor | Beckman-Coulter | ||
| Ultracentrifuge | Beckman-Coulter | Optima L-100K | |
| Ultracentrifuge tubes | Beckman-Coulter | 344059 | 13.2 mL tubes |
| RNA extraction and quality control | |||
| Agarose | Thermo scientific | R0492 | |
| Bioanalyzer | Agilent | Model 2100. Eukaryote total RNA nano assay | |
| Chloroform | DI | 41191 | |
| Ethanol | Dorwil | UN1170 | |
| Isopropanol | Mallinckrodt | 3032-06 | |
| Glycogen | Sigma | 10814-010 | |
| TRIzol LS | Ambion | 102960028 | |
| Miscellaneous | |||
| Falcon tubes 15 mL | Biologix | 10-0152 | |
| Filter tips 10 µL | BioPointe Scientific | 321-4050 | |
| Filter tips 1000 µL | BioPointe Scientific | 361-1050 | |
| Filter tips 20 µL | BioPointe Scientific | 341-4050 | |
| Filter tips 200 µL | Tarsons | 528104 | |
| Microcentrifuge tubes 1.5 mL | Tarsons | 500010-N | |
| Microcentrifuge tubes 2.0 mL | Tarsons | 500020-N | |
| Sequencing company | Macrogen | ||
| Sterile 250 mL flask | Marienfeld | 4110207 |
Referências
- Limpens, E., et al. Cell- and tissue-specific transcriptome analyses of Medicago truncatula root nodules. PLoS ONE. 8 (5), 64377 (2013).
- Masson-Boivin, C., Giraud, E., Perret, X., Batut, J. Establishing nitrogen-fixing symbiosis with legumes: How many rhizobium recipes. Trends in Microbiology. 17 (10), 458-466 (2009).
- King, H. A., Gerber, A. P. Translatome profiling: Methods for genome-scale analysis of mRNA translation. Briefings in Functional Genomics. 15 (1), 22-31 (2014).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Yángüez, E., Castro-Sanz, A. B., Fernández-Bautista, N., Oliveros, J. C., Castellano, M. M. Analysis of genome-wide changes in the translatome of Arabidopsis seedlings subjected to heat stress. PloS ONE. 8 (8), 71425 (2013).
- Brown, P. O., Botstein, D. Exploring the new world of the genome with DNA microarrays. Nature Genetics. 21, 33-37 (1999).
- Krishnamurthy, A., Ferl, R. J., Paul, A. -. L. Comparing RNA-Seq and microarray gene expression data in two zones of the Arabidopsis root apex relevant to spaceflight. Applications in Plant Sciences. 6 (11), 1197 (2018).
- Shulse, C. N., et al. High-throughput single-cell transcriptome profiling of plant cell types. Cell Reports. 27 (7), 2241-2247 (2019).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Reviews Genetics. 10 (1), 57-63 (2009).
- Kawaguchi, R., Girke, T., Bray, E. A., Bailey-Serres, J. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. The Plant Journal. 38 (5), 823-839 (2004).
- Larsson, O., Tian, B., Sonenberg, N. Toward a genome-wide landscape of translational control. Cold Spring Harbor Perspectives in Biology. 5 (1), 012302 (2013).
- Vogel, C., Marcotte, E. M. Insights into regulation of protein abundance from proteomics and transcriptomis analyses. Nature Reviews Genetics. 13 (4), 227-232 (2013).
- Tebaldi, T., et al. Widespread uncoupling between transcriptome and translatome variations after a stimulus in mammalian cells. BMC Genomics. 13 (220), 1-15 (2012).
- Wang, T., et al. Translating mRNAs strongly correlate to proteins in a multivariate manner and their translation ratios are phenotype specific. Nucleic Acids Research. 41 (9), 4743-4754 (2013).
- Ingolia, N. T. Ribosome profiling: New views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Lukoszek, R., Feist, P., Ignatova, Z. Insights into the adaptive response of Arabidopsis thaliana to prolonged thermal stress by ribosomal profiling and RNA-Seq. BMC Plant Biology. 16 (1), 1-13 (2016).
- Broughton, W. J., Dilworth, M. J. Control of leghaemoglobin synthesis in snake beans. Biochemical Journal. 125 (4), 1075-1080 (1971).
- Schroeder, A., et al. The RIN: An RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7, 1-14 (2006).
- Rio, D. C., Ares, M., Hannon, G. J., Nilsen, T. W. Nondenaturing agarose gel electrophoresis of RNA nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harbor Protocols. 2010 (6), 1-3 (2012).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: Summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Marcel, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- . Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) Available from: https://github.com/najoshi/sickle (2011)
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Kim, D., et al. TopHat2: Accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biology. 14 (4), 1-13 (2013).
- Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., Kingsford, C. Salmon provides fast and bias-aware quantification of transcript expression. Nature Methods. 14 (4), 417-419 (2017).
- Liao, Y., Smyth, G. K., Shi, W. featureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (550), 1-21 (2014).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2015).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2018 update. Nucleic Acids Research. 46, 537-544 (2018).
- Hunt, G. P., et al. GEOexplorer: A webserver for gene expression analysis and visualisation. Nucleic Acids Research. , (2022).
- Imbeaud, S., et al. Towards standardization of RNA quality assessment using user-independent classifiers of microcapillary electrophoresis traces. Nucleic Acids Research. 33 (6), 1-12 (2005).
- Di Paolo, A., et al. PDCD4 regulates axonal growth by translational repression of neurite growth-related genes and is modulated during nerve injury responses. RNA. 26 (11), 1637-1653 (2020).
- Smircich, P., et al. Ribosome profiling reveals translation control as a key mechanism generating differential gene expression in Trypanosoma cruzi. BMC Genomics. 16 (1), 443 (2015).
- Eastman, G., Sharlow, E. R., Lazo, J. S., Bloom, G. S., Sotelo-Silveira, J. R. Transcriptome and translatome regulation of pathogenesis in Alzheimer’s disease model mice. Journal of Alzheimer’s Disease. 86 (1), 365-386 (2022).
- Zanetti, M. E., Chang, I. -. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification of polyribosomal complexes of Arabidopsis for global analysis of gene expression. Plant physiology. 138 (2), 624-635 (2005).
- Mustroph, A., et al. Profiling translatomes of discrete cell populations resolves altered cellular priorities during hypoxia in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18843-11848 (2009).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature Reviews. Molecular Cell Biology. 16 (11), 651-664 (2015).
- Eastman, G., Smircich, P., Sotelo-Silveira, J. R. Following ribosome footprints to understand translation at a genome wide level. Computational and Structural Biotechnology Journal. 16, 167-176 (2018).
- Westermann, A. J., Gorski, S. A., Vogel, J. Dual RNA-seq of pathogen and host. Nature Reviews Microbiology. 10 (9), 618-630 (2012).

