Gen digitale kredsløb baseret på CRISPR-Cas systemer og anti-CRISPR proteiner
Summary
CRISPR-Cas-systemer og anti-CRISPR-proteiner blev integreret i ordningen med boolske porte i Saccharomyces cerevisiae. De nye små logiske kredsløb viste god ydeevne og uddybede forståelsen af både dCas9/dCas12a-baserede transkriptionsfaktorer og egenskaberne af anti-CRISPR-proteiner.
Abstract
Syntetiske gen boolske porte og digitale kredsløb har en bred vifte af applikationer, fra medicinsk diagnostik til miljøpleje. Opdagelsen af CRISPR-Cas-systemerne og deres naturlige hæmmere – anti-CRISPR-proteinerne (Acrs) – giver et nyt værktøj til at designe og implementere in vivo genets digitale kredsløb. Her beskriver vi en protokol, der følger ideen om “Design-Build-Test-Learn” biologisk ingeniørcyklus og gør brug af dCas9 / dCas12a sammen med deres tilsvarende Acrs til at etablere små transkriptionelle netværk, hvoraf nogle opfører sig som boolske porte, i Saccharomyces cerevisiae. Disse resultater påpeger egenskaberne af dCas9/dCas12a som transkriptionsfaktorer. Især for at opnå maksimal aktivering af genekspression skal dSpCas9 interagere med et konstrueret stillads-RNA, der indsamler flere kopier af VP64-aktiveringsdomænet (AD). I modsætning hertil skal dCas12a ved C-terminalen smeltes sammen med den stærke VP64-p65-Rta (VPR) AD. Desuden forbedres aktiviteten af begge Cas-proteiner ikke ved at øge mængden af sgRNA/crRNA i cellen. Denne artikel forklarer også, hvordan man bygger boolske porte baseret på CRISPR-dCas-Acr-interaktionen. Det AcrIIA4-smeltede hormonbindende domæne for den humane østrogenreceptor er kernen i en NOT-port, der reagerer på β-østradiol, mens AcrVA’er syntetiseret af den inducerbare GAL1-promotor tillader at efterligne både JA og IKKE porte med galactose som input. I sidstnævnte kredsløb viste AcrVA5 sammen med dLbCas12a den bedste logiske adfærd.
Introduction
I 2011 foreslog forskere en beregningsmetode og udviklede et tilsvarende stykke software til automatisk design af digitale syntetiske genkredsløb1. En bruger skulle angive antallet af indgange (tre eller fire) og udfylde kredsløbets sandhedstabel; Dette gav alle de nødvendige oplysninger til at udlede kredsløbsstrukturen ved hjælp af teknikker fra elektronik. Sandhedstabellen blev oversat til to boolske formler via Karnaugh-kortets metode2. Hver boolsk formel er lavet af klausuler, der beskriver logiske operationer (sum eller multiplikation) blandt (en del af) kredsløbsindgange og deres negationer (de bogstavelige). Klausuler er igen enten opsummeret (OR) eller ganget (AND) for at beregne kredsløbsudgangen. Hvert kredsløb kan realiseres i henhold til en af dets to tilsvarende formler: den ene skrevet i POS (produkt af summer) form og den anden i SOP (sum af produkter) repræsentation. Førstnævnte består af en multiplikation af klausuler (dvs. boolske porte), der indeholder en logisk sum af de bogstavelige. Sidstnævnte er derimod en sum af klausuler, hvor de bogstavelige multipliceres.
Elektriske kredsløb kan realiseres på et brødbræt ved fysisk at forbinde forskellige porte sammen. Den elektriske strøm tillader udveksling af signaler mellem porte, hvilket fører til beregning af udgangen.
I biologi er situationen mere kompleks. En boolsk gate kan realiseres som en transkriptionsenhed (TU; dvs. sekvensen “promotorkodende regionterminator” inde i eukaryote celler), hvor transkription eller oversættelse (eller begge) reguleres. Således etablerer mindst to slags molekyler en biologisk ledning: transkriptionsfaktorproteinerne og de ikke-kodende, antisense RNA’er1.
Et gendigitalt kredsløb er organiseret i to eller tre lag porte, nemlig: 1) inputlaget, der er lavet af JA (buffer) og IKKE porte og omdanner inputkemikalierne til ledningsmolekyler; 2) det indre lag, der består af så mange TU’er, som der er klausuler i den tilsvarende boolske formel. Hvis kredsløbet er designet i henhold til SOP-formlen, vil hver klausul i det indre lag producere kredsløbsudgangen (f.eks. Fluorescens) i en såkaldt distribueret outputarkitektur. Hvis produktet af sum (POS) formel anvendes, kræves der et 3) sidste lag, som vil indeholde en enkelt multiplikativ port, der samler ledningsmolekylerne fra det indre lag.
Samlet set kan der i syntetisk biologi designes mange forskellige ordninger til det samme kredsløb. De adskiller sig i antallet og typen af både TU’er og ledningsmolekyler. For at vælge den nemmeste løsning, der skal implementeres i gærceller, er hvert kredsløbsdesign forbundet med en kompleksitetsscore S, defineret som
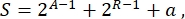
hvor A repræsenterer antallet af aktivatorer, R repræsenterer antallet af repressorer, og a er mængden af antisense RNA-molekyler. Hvis enten aktivatorer eller repressorer er fraværende fra kredsløbet, er deres bidrag til S nul. Derfor er det vanskeligere at realisere et kredsløbsskema i laboratoriet (højt S), når det kræver et stort antal ortogonale transkriptionsfaktorer. Dette betyder, at nye aktivatorer og repressorer skal konstrueres de novo for at realisere den komplette ledning inde i de digitale kredsløb. I princippet kan nye DNA-bindende proteiner samles ved hjælp af zinkfingerproteiner3 og TAL-effektorer4 som skabeloner. Denne mulighed virker imidlertid for besværlig og tidskrævende; derfor bør man hovedsagelig stole på små RNA’er og translationsregulering for at færdiggøre komplekse genkredsløb.
Oprindeligt blev denne metode udviklet til at fremstille digitale kredsløb i bakterier. Faktisk er det i eukaryote celler i stedet for antisense-RNA’er mere egnet at tale om mikroRNA’er (miRNA’er) eller små interfererende RNA’er (siRNA’er)5. RNAi-vejen er imidlertid ikke til stede i gæren S. cerevisiae. Derfor bør man vælge fuldt transkriptionsnetværk. Antag, at et kredsløb har brug for fem aktivatorer og fem undertrykkere; dens kompleksitetsscore ville være S = 32. Kredsløbskompleksiteten kan reduceres ved at erstatte de 10 transkriptionsfaktorer med en enkelt dCas96 (nukleasemangel Cas9) smeltet sammen til et aktiveringsdomæne (AD). Som vist i7 fungerer dCas9-AD som en repressor i gær, når der bindes en promotor mellem TATA-boksen og TSS (transkriptionsstartstedet) og som aktivator, når den binder godt opstrøms for TATA-boksen. Således kan man erstatte 10 transkriptionsfaktorer med et enkelt dCas9-AD fusionsprotein og 10 sgRNA’er (single guide RNA’er) for en total kompleksitetsscore på S = 11. Det er hurtigt og nemt at syntetisere ti sgRNA’er, mens samlingen af 10 proteiner som tidligere kommenteret ville kræve meget længere og mere kompliceret arbejde.
Alternativt kan man bruge to ortogonale dCas-proteiner (f.eks. dCas9 og dCas12a): den ene til at smelte sammen til en AD, og den anden bar eller i kombination med et undertrykkelsesdomæne. Kompleksitetsscoren ville kun stige med én enhed (S = 12). Derfor er CRISPR-dCas-systemer nøglen til konstruktionen af meget indviklede gendigitale kredsløb i S. cerevisiae.
Dette papir karakteriserer dybt effektiviteten af både dCas9- og dCas12a-baserede repressorer og aktivatorer i gær. Resultaterne viser, at de ikke kræver en høj mængde sgRNA for at optimere deres aktivitet, så episomale plasmider undgås fortrinsvis. Desuden er dCas9-baserede aktivatorer langt mere effektive, når man bruger et stillads-RNA (scRNA), der rekrutterer kopier af VP64 AD. I modsætning hertil fungerer dCas12a godt, når den smeltes direkte sammen med den stærke VPR AD. Desuden kræver en syntetisk aktiveret promotor et variabelt antal målsteder afhængigt af aktivatorens konfiguration (f.eks. tre ved brug af dCas12a-VPR, seks for dCas9-VP64 og kun en med dCas9 og et scRNA). Som repressor virker dCas12a mere skarp, når man binder kodningsregionen frem for promotoren.
Som en ulempe interagerer CRISPR-dCas9/dCas12a dog ikke direkte med kemikalier. Derfor er de muligvis ikke til nogen nytte i inputlaget. Af denne grund er alternative boolske gate-design, der indeholder anti-CRISPR-proteiner (Acrs), blevet undersøgt. Acrs virker på (d)Cas-proteiner og hæmmer deres arbejde8. Derfor er de et middel til at modulere aktiviteten af CRISPR-(d)Cas-systemer. Dette papir analyserer grundigt interaktionerne mellem type II Acrs og (d) Cas9, såvel som type V Acrs og (d) Cas12a i S. cerevisiae. Da Acrs er meget mindre end Cas-proteiner, blev en NOT-port, der reagerer på østrogen-β-østradiol, bygget ved at fusionere det hormonbindende domæne af den humane østrogenreceptor9-HBD (hER) til AcrIIA4. Desuden blev en håndfuld JA og IKKE porte, der udtrykte dCas12a (-AD) konstitutivt og AcrVA’er ved induktion med galactose, realiseret. På nuværende tidspunkt tjener disse porte kun som et bevis på konceptet. Men de repræsenterer også det første skridt mod en dyb nytænkning af algoritmen til at udføre det beregningsmæssige automatiske design af syntetiske gendigitale kredsløb i gærceller.
Protocol
Representative Results
Discussion
Protokollen viste en mulig komplet arbejdsgang for syntetiske gendigitale kredsløb efter “Design-Build-Test-Learn” (DBTL) biologisk ingeniørcyklus og vedrørende både tørlaboratorie- og vådlaboratorieeksperimenter. Her fokuserede vi på CRISPR-Cas-systemet, hovedsageligt dSpCas9, denAsCas12a, dLbCas12a og de tilsvarende anti-CRISPR-proteiner, ved at designe og bygge i S. cerevisiae små transkriptionelle netværk. Nogle af dem efterlignede boolske porte, som er de grundlæggende komponenter i digitale kreds…
Declarações
The authors have nothing to disclose.
Acknowledgements
Vi vil gerne takke alle studerende på Synthetic Biology lab-SPST, TJU-for deres generelle hjælp, sammen med Zhi Li og Xiangyang Zhang for deres hjælp i FACS-eksperimenter.
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
Referências
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

