Gene digitale circuits op basis van CRISPR-Cas-systemen en anti-CRISPR-eiwitten
Summary
CRISPR-Cas-systemen en anti-CRISPR-eiwitten werden geïntegreerd in het schema van Booleaanse poorten in Saccharomyces cerevisiae. De nieuwe kleine logische circuits toonden goede prestaties en verdiepten het begrip van zowel op dCas9/dCas12a gebaseerde transcriptiefactoren als de eigenschappen van anti-CRISPR-eiwitten.
Abstract
Synthetisch gen Booleaanse poorten en digitale circuits hebben een breed scala aan toepassingen, van medische diagnostiek tot milieuzorg. De ontdekking van de CRISPR-Cas-systemen en hun natuurlijke remmers – de anti-CRISPR-eiwitten (Acrs) – biedt een nieuw hulpmiddel om in vivo digitale gencircuits te ontwerpen en te implementeren. Hier beschrijven we een protocol dat het idee van de “Design-Build-Test-Learn” biologische engineeringcyclus volgt en gebruik maakt van dCas9 / dCas12a samen met hun bijbehorende Acrs om kleine transcriptionele netwerken op te zetten, waarvan sommige zich gedragen als Booleaanse poorten, in Saccharomyces cerevisiae. Deze resultaten wijzen op de eigenschappen van dCas9/dCas12a als transcriptiefactoren. In het bijzonder, om maximale activering van genexpressie te bereiken, moet dSpCas9 interageren met een gemanipuleerd steiger-RNA dat meerdere kopieën van het VP64-activeringsdomein (AD) verzamelt. Daarentegen wordt dCas12a bij het C-eindpunt gefuseerd met het sterke VP64-p65-Rta (VPR) AD. Bovendien wordt de activiteit van beide Cas-eiwitten niet versterkt door de hoeveelheid sgRNA/crRNA in de cel te verhogen. In dit artikel wordt ook uitgelegd hoe u Booleaanse poorten kunt bouwen op basis van de CRISPR-dCas-Acr-interactie. Het AcrIIA4 gefuseerde hormoonbindende domein van de menselijke oestrogeenreceptor is de kern van een NOT-poort die reageert op β-oestradiol, terwijl AcrVA’s gesynthetiseerd door de induceerbare GAL1-promotor het mogelijk maken om zowel JA- als NOT-poorten na te bootsen met galactose als input. In de laatste circuits vertoonde AcrVA5, samen met dLbCas12a, het beste logische gedrag.
Introduction
In 2011 stelden onderzoekers een computationele methode voor en ontwikkelden een bijbehorend stuk software voor het automatische ontwerp van digitale synthetische gencircuits1. Een gebruiker moest het aantal ingangen (drie of vier) opgeven en de waarheidstabel van het circuit invullen; Dit leverde alle benodigde informatie op om de circuitstructuur af te leiden met behulp van technieken uit elektronica. De waarheidstabel werd vertaald in twee Booleaanse formules via de Karnaugh-kaartmethode2. Elke Booleaanse formule bestaat uit clausules die logische bewerkingen (som of vermenigvuldiging) beschrijven tussen (een deel van) de circuitingangen en hun negaties (de letterlijke elementen). Clausules worden op hun beurt opgeteld (OF) of vermenigvuldigd (EN) om de circuituitgang te berekenen. Elk circuit kan worden gerealiseerd volgens een van de twee overeenkomstige formules: een geschreven in POS-vorm (product van sommen) en de andere in SOP-representatie (som van producten). De eerste bestaat uit een vermenigvuldiging van clausules (d.w.z. Booleaanse poorten) die een logische som van de letterlijke poorten bevatten. Dit laatste daarentegen is een optelsom van clausules waarbij de letterlijke woorden worden vermenigvuldigd.
Elektrische circuits kunnen worden gerealiseerd, op een breadboard, door verschillende poorten fysiek met elkaar te bedraden. De elektrische stroom maakt de uitwisseling van signalen tussen poorten mogelijk, wat leidt tot de berekening van de uitgang.
In de biologie is de situatie complexer. Een Booleaanse poort kan worden gerealiseerd als een transcriptie-eenheid (TU; d.w.z. de sequentie “promotor-coderende regio-terminator” in eukaryote cellen), waar transcriptie of translatie (of beide) worden gereguleerd. Zo vormen ten minste twee soorten moleculen een biologische bedrading: de transcriptiefactoreiwitten en de niet-coderende, antisense RNA’s1.
Een gen digitaal circuit is georganiseerd in twee of drie lagen poorten, namelijk: 1) de ingangslaag, die is gemaakt van YES (buffer) en NOT poorten en zet de input chemicaliën om in bedradingsmoleculen; 2) de interne laag, die bestaat uit evenveel TU’s als er clausules zijn in de overeenkomstige Booleaanse formule. Als het circuit is ontworpen volgens de SOP-formule, produceert elke clausule in de interne laag de circuituitgang (bijvoorbeeld fluorescentie) in een zogenaamde gedistribueerde uitgangsarchitectuur. Als het product van de som (POS) -formule wordt gebruikt, is een 3) laatste laag vereist, die een enkele multiplicatieve poort bevat die de bedradingsmoleculen van de interne laag verzamelt.
Over het algemeen kunnen in de synthetische biologie veel verschillende schema’s worden ontworpen voor hetzelfde circuit. Ze verschillen in het aantal en het soort van zowel TU’s als bedradingsmoleculen. Om de eenvoudigste oplossing te kiezen die in gistcellen kan worden geïmplementeerd, wordt elk circuitontwerp geassocieerd met een complexiteitsscore S, gedefinieerd als
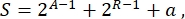
waarbij A staat voor het aantal activatoren, R voor het aantal repressoren en a voor de hoeveelheid antisense RNA-moleculen. Als activators of repressors afwezig zijn in het circuit, is hun bijdrage aan S nul. Daarom is het moeilijker om een circuitschema in het laboratorium (hoge S) te realiseren wanneer het een groot aantal orthogonale transcriptiefactoren vereist. Dit betekent dat nieuwe activatoren en repressors de novo moeten worden ontworpen om de volledige bedrading in de digitale circuits te realiseren. In principe kunnen nieuwe DNA-bindende eiwitten worden samengesteld met behulp van Zinc Finger-eiwitten3 en TAL-effectoren4 als sjablonen. Deze optie lijkt echter te zwaar en tijdrovend; daarom moet men vooral vertrouwen op kleine RNA’s en translatieregulatie om complexe gencircuits te voltooien.
Oorspronkelijk werd deze methode ontwikkeld om digitale schakelingen in bacteriën te fabriceren. Inderdaad, in eukaryote cellen is het in plaats van antisense RNA’s geschikter om te spreken van microRNA’s (miRNA’s) of kleine interfererende RNA’s (siRNA’s)5. De RNAi-route is echter niet aanwezig in de gist S. cerevisiae. Daarom moet men kiezen voor volledig transcriptionele netwerken. Stel dat een circuit vijf activators en vijf repressors nodig heeft; de complexiteitsscore zou S = 32 zijn. De complexiteit van het circuit kan worden verminderd door de 10 transcriptiefactoren te vervangen door een enkele dCas96 (nuclease-deficiënt Cas9) gefuseerd tot een activeringsdomein (AD). Zoals getoond in7, werkt dCas9-AD als een repressor in gist bij het binden van een promotor tussen de TATA-box en de TSS (transcriptiestartplaats) en als een activator bij het goed binden stroomopwaarts van de TATA-box. Zo kan men 10 transcriptiefactoren vervangen door een enkel dCas9-AD fusie-eiwit en 10 sgRNA’s (single guide RNA’s) voor een totale complexiteitsscore van S = 11. Het is snel en gemakkelijk om tien sgRNA’s te synthetiseren, terwijl, zoals eerder opgemerkt, de assemblage van 10 eiwitten veel langer en ingewikkelder werk zou vereisen.
Als alternatief kan men twee orthogonale dCas-eiwitten gebruiken (bijv. dCas9 en dCas12a): één om te fuseren tot een AD, en de andere kaal of in combinatie met een repressiedomein. De complexiteitsscore zou met slechts één eenheid toenemen (S = 12). Vandaar dat CRISPR-dCas-systemen de sleutel zijn tot de constructie van zeer ingewikkelde digitale gencircuits in S. cerevisiae.
Dit artikel karakteriseert diep de efficiëntie van zowel dCas9- als dCas12a-gebaseerde repressoren en activatoren in gist. De resultaten tonen aan dat ze geen hoge hoeveelheid sgRNA nodig hebben om hun activiteit te optimaliseren, dus episomale plasmiden worden bij voorkeur vermeden. Bovendien zijn op dCas9 gebaseerde activatoren veel effectiever bij het gebruik van een steiger-RNA (scRNA) dat kopieën van de VP64 AD rekruteert. Daarentegen werkt dCas12a goed wanneer het rechtstreeks is samengevoegd met de sterke VPR AD. Bovendien vereist een synthetisch geactiveerde promotor een variabel aantal doellocaties, afhankelijk van de configuratie van de activator (bijvoorbeeld drie bij gebruik van dCas12a-VPR, zes voor dCas9-VP64 en slechts één met dCas9 en een scRNA). Als repressor lijkt dCas12a scherper bij het binden van het coderende gebied in plaats van de promotor.
Als nadeel hebben CRISPR-dCas9/dCas12a echter geen directe interactie met chemicaliën. Daarom kunnen ze van geen nut zijn in de invoerlaag. Om deze reden zijn alternatieve Booleaanse poortontwerpen met anti-CRISPR-eiwitten (Acrs) onderzocht. Acrs werken in op (d)Cas-eiwitten en remmen hun werking8. Daarom zijn ze een middel om de activiteit van CRISPR-(d)Cas-systemen te moduleren. Dit artikel analyseert grondig de interacties tussen type II Acrs en (d)Cas9, evenals type V Acrs en (d)Cas12a in S. cerevisiae. Omdat Acrs veel kleiner zijn dan Cas-eiwitten, werd een NOT-poort die reageert op het oestrogeen β-estradiol gebouwd door het hormoonbindende domein van de menselijke oestrogeenreceptor9-HBD (hER) – te fuseren met AcrIIA4. Daarnaast werden een handvol JA- en NOT-poorten gerealiseerd die dCas12a(-AD) constitutief en AcrVA’s uitdrukten bij inductie met galactose. Op dit moment dienen deze poorten alleen als proof of concept. Ze vertegenwoordigen echter ook de eerste stap naar een diepgaande heroverweging van het algoritme om het computationele automatische ontwerp van synthetische gen-digitale circuits in gistcellen uit te voeren.
Protocol
Representative Results
Discussion
Het protocol toonde een mogelijke complete workflow voor synthetische gen-digitale circuits, volgens de “Design-Build-Test-Learn” (DBTL) biologische engineeringcyclus en met betrekking tot zowel dry-lab als wet-lab experimenten. Hier hebben we ons gericht op het CRISPR-Cas-systeem, voornamelijk dSpCas9, denAsCas12a, dLbCas12a en de bijbehorende anti-CRISPR-eiwitten, door kleine transcriptionele netwerken van S. cerevisiae te ontwerpen en in te bouwen. Sommigen van hen bootsten Booleaanse poorten na, die de basis…
Declarações
The authors have nothing to disclose.
Acknowledgements
We willen alle studenten van het Synthetic Biology lab-SPST, TJU-bedanken voor hun algemene hulp, samen met Zhi Li en Xiangyang Zhang voor hun hulp bij FACS-experimenten.
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
Referências
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

