מעגלים דיגיטליים גנטיים המבוססים על מערכות CRISPR-Cas וחלבונים אנטי-קריספר
Summary
מערכות CRISPR-Cas וחלבונים אנטי-קריספר שולבו בתוכנית של שערים בוליאניים בשמרי אפייה (Saccharomyces cerevisiae). המעגלים הלוגיים הקטנים החדשים הראו ביצועים טובים והעמיקו את ההבנה של גורמי שעתוק מבוססי dCas9/dCas12a והתכונות של חלבונים אנטי-קריספר.
Abstract
גנים סינתטיים לשערים בוליאניים ולמעגלים דיגיטליים יש מגוון רחב של יישומים, החל מאבחון רפואי ועד לטיפול סביבתי. גילוי מערכות CRISPR-Cas והמעכבים הטבעיים שלהן – חלבוני האנטי-קריספר (Acrs) – מספק כלי חדש לתכנון ויישום מעגלים דיגיטליים של גנים in vivo . כאן, אנו מתארים פרוטוקול העוקב אחר הרעיון של מחזור ההנדסה הביולוגית “תכנון-בנייה-בדיקה-למידה” ועושה שימוש ב- dCas9/dCas12a יחד עם ה- Acrs המתאימים שלהם כדי ליצור רשתות שעתוק קטנות, שחלקן מתנהגות כמו שערים בוליאניים, ב – Saccharomyces cerevisiae. תוצאות אלה מציינות את המאפיינים של dCas9/dCas12a כגורמי שעתוק. בפרט, כדי להשיג הפעלה מקסימלית של ביטוי גנים, dSpCas9 צריך לקיים אינטראקציה עם RNA פיגום מהונדס שאוסף עותקים מרובים של תחום ההפעלה VP64 (AD). לעומת זאת, dCas12a יאוחה, במסוף C, עם VP64-p65-Rta (VPR) AD חזק. יתר על כן, הפעילות של שני חלבוני Cas אינה משופרת על ידי הגדלת כמות sgRNA/crRNA בתא. מאמר זה גם מסביר כיצד לבנות שערים בוליאניים בהתבסס על אינטראקציית CRISPR-dCas-Acr. תחום קשירת ההורמונים המאוחה AcrIIA4 של קולטן האסטרוגן האנושי הוא הליבה של שער NOT המגיב ל-β-אסטרדיול, בעוד ש-AcrVAs המסונתזים על ידי מקדם GAL1 המושרה מאפשרים לחקות שערי כן ו-NOT עם גלקטוז כקלט. במעגלים האחרונים, AcrVA5, יחד עם dLbCas12a, הראו את ההתנהגות הלוגית הטובה ביותר.
Introduction
בשנת 2011, חוקרים הציעו שיטה חישובית ופיתחו תוכנה מתאימה לתכנון אוטומטי של מעגלי גנים סינתטיים דיגיטליים1. המשתמש היה צריך לציין את מספר הקלטים (שלוש או ארבע) ולמלא את טבלת האמת המעגלית; זה סיפק את כל המידע הדרוש כדי לגזור את מבנה המעגל באמצעות טכניקות מאלקטרוניקה. טבלת האמת תורגמה לשתי נוסחאות בוליאניות בשיטת מפת קרנאו2. כל נוסחה בוליאנית מורכבת ממשפטים המתארים פעולות לוגיות (סכום או כפל) בין (חלק) קלטי המעגל ושלילותיהם (הליטרלים). סעיפים, בתורם, מסוכמים (OR) או מוכפלים (AND) כדי לחשב את פלט המעגל. כל מעגל יכול להתממש על פי כל אחת משתי הנוסחאות המתאימות: האחת כתובה בצורת קופה (מכפלה של סכומים) והשנייה בצורת SOP (סכום מוצרים). הראשון מורכב מכפל של סעיפים (כלומר, שערים בוליאניים) המכילים סכום לוגי של הליטרלים. האחרון, לעומת זאת, הוא סכום של סעיפים שבהם מכפילים את המילוליים.
מעגלים חשמליים יכולים להתממש, על קרש לחם, על ידי חיווט פיזי של שערים שונים יחד. הזרם החשמלי מאפשר חילופי אותות בין השערים, מה שמוביל לחישוב הפלט.
בביולוגיה המצב מורכב יותר. שער בוליאני יכול להתממש כיחידת שעתוק (TU; כלומר, הרצף “מקדם-קידוד אזור-מסיים” בתוך תאים איקריוטים), שבו שעתוק או תרגום (או שניהם) מוסדרים. לפיכך, לפחות שני סוגים של מולקולות יוצרים חיווט ביולוגי: חלבוני גורם השעתוק והרנ”א האנטיסנס שאינו מקודד1.
מעגל דיגיטלי גנטי מאורגן בשתיים או שלוש שכבות של שערים, כלומר: 1) שכבת הקלט, העשויה משערי כן (חיץ) ולא וממירה את כימיקלי הקלט למולקולות חיווט; 2) השכבה הפנימית, המורכבת מכמה TUs שיש סעיפים בנוסחה הבוליאנית המתאימה. אם המעגל מתוכנן על פי נוסחת SOP, כל סעיף בשכבה הפנימית יפיק את פלט המעגל (למשל, פלואורסצנציה) במה שנקרא ארכיטקטורת פלט מבוזר. אם משתמשים בנוסחת המכפלה של סכום (POS), נדרשת שכבה סופית 3), שתכיל שער כפל יחיד האוסף את מולקולות החיווט מהשכבה הפנימית.
בסך הכל, בביולוגיה סינתטית, ניתן לתכנן סכמות רבות ושונות עבור אותו מעגל. הם נבדלים במספר ובסוג של TUs ומולקולות חיווט. על מנת לבחור את הפתרון הקל ביותר ליישום בתאי שמרים, כל תכנון מעגל משויך לציון סיבוכיות S, המוגדר כ
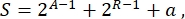
כאשר A מייצג את מספר המפעילים, R מייצג את מספר המדכאים, ו-A הוא כמות מולקולות האנטיסנס RNA. אם מפעילים או מדכאים נעדרים מהמעגל, תרומתם ל-S היא אפס. לכן, קשה יותר לממש סכמת מעגלים במעבדה (S גבוהה) כאשר היא דורשת מספר גבוה של גורמי שעתוק אורתוגונליים. משמעות הדבר היא כי מפעילים ומדכאים חדשים יונדסו דה נובו על מנת לממש את החיווט המלא בתוך המעגלים הדיגיטליים. באופן עקרוני, ניתן להרכיב חלבונים קושרי דנ”א חדשים באמצעות חלבוני אצבע אבץ3 ואפקטי TAL4 כתבניות. עם זאת, אפשרות זו נראית מפרכת מדי וגוזלת זמן; לכן, יש להסתמך בעיקר על רנ”א קטן ובקרת תרגום כדי לסיים מעגלי גנים מורכבים.
במקור, שיטה זו פותחה כדי לייצר מעגלים דיגיטליים בחיידקים. ואכן, בתאים איקריוטים, במקום רנ”א אנטיסנס, מתאים יותר לדבר על מיקרו-רנ”א (miRNAs) או על רנ”א מפריע קטן (siRNAs)5. עם זאת, מסלול RNAi אינו קיים שמרים S. cerevisiae. לפיכך, יש לבחור ברשתות תמלול מלאות. נניח שמעגל חשמלי זקוק לחמישה מפעילים וחמישה מדכאים; ציון הסיבוכיות שלו יהיה S = 32. ניתן להפחית את מורכבות המעגל על ידי החלפת 10 גורמי השעתוק ב- dCas96 יחיד (Cas9 חסר נוקלאז) המאוחה לתחום הפעלה (AD). כפי שמוצגב-7, dCas9-AD פועל כמדכא בשמרים בעת קשירת מקדם בין תיבת TATA ל-TSS (אתר התחלת שעתוק) וכמפעיל בעת קשירה טובה במעלה הזרם של תיבת TATA. לפיכך, ניתן להחליף 10 גורמי שעתוק בחלבון היתוך dCas9-AD יחיד ו-10 sgRNA (RNA מנחה יחיד) לקבלת ציון סיבוכיות כולל של S = 11. זה מהיר וקל לסנתז עשרה sgRNAs, בעוד שכפי שצוין קודם לכן, הרכבה של 10 חלבונים תדרוש עבודה ארוכה ומסובכת הרבה יותר.
לחלופין, ניתן להשתמש בשני חלבוני dCas אורתוגונליים (למשל, dCas9 ו-dCas12a): אחד כדי להתיך לאלצהיימר, והשני חשוף או בשילוב עם תחום דיכוי. ציון הסיבוכיות יגדל ביחידה אחת בלבד (S = 12). לפיכך, מערכות CRISPR-dCas הן המפתח לבניית מעגלים ספרתיים מורכבים מאוד של גנים בשמרי אפייה (S. cerevisiae).
מאמר זה מאפיין באופן עמוק את היעילות של מדכאים ומפעילים מבוססי dCas9 ו-dCas12a בשמרים. התוצאות מראות כי הם אינם דורשים כמות גבוהה של sgRNA כדי לייעל את פעילותם, ולכן פלסמידים אפיזומליים הם העדיפו להימנע. יתר על כן, מפעילים מבוססי dCas9 יעילים הרבה יותר בעת שימוש ב-RNA פיגום (scRNA) המגייס עותקים של VP64 AD. לעומת זאת, dCas12a עובד היטב כאשר הוא מאוחה ישירות ל-VPR AD החזק. יתר על כן, מקדם מופעל סינתטי דורש מספר משתנה של אתרי מטרה, בהתאם לתצורה של מפעיל (למשל, שלושה בעת שימוש dCas12a-VPR, שישה עבור dCas9-VP64, ורק אחד עם dCas9 ו scRNA). כמדחיק, dCas12a נראה נוקב יותר בעת קשירת אזור הקידוד ולא המקדם.
כחיסרון, עם זאת, CRISPR-dCas9/dCas12a אינם מתקשרים ישירות עם כימיקלים. לכן, ייתכן שלא יועיל להם בשכבת הקלט. מסיבה זו נחקרו עיצובי שערים בוליאניים חלופיים המכילים חלבונים אנטי-קריספר (Acrs). Acrs לפעול על (d)Cas חלבונים ולעכב את פעולתם8. לפיכך, הם אמצעי לווסת את הפעילות של מערכות CRISPR-(d)Cas. מאמר זה מנתח ביסודיות את האינטראקציות בין סוג II Acrs ו- (d)Cas9, כמו גם סוג V Acrs ו- (d)Cas12a ב- S. cerevisiae. מכיוון ש-Acrs קטנים בהרבה מחלבוני Cas, שער NOT המגיב לאסטרוגן β-אסטרדיול נבנה על ידי איחוי תחום קשירת ההורמונים של קולטן האסטרוגן האנושי 9-HBD(hER)-ל-AcrIIA4. חוץ מזה, קומץ שערים של כן ו-NOT שביטאו dCas12a(-AD) באופן מכונן ו-AcrVAs עם אינדוקציה עם גלקטוז מומשו. כיום, שערים אלה משמשים רק כהוכחת היתכנות. עם זאת, הם גם מייצגים את הצעד הראשון לקראת חשיבה מחודשת עמוקה על האלגוריתם לביצוע התכנון האוטומטי החישובי של מעגלים דיגיטליים של גנים סינתטיים בתאי שמרים.
Protocol
Representative Results
Discussion
הפרוטוקול הראה זרימת עבודה מלאה אפשרית עבור מעגלים דיגיטליים של גנים סינתטיים, לאחר מחזור ההנדסה הביולוגית “תכנון-בנייה-בדיקה-למידה” (DBTL) ובנוגע לניסויי מעבדה יבשה ומעבדה רטובה. כאן, התמקדנו במערכת CRISPR-Cas, בעיקר dSpCas9, denAsCas12a, dLbCas12a וחלבוני האנטי-קריספר המתאימים, על ידי תכנון ובנייה ברשתות שעת…
Declarações
The authors have nothing to disclose.
Acknowledgements
אנו רוצים להודות לכל הסטודנטים במעבדה לביולוגיה סינתטית – SPST, TJU – על עזרתם הכללית, יחד עם ג’י לי ושיאנגיאנג ז’אנג על עזרתם בניסויי FACS.
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
Referências
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

