CRISPR-CAS सिस्टम और एंटी-CRISPR प्रोटीन पर आधारित जीन डिजिटल सर्किट
Summary
CRISPR-CAS सिस्टम और एंटी-CRISPR प्रोटीन को Saccharomyces cerevisiae में बूलियन गेट्स की योजना में एकीकृत किया गया था। नए छोटे तर्क सर्किट ने अच्छा प्रदर्शन दिखाया और डीसीएएस 9 / डीसीएएस 12 ए-आधारित प्रतिलेखन कारकों और एंटी-सीआरआईएसपीआर प्रोटीन के गुणों दोनों की समझ को गहरा कर दिया।
Abstract
सिंथेटिक जीन बूलियन गेट्स और डिजिटल सर्किट में चिकित्सा निदान से लेकर पर्यावरण देखभाल तक अनुप्रयोगों की एक विस्तृत श्रृंखला है। CRISPR-Cas सिस्टम और उनके प्राकृतिक अवरोधकों की खोज – एंटी-CRISPR प्रोटीन (Acrs) – विवो जीन डिजिटल सर्किट में डिजाइन और कार्यान्वित करने के लिए एक नया उपकरण प्रदान करता है। यहां, हम एक प्रोटोकॉल का वर्णन करते हैं जो “डिजाइन-बिल्ड-टेस्ट-लर्न” जैविक इंजीनियरिंग चक्र के विचार का पालन करता है और छोटे ट्रांसक्रिप्शनल नेटवर्क स्थापित करने के लिए उनके संबंधित एसीआर के साथ मिलकर डीसीएएस 9 / डीसीएएस 12 ए का उपयोग करता है, जिनमें से कुछ बुलियन गेट्स की तरह व्यवहार करते हैं। ये परिणाम प्रतिलेखन कारकों के रूप में dCas9 / dCas12a के गुणों को इंगित करते हैं। विशेष रूप से, जीन अभिव्यक्ति के अधिकतम सक्रियण को प्राप्त करने के लिए, dSpCas9 को एक इंजीनियर मचान आरएनए के साथ बातचीत करने की आवश्यकता होती है जो VP64 सक्रियण डोमेन (AD) की कई प्रतियां एकत्र करता है। इसके विपरीत, डीसीएएस 12 ए को सी टर्मिनस पर, मजबूत वीपी 64-पी 65-आरटीए (वीपीआर) एडी के साथ जोड़ा जाएगा। इसके अलावा, सेल में एसजीआरएनए / सीआरआरएनए की मात्रा में वृद्धि करके दोनों कैस प्रोटीन की गतिविधि को बढ़ाया नहीं जाता है। यह लेख यह भी बताता है कि CRISPR-dCas-Acr इंटरैक्शन के आधार पर बूलियन गेट कैसे बनाएं। मानव एस्ट्रोजन रिसेप्टर का एसीआरआईआईए 4 फ्यूज्ड हार्मोन-बाइंडिंग डोमेन β-एस्ट्राडियोल के प्रति उत्तरदायी नॉट गेट का मूल है, जबकि इंड्यूसेबल जीएएल 1 प्रमोटर द्वारा संश्लेषित एसीआरवीए एक इनपुट के रूप में गैलेक्टोज के साथ यस और नॉट गेट्स दोनों की नकल करने की अनुमति देता है। बाद के सर्किट में, AcrVA5, dLbCas12a के साथ, सबसे अच्छा तर्क व्यवहार दिखाया।
Introduction
2011 में, शोधकर्ताओं ने एक कम्प्यूटेशनल विधि का प्रस्ताव दिया और डिजिटल सिंथेटिक जीन सर्किट1 के स्वचालित डिजाइन के लिए सॉफ्टवेयर का एक संबंधित टुकड़ा विकसित किया। एक उपयोगकर्ता को इनपुट (तीन या चार) की संख्या निर्दिष्ट करनी थी और सर्किट सत्य तालिका भरनी थी; इसने इलेक्ट्रॉनिक्स से तकनीकों का उपयोग करके सर्किट संरचना प्राप्त करने के लिए सभी आवश्यक जानकारी प्रदान की। सत्य तालिका का कर्णौघ मानचित्र विधि2 के माध्यम से दो बूलियन सूत्रों में अनुवाद किया गया था। प्रत्येक बूलियन सूत्र खंडों से बना होता है जो सर्किट इनपुट और उनके निषेधों (शाब्दिक) के बीच तर्क संचालन (योग या गुणन) का वर्णन करते हैं। सर्किट आउटपुट की गणना करने के लिए खंड, उनकी बारी में, या तो अभिव्यक्त (ओआर) या गुणा (एएनडी) होते हैं। प्रत्येक परिपथ को इसके दो संगत सूत्रों में से किसी एक के अनुसार महसूस किया जा सकता है: एक पीओएस (रकम का उत्पाद) रूप में लिखा गया है और दूसरा एसओपी (उत्पादों का योग) प्रतिनिधित्व में लिखा गया है। पूर्व में खंडों (यानी, बूलियन गेट) का गुणन होता है जिसमें शाब्दिक का तर्क योग होता है। उत्तरार्द्ध, इसके विपरीत, खंडों का एक योग है जहां शाब्दिक गुणा किए जाते हैं।
इलेक्ट्रिक सर्किट को ब्रेडबोर्ड पर, शारीरिक रूप से विभिन्न द्वारों को एक साथ वायर करके महसूस किया जा सकता है। विद्युत प्रवाह फाटकों के बीच संकेतों के आदान-प्रदान की अनुमति देता है, जिससे आउटपुट की गणना होती है।
जीव विज्ञान में, स्थिति अधिक जटिल है। एक बूलियन गेट को प्रतिलेखन इकाई (टीयू; यानी, यूकेरियोटिक कोशिकाओं के अंदर अनुक्रम “प्रमोटर-कोडिंग क्षेत्र-टर्मिनेटर” के रूप में महसूस किया जा सकता है, जहां प्रतिलेखन या अनुवाद (या दोनों) को विनियमित किया जाता है। इस प्रकार, कम से कम दो प्रकार के अणु एक जैविक वायरिंग स्थापित करते हैं: प्रतिलेखन कारक प्रोटीन और गैर-कोडिंग, एंटीसेंस आरएनए1।
एक जीन डिजिटल सर्किट को गेट की दो या तीन परतों में व्यवस्थित किया जाता है, अर्थात्: 1) इनपुट परत, जो हां (बफर) से बनी होती है और गेट नहीं होती है और इनपुट रसायनों को वायरिंग अणुओं में परिवर्तित करती है; 2) आंतरिक परत, जिसमें उतने ही टीयू होते हैं जितने कि संबंधित बूलियन सूत्र में खंड होते हैं। यदि सर्किट एसओपी सूत्र के अनुसार डिज़ाइन किया गया है, तो आंतरिक परत में प्रत्येक खंड तथाकथित वितरित आउटपुट आर्किटेक्चर में सर्किट आउटपुट (जैसे, प्रतिदीप्ति) का उत्पादन करेगा। यदि योग (पीओएस) सूत्र के उत्पाद का उपयोग किया जाता है, तो एक 3) अंतिम परत की आवश्यकता होती है, जिसमें आंतरिक परत से तारों के अणुओं को इकट्ठा करने वाला एक एकल गुणक द्वार होगा।
कुल मिलाकर, सिंथेटिक जीव विज्ञान में, एक ही सर्किट के लिए कई अलग-अलग योजनाएं तैयार की जा सकती हैं। वे टीयू और वायरिंग अणुओं दोनों की संख्या और प्रकार में भिन्न होते हैं। खमीर कोशिकाओं में लागू किए जाने वाले सबसे आसान समाधान को चुनने के लिए, प्रत्येक सर्किट डिज़ाइन एक जटिलता स्कोर एस के साथ जुड़ा हुआ है, जिसे
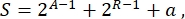
जहां ए सक्रियकर्ताओं की संख्या का प्रतिनिधित्व करता है, आर दमनकारियों की संख्या का प्रतिनिधित्व करता है, और ए एंटीसेंस आरएनए अणुओं की मात्रा है। यदि या तो सक्रियकर्ता या दमनकारी सर्किट से अनुपस्थित हैं, तो एस में उनका योगदान शून्य है। इसलिए, प्रयोगशाला (उच्च एस) में एक सर्किट योजना का एहसास करना अधिक कठिन है जब इसे उच्च संख्या में ऑर्थोगोनल प्रतिलेखन कारकों की आवश्यकता होती है। इसका मतलब है कि डिजिटल सर्किट के अंदर पूर्ण तारों का एहसास करने के लिए नए सक्रियकर्ताओं और दमनकारियों को नए सिरे से इंजीनियर किया जाएगा। सिद्धांत रूप में, नोवेल डीएनए-बाइंडिंग प्रोटीन को जिंक फिंगर प्रोटीन3 और टीएएल प्रभावकों4 का उपयोग करके टेम्पलेट के रूप में इकट्ठा किया जा सकता है। हालांकि, यह विकल्प बहुत कठिन और समय लेने वाला प्रतीत होता है; इसलिए, जटिल जीन सर्किट को अंतिम रूप देने के लिए ज्यादातर छोटे आरएनए और अनुवाद विनियमन पर भरोसा करना चाहिए।
मूल रूप से, यह विधि बैक्टीरिया में डिजिटल सर्किट बनाने के लिए विकसित की गई थी। दरअसल, यूकेरियोटिक कोशिकाओं में, एंटीसेंस आरएनए के बजाय, माइक्रोआरएनए (एमआईआरएनए) या छोटे हस्तक्षेप करने वाले आरएनए (एसआईआरएनए) 5 के बारे में बात करना अधिक उपयुक्त है। हालांकि, आरएनएआई मार्ग खमीर एस सेरेविसिया में मौजूद नहीं है। इसलिए, किसी को पूरी तरह से ट्रांसक्रिप्शनल नेटवर्क का विकल्प चुनना चाहिए। मान लीजिए कि एक सर्किट को पांच सक्रियकर्ताओं और पांच दमनकारियों की आवश्यकता होती है; इसका जटिलता स्कोर एस = 32 होगा। सर्किट जटिलता को 10 प्रतिलेखन कारकों को एक सक्रियण डोमेन (एडी) से जुड़े एकल डीसीएएस 96 (न्यूक्लियस की कमी वाले कैस 9) के साथ बदलकर कम किया जा सकता है। जैसा कि7 में दिखाया गया है, डीसीएएस 9-एडी टाटा बॉक्स और टीएसएस (ट्रांसक्रिप्शन स्टार्ट साइट) के बीच प्रमोटर को बांधते समय खमीर में एक दमनकारी के रूप में काम करता है और टाटा बॉक्स के ऊपर अच्छी तरह से बांधने पर एक उत्प्रेरक के रूप में काम करता है। इस प्रकार, एस = 11 के कुल जटिलता स्कोर के लिए 10 प्रतिलेखन कारकों को एकल डीसीएएस 9-एडी संलयन प्रोटीन और 10 एसजीआरएनए (एकल गाइड आरएनए) के साथ प्रतिस्थापित किया जा सकता है। दस एसजीआरएनए को संश्लेषित करना त्वरित और आसान है, जबकि, जैसा कि पहले टिप्पणी की गई थी, 10 प्रोटीन ों की असेंबली बहुत लंबे और अधिक जटिल काम की मांग करेगी।
वैकल्पिक रूप से, कोई दो ऑर्थोगोनल डीसीएएस प्रोटीन (जैसे, डीसीएएस 9 और डीसीएएस 12 ए) का उपयोग कर सकता है: एक एडी में फ्यूज करने के लिए, और दूसरा नंगे या दमन डोमेन के साथ संयोजन में। जटिलता स्कोर केवल एक इकाई (एस = 12) से बढ़ेगा। इसलिए, CRISPR-dCas सिस्टम S. Cerevisiae में बहुत जटिल जीन डिजिटल सर्किट के निर्माण की कुंजी हैं।
यह पेपर खमीर में dCas9- और dCas12a-आधारित रिप्रेसर्स और एक्टिवेटर्स दोनों की दक्षता को गहराई से दर्शाता है। परिणाम बताते हैं कि वे अपनी गतिविधि को अनुकूलित करने के लिए एसजीआरएनए की उच्च मात्रा की मांग नहीं करते हैं, इसलिए एपिसोमल प्लास्मिड को अधिमानतः टाला जाता है। इसके अलावा, डीसीएएस 9-आधारित सक्रियकर्ता एक मचान आरएनए (एससीआरएनए) का उपयोग करते समय कहीं अधिक प्रभावी होते हैं जो वीपी 64 एडी की प्रतियां भर्ती करता है। इसके विपरीत, मजबूत वीपीआर एडी से सीधे जुड़े होने पर डीसीएएस 12 ए अच्छी तरह से काम करता है। इसके अलावा, एक सिंथेटिक सक्रिय प्रमोटर एक्टिवेटर के कॉन्फ़िगरेशन के आधार पर लक्ष्य साइटों की एक चर संख्या की मांग करता है (उदाहरण के लिए, डीसीएएस 12 ए-वीपीआर का उपयोग करते समय तीन, डीसीएएस 9-वीपी 64 के लिए छह, और डीसीएएस 9 और एससीआरएनए के साथ केवल एक)। एक दमनकारी के रूप में, प्रमोटर के बजाय कोडिंग क्षेत्र को बांधते समय डीसीएएस 12 ए अधिक तीक्ष्ण दिखाई देता है।
एक दोष के रूप में, हालांकि, CRISPR-dCas9 / dCas12a सीधे रसायनों के साथ बातचीत नहीं करते हैं। इसलिए, इनपुट परत में उनका कोई उपयोग नहीं हो सकता है। इस कारण से, एंटी-सीआरआईएसपीआर प्रोटीन (एसीआर) युक्त वैकल्पिक बूलियन गेट डिजाइनों की जांच की गई है। एसीआरएस (डी) सीएएस प्रोटीन पर कार्य करते हैं औरउनके काम को रोकते हैं। इसलिए, वे CRISPR-(d) CAS सिस्टम की गतिविधि को संशोधित करने का एक साधन हैं। यह पेपर टाइप II Acrs और (d) Cas9 के बीच बातचीत का पूरी तरह से विश्लेषण करता है, साथ ही साथ टाइप V Acrs और (d) Cas12a में S. cerevisiae में। चूंकि एसीआरएस सीएएस प्रोटीन की तुलना में बहुत छोटे होते हैं, इसलिए एस्ट्रोजेन β-एस्ट्राडियोल के प्रति उत्तरदायी एक नॉट गेट मानव एस्ट्रोजन रिसेप्टर 9-एचबीडी (एचईआर) -से एसीआरआईआईए 4 के हार्मोन-बाइंडिंग डोमेन को फ्यूज करके बनाया गया था। इसके अलावा, गैलेक्टोज के साथ प्रेरण पर डीसीएएस 12 ए (-एडी) और एसीआरवीए को संवैधानिक रूप से व्यक्त करने वाले मुट्ठी भर हां और नॉट गेट्स महसूस किए गए थे। वर्तमान में, ये द्वार केवल अवधारणा के प्रमाण के रूप में कार्य करते हैं। हालांकि, वे खमीर कोशिकाओं में सिंथेटिक जीन डिजिटल सर्किट के कम्प्यूटेशनल स्वचालित डिजाइन को पूरा करने के लिए एल्गोरिदम के गहरे पुनर्विचार की दिशा में पहले कदम का भी प्रतिनिधित्व करते हैं।
Protocol
Representative Results
Discussion
प्रोटोकॉल ने “डिजाइन-बिल्ड-टेस्ट-लर्न” (डीबीटीएल) जैविक इंजीनियरिंग चक्र और ड्राई-लैब और वेट-लैब प्रयोगों दोनों के संबंध में सिंथेटिक जीन डिजिटल सर्किट के लिए एक संभावित पूर्ण वर्कफ़्लो दिखाया। यहां, ?…
Declarações
The authors have nothing to disclose.
Acknowledgements
हम सिंथेटिक जीवविज्ञान प्रयोगशाला-एसपीएसटी, टीजेयू-के सभी छात्रों को उनकी सामान्य मदद के लिए धन्यवाद देना चाहते हैं, साथ ही एफएसीएस प्रयोगों में उनकी सहायता के लिए झी ली और जियांगयांग झांग के साथ।
Materials
| 0.1 mL PCR 8-strip tubes | NEST | 403112 | |
| 0.2 mL PCR tubes | Axygen | PCR-02-C | |
| 1.5 mL Microtubes | Axygen | MCT-150-C | |
| 15 mL Centrifuge tubes | BIOFIL | CFT011150 | |
| 2 mL Microtubes | Axygen | MCT-200-C | |
| 50 mL Centrifuge tubes | BIOFIL | CFT011500 | |
| Agarose-molecular biology grade | Invitrogen | 75510-019 | |
| Ampicillin sodium salt | Solarbio | 69-52-3 | |
| Applied biosystems veriti 96-well thermal cycler | Thermo Fisher Scientific | 4375786 | |
| AxyPrep DNA gel extraction kit | Axygen | AP-GX-250 | |
| BD FACSuite CS&T research beads | BD | 650621 | Fluorescent beads |
| BD FACSVerse flow cytometer | BD | – | |
| Centrifuge | Eppendorf | 5424 | |
| Centrifuge Sorvall ST 16R | Thermo Fisher Scientific | 75004380 | |
| E. coli competent cells (Strain DH5α) | Life Technologies | 18263-012 | |
| ECL select Western Blotting detection reagent | GE Healthcare | RPN2235 | |
| Electrophoresis apparatus | Beijing JUNYI Electrophoresis Co., Ltd | JY300C | |
| Flat 8-strip caps | NEST | 406012 | |
| Gene synthesis company | Azenta Life Sciences | https://web.azenta.com/zh-cn/azenta-life-sciences | |
| Goat anti-Mouse IgG (H+L) cross-adsorbed secondary antibody Alexa Fluor 568 | Invitrogen | A-11004 | |
| HiFiScript cDNA synthesis kit | CWBIO | CW2569M | Kit used in step 6.2.2.1 |
| Lysate solution (Zymolyase) | zymoresearch | E1004-A | |
| Nikon Eclipse 80i fluorescence microscope | Nikon | – | Fluorescence microscope |
| Pipet tips—10 μL | Axygen | T-300-R-S | |
| Pipet tips—1000 μL | Axygen | T-1000-B-R-S | |
| Pipet tips—200 μL | Axygen | T-200-Y-R-S | |
| pRSII403 | Addgene | 35436 | |
| pRSII404 | Addgene | 35438 | |
| pRSII405 | Addgene | 35440 | |
| pRSII406 | Addgene | 35442 | |
| pRSII424 | Addgene | 35466 | |
| pTPGI_dSpCas9_VP64 | Addgene | 49013 | |
| Q5 High-fidelity DNApolymerase | New England Biolabs | M0491 | |
| Restriction enzyme-Acc65I | New England Biolabs | R0599 | |
| Restriction enzyme-BamHI | New England Biolabs | R0136 | |
| Restriction enzyme-SacI-HF | New England Biolabs | R3156 | |
| Restriction enzyme-XhoI | New England Biolabs | R0146 | |
| Roche LightCycler 96 | Roche | – | Real-Time PCR Instrument |
| S. cerevisiae CEN.PK2-1C | – | – | The parent strain. The genotype is: MATa; his3D1; leu2-3_112; ura3-52; trp1-289; MAL2-8c; SUC2 |
| Stem-Loop Kit | SparkJade | AG0502 | Kit used in step 6.2.1.3 |
| T100 Thermal Cycler | BIO-RAD | 186-1096 | |
| T4 DNA ligase | New England Biolabs | M0202 | |
| T5 Exonuclease | New England Biolabs | M0363 | |
| Taq DNA ligase | New England Biolabs | M0208 | |
| Taq DNA polymerase | New England Biolabs | M0495 | |
| TB Green Premix Ex Taq II (Tli RNaseH Plus)(2x) (SYBR Green I dye) | Takara | RR820Q | |
| YeaStar RNA kit | Zymo Research | R1002 | |
| β-estradiol | Sigma-Aldrich | E8875 |
Referências
- Marchisio, M. A., Stelling, J. Automatic design of digital synthetic gene circuits. PLOS Computational Biology. 7 (2), 1001083 (2011).
- Karnaugh, M. The map method for synthesis of combinational logic circuits. Transactions of the American Institute of Electrical Engineers. 72 (9), 593-599 (1953).
- Mandell, J. G., Barbas, C. F. Zinc finger tools: custom DNA-binding domains for transcription factors and nucleases. Nucleic Acids Research. 34, 516-523 (2006).
- Bogdanove, A. J., Voytas, D. F. TAL effectors: customizable proteins for DNA targeting. Science. 333 (6051), 1843-1846 (2011).
- Drinnenberg, I. A., et al. RNAi in budding yeast. Science. 326 (5952), 544-550 (2009).
- Gander, M. W., Vrana, J. D., Voje, W. E., Carothers, J. M., Klavins, E. Digital logic circuits in yeast with CRISPR-dCas9 NOR gates. Nature Communications. 8, 15459 (2017).
- Farzadfard, F., Perli, S. D., Lu, T. K. Tunable and multifunctional eukaryotic transcription factors based on CRISPR/Cas. ACS Synthetic Biology. 2 (10), 604-613 (2013).
- Nakamura, M., et al. Anti-CRISPR-mediated control of gene editing and synthetic circuits in eukaryotic cells. Nature Communications. 10 (1), 194 (2019).
- Louvion, J. F., Havaux-Copf, B., Picard, D. Fusion of GAL4-VP16 to a steroid-binding domain provides a tool for gratuitous induction of galactose-responsive genes in yeast. Gene. 131 (1), 129-134 (1993).
- DiCarlo, J. E., et al. Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. Nucleic Acids Research. 41 (7), 4336-4343 (2013).
- Gao, Y., Zhao, Y. Self-processing of ribozyme-flanked RNAs into guide RNAs in vitro and in vivo for CRISPR-mediated genome editing. Journal of Integrative Plant Biology. 56 (4), 343-349 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Fonfara, I., Richter, H., Bratovic, M., Le Rhun, A., Charpentier, E. The CRISPR-associated DNA-cleaving enzyme Cpf1 also processes precursor CRISPR RNA. Nature. 532 (7600), 517-521 (2016).
- Yu, L., Marchisio, M. A. Saccharomyces cerevisiae synthetic transcriptional networks harnessing dCas12a and Type V-A anti-CRISPR proteins. ACS Synthetic Biology. 10 (4), 870-883 (2021).
- Zhang, Y., Marchisio, M. A. Interaction of bare dSpCas9, scaffold gRNA, and type II anti-CRISPR proteins highly favors the control of gene expression in the yeast S. cerevisiae. ACS Synthetic Biology. 11 (1), 176-190 (2022).
- Sheff, M. A., Thorn, K. S. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae. Yeast. 21 (8), 661-670 (2004).
- Naito, Y., Hino, K., Bono, H., Ui-Tei, K. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites. Bioinformatics. 31 (7), 1120-1123 (2015).
- Chee, M. K., Haase, S. B. New and redesigned pRS plasmid shuttle vectors for genetic manipulation of Saccharomyces cerevisiae. G3: Genes|Genomes|Genetics. 2 (5), 515-526 (2012).
- Gibson, D. G. Synthesis of DNA fragments in yeast by one-step assembly of overlapping oligonucleotides. Nucleic Acids Research. 37 (20), 6984-6990 (2009).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. Journal of Visualized Experiments. (6), e253 (2007).
- Green, M. R., Sambrook, J. . Molecular Cloning. Fourth edition. , (2012).
- Sanger, F. Determination of nucleotide sequences in DNA. Science. 214 (4526), 1205-1210 (1981).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Zalatan, J. G., et al. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell. 160 (1-2), 339-350 (2015).
- Song, W., Li, J., Liang, Q., Marchisio, M. A. Can terminators be used as insulators into yeast synthetic gene circuits. Journal of Biological Engineering. 10, 19 (2016).
- Rauch, B. J., et al. Inhibition of CRISPR-Cas9 with bacteriophage proteins. Cell. 168 (1-2), 150-158 (2017).
- Hynes, A. P., et al. An anti-CRISPR from a virulent streptococcal phage inhibits Streptococcus pyogenes Cas9. Nature Microbiology. 2 (10), 1374-1380 (2017).
- Watters, K. E., Fellmann, C., Bai, H. B., Ren, S. M., Doudna, J. A. Systematic discovery of natural CRISPR-Cas12a inhibitors. Science. 362 (6411), 236-239 (2018).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Research. 33 (20), 179 (2005).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 29 (9), 45 (2001).
- Hahne, F., et al. flowCore: a Bioconductor package for high throughput flow cytometry. BMC Bioinformatics. 10 (1), 106 (2009).
- Li, J., Xu, Z., Chupalov, A., Marchisio, M. A. Anti-CRISPR-based biosensors in the yeast S. cerevisiae. Journal of Biological Engineering. 12, 11 (2018).
- Dong, L., et al. An anti-CRISPR protein disables type V Cas12a by acetylation. Nature Structural & Molecular Biology. 26 (4), 308-314 (2019).

