In vitro Kemisk kortlægning af G-quadruplex DNA-strukturer af bis-3-chloropiperidiner
Summary
Bis-3-chlorpiperidiner (B-CeP’er) er nyttige kemiske sonder til at identificere og karakterisere G-quadruplex strukturer i DNA-skabeloner in vitro. Denne protokol beskriver proceduren til udførelse af sondereaktioner med B-CeP’er og til opløsning af reaktionsprodukter ved polyacrylamidgelelektroforese med høj opløsning.
Abstract
G-quadruplexes (G4s) er biologisk relevante, ikke-kanoniske DNA-strukturer, der spiller en vigtig rolle i genekspression og sygdomme, der repræsenterer betydelige terapeutiske mål. Tilgængelige metoder er nødvendige til in vitro karakterisering af DNA inden for potentielle G-quadruplex-dannende sekvenser (PQS’er). B-CeP’er er en klasse af alkyleringsmidler, der har vist sig at være nyttige kemiske sonder til undersøgelse af nukleinsyrers højere ordens struktur. Dette papir beskriver et nyt kemisk kortlægningsassay, der udnytter den specifikke reaktivitet af B-CeP’er med N7 af guaniner, efterfulgt af direkte strengspaltning ved de alkylerede Gs.
For at skelne G4-folder fra udfoldede DNA-former bruger vi nemlig B-CeP 1 til at sonde trombinbindende aptamer (TBA), et 15-mer DNA, der er i stand til at antage G4-arrangementet. Reaktion af B-CeP-responderende guaniner med B-CeP 1 giver produkter, der kan opløses ved polyacrylamidgelelektroforese (PAGE) med høj opløsning på et enkeltnukleotidniveau ved at lokalisere individuelle alkyleringsaddukter og DNA-strengspaltning ved de alkylerede guaniner. Kortlægning ved hjælp af B-CeP’er er et simpelt og kraftfuldt værktøj til in vitro-karakterisering af G-quadruplex-dannende DNA-sekvenser, der muliggør den præcise placering af guaniner involveret i dannelsen af G-tetrader.
Introduction
Ud over den typiske Watson-Crick-dobbelthelix kan nukleinsyrer vedtage forskellige sekundære strukturer, såsom den alternative G-quadruplex (G4) form på grund af deres guaninrige sekvenser. G4-strukturen er baseret på dannelsen af plane tetramerer, kaldet G-tetrader, hvor fire guaniner interagerer gennem Hoogsteen-hydrogenbindinger. G-tetrader stables og stabiliseres yderligere af monovalente kationer, der koordineres i midten af guaninkernen (figur 1)1.
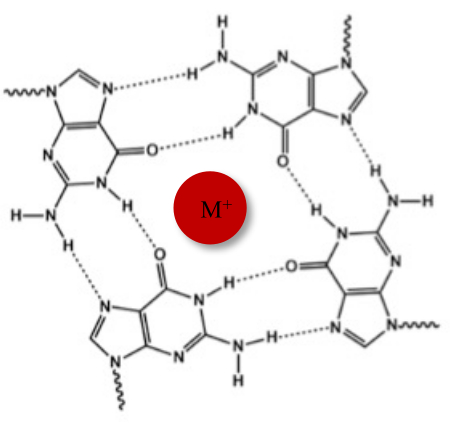
Figur 1: Skematisk gengivelse af en G-quadruplex-struktur. (A) Skematisk gengivelse af en G-tetrad. Det plane array stabiliseres ved Hoogsteen baseparring og ved en central kation (M +). Klik her for at se en større version af denne figur.
Sekvenser med fire eller flere kørsler af mindst to på hinanden følgende guaninnukleotider er potentielle G-quadruplex-dannende sekvenser (PQS’er), der kan foldes i G-quadruplex-strukturer. PQS’er er placeret i mange forskellige cellulære sammenhænge, såsom telomerer, genpromotorer, ribosomalt DNA og rekombinationssteder, og er involveret i reguleringen af mange biologiske processer2. Derfor er identifikation og eksperimentel validering af G4s i det menneskelige genom, som i øjeblikket primært udføres ved hjælp af beregningsværktøjer, et biologisk relevant spørgsmål3. For at understøtte beregningsmæssige forudsigelser eller detektere uforudsigelige G4-strukturer vises her en tilgængelig metode baseret på kemisk kortlægning til at identificere G4-dannelsen i en DNA-skabelon, hvilket muliggør præcis identifikation af guaniner, der danner G-tetradstrukturen.
Det rapporterede kemiske kortlægningsassay udnytter den forskellige reaktivitet af bis-3-chlorpiperidiner (B-CeP’er) med guaniner efter dannelsen af G4-strukturer. På grund af deres høje reaktivitet med nukleofiler 4,5,6,7,8,9 er B-CeP’er nukleinsyre-alkyleringsmidler med evnen til at reagere meget effektivt med N7-positionen af guaninnukleotider10. Alkylering efterfølges af depurination og strengspaltning i enkelt- og dobbeltstrengede DNA-konstruktioner. Tværtimod er guaniner, der er involveret i dannelsen af G-tetraderne i G4-arrangementer, uigennemtrængelige for B-CeP-alkylering, da guaninernes N7-positioner impliceret i Hoogsteen-hydrogenbindingerne. Denne specifikke reaktivitet af B-CeP’er tillader ikke kun påvisning af G4-strukturer, men også identifikation af guaninerne, der danner tetraden (e), da de kan udledes af deres relative beskyttelse mod alkylering sammenlignet med guaniner i enkelt- og dobbeltstrenget DNA.
Den kemiske kortlægningsprotokol rapporteres her ved hjælp af B-CeP 1 (figur 2A) som en sonde til karakterisering af thrombinbindende aptamer (TBA), et 15-mer DNA, der er i stand til at antage G4-arrangementet i nærvær af kaliumkationer11,12. G4-arrangementet af TBA (G4-TBA) sammenlignes direkte med to kontroller, nemlig TBA i enkeltstrenget form (ssTBA) og TBA udglødet til dets komplementære sekvens for at danne den dobbeltstrengede konstruktion (dsTBA) (tabel 1). Produkter af sondereaktioner opløses ved polyacrylamidgelelektroforese med høj opløsning (PAGE) på enkeltnukleotidniveau ved at lokalisere individuelle alkyleringsaddukter og DNA-strengspaltning ved de alkylerede guaniner. Visualisering på gelen muliggøres ved konjugering af TBA-oligonukleotiden med en fluorofor i dens 3′-ende (tabel 1). Denne protokol viser, hvordan man folder TBA i dens forskellige konformationer (G4 og kontroller), og hvordan man udfører sonderende reaktioner med B-CeP’er efterfulgt af PAGE.
Protocol
Representative Results
Discussion
G-quadruplexes er nukleinsyre sekundære strukturer, der typisk foldes inden for guaninrige DNA-sekvenser, og er betydelige forskningsmål på grund af deres tilknytning til genetisk kontrol og sygdomme. Kemisk kortlægning af B-CeP’er er en nyttig protokol til karakterisering af DNA G4’er, som kan bruges til at identificere guaninbaserne involveret i dannelsen af G-tetrader under fysiologiske saltforhold.
Den kemiske sonde, der anvendes i denne protokol, er B-CeP 1 (figur…
Declarações
The authors have nothing to disclose.
Acknowledgements
Dette arbejde blev støttet af Institut for Farmaceutiske og Farmakologiske Videnskaber, University of Padova (PRIDJ-BIRD2019).
Materials
| Acrylamide/bis-acrylamide solution 40% | Applichem | A3658 | R45-46-20/21-25-36/38-43-48/23/ 24/25-62 |
| Ammonium per-sulfate (APS) | Sigma Aldrich | A7460 | |
| Analytical balance | Mettler Toledo | ||
| Autoclave | pbi international | ||
| Boric acid | Sigma Aldrich | B0252 | |
| Bromophenol blue Brilliant blue R | Sigma Aldrich | B0149 | |
| di-Sodium hydrogen phosphate dodecahydrate | Fluka | 71649 | |
| DMSO | Sigma Aldrich | 276855 | |
| DNA oligonucleotides | Integrated DNA Technologies | synthesis of custom sequences | |
| EDTA disodium | Sigma Aldrich | E5134 | |
| Formamide | Fluka | 40248 | H351-360D-373 |
| Gel imager | GE Healtcare | STORM B40 | |
| Glycerol | Sigma Aldrich | G5516 | |
| Micro tubes 0.5 mL | Sarstedt | 72.704 | |
| Potassium Chloride | Sigma Aldrich | P9541 | |
| Sequencing apparatus | Biometra | Model S2 | |
| Silanization solution I | Fluka | 85126 | H225, 314, 318, 336, 304, 400, 410 |
| Sodium phosphate monobasic | Carlo Erba | 480086 | |
| Speedvac concentrator | Thermo Scientific | Savant DNA 120 | |
| TEMED | Fluka | 87689 | R11-21/22-23-34 |
| Tris-HCl | MERCK | 1.08387.2500 | |
| Urea | Sigma Aldrich | 51456 | |
| UV-Vis spectrophotometer | Thermo Scientific | Nanodrop 1000 |
Referências
- Davis, J. T. G-quartets 40 years later: from 5′-GMP to molecular biology and supramolecular chemistry. Angewandte Chemie. 43 (6), 668-698 (2004).
- Varshney, D., Spiegel, J., Zyner, K., Tannahill, D., Balasubramanian, S. The regulation and functions of DNA and RNA G-quadruplexes. Nature Reviews Molecular Cell Biology. 21 (8), 459-474 (2020).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nature Biotechnology. 33 (8), 877-881 (2015).
- Zuravka, I., Sosic, A., Gatto, B., Gottlich, R. Synthesis and evaluation of a bis-3-chloropiperidine derivative incorporating an anthraquinone pharmacophore. Bioorganic & Medicinal Chemistry Letters. 25 (20), 4606-4609 (2015).
- Zuravka, I., Roesmann, R., Sosic, A., Gottlich, R., Gatto, B. Bis-3-chloropiperidines containing bridging lysine linkers: Influence of side chain structure on DNA alkylating activity. Bioorganic & Medicinal Chemistry. 23 (6), 1241-1250 (2015).
- Zuravka, I., et al. Synthesis and DNA cleavage activity of bis-3-chloropiperidines as alkylating agents. ChemMedChem. 9 (9), 2178-2185 (2014).
- Sosic, A., Gottlich, R., Fabris, D., Gatto, B. B-CePs as cross-linking probes for the investigation of RNA higher-order structure. Nucleic Acids Research. 49 (12), 6660-6672 (2021).
- Sosic, A., et al. Bis-3-chloropiperidines targeting TAR RNA as a novel strategy to impair the HIV-1 nucleocapsid protein. Molecules. 26 (7), 1874 (2021).
- Sosic, A., et al. In vitro evaluation of bis-3-chloropiperidines as RNA modulators targeting TAR and TAR-protein interaction. International Journal of Molecular Sciences. 23 (2), 582 (2022).
- Sosic, A., et al. Direct and topoisomerase II mediated DNA damage by bis-3-chloropiperidines: The importance of being an earnest G. ChemMedChem. 12 (17), 1471-1479 (2017).
- Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H., Toole, J. J. Selection of single-stranded DNA molecules that bind and inhibit human thrombin. Nature. 355 (6360), 564-566 (1992).
- Paborsky, L. R., McCurdy, S. N., Griffin, L. C., Toole, J. J., Leung, L. L. The single-stranded DNA aptamer-binding site of human thrombin. The Journal of Biological Chemistry. 268 (28), 20808-20811 (1993).
- Carraro, C., et al. Behind the mirror: chirality tunes the reactivity and cytotoxicity of chloropiperidines as potential anticancer agents. ACS Medicinal Chemistry Letters. 10 (4), 552-557 (2019).
- Carraro, C., et al. Appended aromatic moieties in flexible bis-3-chloropiperidines confer tropism against pancreatic cancer cells. ChemMedChem. 16 (5), 860-868 (2021).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Onel, B., Wu, G., Sun, D., Lin, C., Yang, D. Electrophoretic mobility shift assay and dimethyl sulfate footprinting for characterization of G-quadruplexes and G-quadruplex-protein complexes. Methods in Molecular Biology. 2035, 201-222 (2019).

