In vitro Kjemisk kartlegging av G-quadruplex DNA-strukturer av Bis-3-kloropiperidiner
Summary
Bis-3-kloropiperidiner (B-CePs) er nyttige kjemiske prober for å identifisere og karakterisere G-quadruplex strukturer i DNA-maler in vitro. Denne protokollen beskriver prosedyren for å utføre sonderingsreaksjoner med B-CePs og for å løse reaksjonsprodukter ved høyoppløselig polyakrylamidgelelektroforese.
Abstract
G-quadruplexes (G4s) er biologisk relevante, ikke-kanoniske DNA-strukturer som spiller en viktig rolle i genuttrykk og sykdommer, som representerer betydelige terapeutiske mål. Tilgjengelige metoder er nødvendige for in vitro-karakterisering av DNA innenfor potensielle G-quadruplex-formende sekvenser (PQS). B-CePs er en klasse av alkyleringsmidler som har vist seg å være nyttige kjemiske prober for undersøkelse av høyere ordens struktur av nukleinsyrer. Dette papiret beskriver en ny kjemisk kartleggingsanalyse som utnytter den spesifikke reaktiviteten til B-CePs med N7 av guaniner, etterfulgt av direkte strengspaltning ved de alkylerte Gs.
For å skille G4-folder fra utfoldede DNA-former, bruker vi B-CeP 1 til å undersøke trombinbindende aptamer (TBA), et 15-mer DNA som er i stand til å anta G4-arrangementet. Reaksjon av B-CeP-responderende guaniner med B-CeP 1 gir produkter som kan løses ved høyoppløselig polyakrylamidgelelektroforese (PAGE) på et enkeltnukleotidnivå ved å lokalisere individuelle alkyleringsaddukter og DNA-strengspaltning ved de alkylerte guaninene. Kartlegging ved hjelp av B-CePs er et enkelt og kraftig verktøy for in vitro-karakterisering av G-quadruplex-dannende DNA-sekvenser, noe som muliggjør den nøyaktige plasseringen av guaniner involvert i dannelsen av G-tetrads.
Introduction
I tillegg til den typiske Watson-Crick dobbeltspiralen, kan nukleinsyrer vedta forskjellige sekundære strukturer, slik som den alternative G-quadruplex (G4) formen, på grunn av deres guaninrike sekvenser. G4-strukturen er basert på dannelsen av plane tetramere, kalt G-tetrads, hvor fire guaniner interagerer gjennom Hoogsteen hydrogenbindinger. G-tetrads stables og stabiliseres videre av monovalente kationer som koordineres i midten av guaninkjernen (figur 1)1.
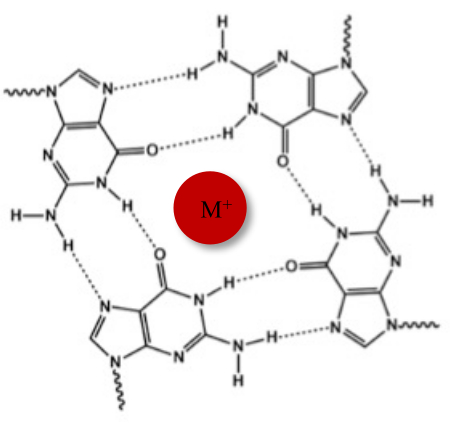
Figur 1: Skjematisk fremstilling av en G-quadruplex struktur. (A) Skjematisk fremstilling av en G-tetrad. Den plane matrisen stabiliseres av Hoogsteen-baseparing og av et sentralt kation (M+). Klikk her for å se en større versjon av denne figuren.
Sekvenser med fire eller flere kjøringer av minst to påfølgende guaninnukleotider er potensielle G-quadruplex-dannende sekvenser (PQS) som kan foldes i G-quadruplex strukturer. PQS er lokalisert i mange forskjellige cellulære sammenhenger, for eksempel ved telomerer, genpromotorer, ribosomalt DNA og rekombinasjonssteder, og er involvert i reguleringen av mange biologiske prosesser2. Derfor er identifisering og eksperimentell validering av G4s i det menneskelige genom, som for tiden utføres primært gjennom beregningsverktøy, et biologisk relevant problem3. For å støtte beregningsmessige prediksjoner eller oppdage uforutsette G4-strukturer, vises en tilgjengelig metode basert på kjemisk kartlegging for å identifisere G4-dannelsen i en DNA-mal her, noe som muliggjør presis identifisering av guaniner som danner G-tetradstrukturen.
Den rapporterte kjemiske kartleggingsanalysen utnytter den forskjellige reaktiviteten til bis-3-kloropiperidiner (B-CePs) med guaniner etter dannelsen av G4-strukturer. På grunn av deres høye reaktivitet med nukleofiler 4,5,6,7,8,9, er B-CePs nukleinsyre-alkyleringsmidler med evnen til å reagere meget effektivt med N7-posisjonenav guaninnukleotider10. Alkylering etterfølges av rensing og strengspaltning i enkelt- og dobbeltstrengede DNA-konstruksjoner. Tvert imot er guaniner involvert i dannelsen av G-tetrads i G4-arrangementer ugjennomtrengelige for B-CeP-alkylering, da N7-posisjonen til guaniner er involvert i Hoogsteen-hydrogenbindingene. Denne spesifikke reaktiviteten til B-CePs tillater ikke bare deteksjon av G4-strukturer, men også identifisering av guaninene som danner tetrad (e), da de kan utledes fra deres relative beskyttelse mot alkylering sammenlignet med guaniner i enkelt- og dobbeltstrenget DNA.
Den kjemiske kartleggingsprotokollen er rapportert her ved hjelp av B-CeP 1 (figur 2A) som en sonde for karakterisering av trombinbindende aptamer (TBA), et 15-mer DNA som er i stand til å anta G4-arrangementet i nærvær av kaliumkationer11,12. G4-arrangementet av TBA (G4-TBA) sammenlignes direkte med to kontroller, nemlig TBA i enkeltstrenget form (ssTBA) og TBA glødet til sin komplementære sekvens for å danne den dobbeltstrengede konstruksjonen (dsTBA) (tabell 1). Produkter av sonderingsreaksjoner løses ved høyoppløselig polyakrylamidgelelektroforese (PAGE) på enkeltnukleotidnivå ved å lokalisere individuelle alkyleringsaddukter og DNA-strengspaltning ved de alkylerte guaninene. Visualisering på gelen muliggjøres ved konjugering av TBA-oligonukleotidet med en fluorofor i 3′-enden (tabell 1). Denne protokollen viser hvordan man bretter TBA i sine forskjellige konformasjoner (G4 og kontroller), og hvordan man utfører sonderingsreaksjoner med B-CePs etterfulgt av PAGE.
Protocol
Representative Results
Discussion
G-quadruplexes er nukleinsyre sekundære strukturer som vanligvis brettes innenfor guaninrike DNA-sekvenser, og er betydelige forskningsmål på grunn av deres tilknytning til genetisk kontroll og sykdommer. Kjemisk kartlegging av B-CePs er en nyttig protokoll for karakterisering av DNA G4s, som kan brukes til å identifisere guaninbasene som er involvert i dannelsen av G-tetrads under fysiologiske saltforhold.
Den kjemiske sonden som brukes i denne protokollen er B-CeP 1 (<strong class="xfig"…
Declarações
The authors have nothing to disclose.
Acknowledgements
Dette arbeidet ble støttet av Institutt for farmasøytiske og farmakologiske vitenskap, Universitetet i Padova (PRIDJ-BIRD2019).
Materials
| Acrylamide/bis-acrylamide solution 40% | Applichem | A3658 | R45-46-20/21-25-36/38-43-48/23/ 24/25-62 |
| Ammonium per-sulfate (APS) | Sigma Aldrich | A7460 | |
| Analytical balance | Mettler Toledo | ||
| Autoclave | pbi international | ||
| Boric acid | Sigma Aldrich | B0252 | |
| Bromophenol blue Brilliant blue R | Sigma Aldrich | B0149 | |
| di-Sodium hydrogen phosphate dodecahydrate | Fluka | 71649 | |
| DMSO | Sigma Aldrich | 276855 | |
| DNA oligonucleotides | Integrated DNA Technologies | synthesis of custom sequences | |
| EDTA disodium | Sigma Aldrich | E5134 | |
| Formamide | Fluka | 40248 | H351-360D-373 |
| Gel imager | GE Healtcare | STORM B40 | |
| Glycerol | Sigma Aldrich | G5516 | |
| Micro tubes 0.5 mL | Sarstedt | 72.704 | |
| Potassium Chloride | Sigma Aldrich | P9541 | |
| Sequencing apparatus | Biometra | Model S2 | |
| Silanization solution I | Fluka | 85126 | H225, 314, 318, 336, 304, 400, 410 |
| Sodium phosphate monobasic | Carlo Erba | 480086 | |
| Speedvac concentrator | Thermo Scientific | Savant DNA 120 | |
| TEMED | Fluka | 87689 | R11-21/22-23-34 |
| Tris-HCl | MERCK | 1.08387.2500 | |
| Urea | Sigma Aldrich | 51456 | |
| UV-Vis spectrophotometer | Thermo Scientific | Nanodrop 1000 |
Referências
- Davis, J. T. G-quartets 40 years later: from 5′-GMP to molecular biology and supramolecular chemistry. Angewandte Chemie. 43 (6), 668-698 (2004).
- Varshney, D., Spiegel, J., Zyner, K., Tannahill, D., Balasubramanian, S. The regulation and functions of DNA and RNA G-quadruplexes. Nature Reviews Molecular Cell Biology. 21 (8), 459-474 (2020).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nature Biotechnology. 33 (8), 877-881 (2015).
- Zuravka, I., Sosic, A., Gatto, B., Gottlich, R. Synthesis and evaluation of a bis-3-chloropiperidine derivative incorporating an anthraquinone pharmacophore. Bioorganic & Medicinal Chemistry Letters. 25 (20), 4606-4609 (2015).
- Zuravka, I., Roesmann, R., Sosic, A., Gottlich, R., Gatto, B. Bis-3-chloropiperidines containing bridging lysine linkers: Influence of side chain structure on DNA alkylating activity. Bioorganic & Medicinal Chemistry. 23 (6), 1241-1250 (2015).
- Zuravka, I., et al. Synthesis and DNA cleavage activity of bis-3-chloropiperidines as alkylating agents. ChemMedChem. 9 (9), 2178-2185 (2014).
- Sosic, A., Gottlich, R., Fabris, D., Gatto, B. B-CePs as cross-linking probes for the investigation of RNA higher-order structure. Nucleic Acids Research. 49 (12), 6660-6672 (2021).
- Sosic, A., et al. Bis-3-chloropiperidines targeting TAR RNA as a novel strategy to impair the HIV-1 nucleocapsid protein. Molecules. 26 (7), 1874 (2021).
- Sosic, A., et al. In vitro evaluation of bis-3-chloropiperidines as RNA modulators targeting TAR and TAR-protein interaction. International Journal of Molecular Sciences. 23 (2), 582 (2022).
- Sosic, A., et al. Direct and topoisomerase II mediated DNA damage by bis-3-chloropiperidines: The importance of being an earnest G. ChemMedChem. 12 (17), 1471-1479 (2017).
- Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H., Toole, J. J. Selection of single-stranded DNA molecules that bind and inhibit human thrombin. Nature. 355 (6360), 564-566 (1992).
- Paborsky, L. R., McCurdy, S. N., Griffin, L. C., Toole, J. J., Leung, L. L. The single-stranded DNA aptamer-binding site of human thrombin. The Journal of Biological Chemistry. 268 (28), 20808-20811 (1993).
- Carraro, C., et al. Behind the mirror: chirality tunes the reactivity and cytotoxicity of chloropiperidines as potential anticancer agents. ACS Medicinal Chemistry Letters. 10 (4), 552-557 (2019).
- Carraro, C., et al. Appended aromatic moieties in flexible bis-3-chloropiperidines confer tropism against pancreatic cancer cells. ChemMedChem. 16 (5), 860-868 (2021).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Onel, B., Wu, G., Sun, D., Lin, C., Yang, D. Electrophoretic mobility shift assay and dimethyl sulfate footprinting for characterization of G-quadruplexes and G-quadruplex-protein complexes. Methods in Molecular Biology. 2035, 201-222 (2019).

