RGBradford: Proteinkvantificering med et smartphone-kamera
Summary
Dette papir giver en protokol til proteinkvantificering ved hjælp af Bradford-analysen og en smartphone som analytisk enhed. Proteinniveauer i prøver kan kvantificeres ved hjælp af farvedata ekstraheret fra et billede af en mikroplade taget med en smartphone.
Abstract
Proteinkvantificering er en vigtig procedure inden for biovidenskabelig forskning. Blandt flere andre metoder er Bradford-analysen en af de mest anvendte. På grund af dets udbredelse er begrænsningerne og fordelene ved Bradford-analysen blevet udtømmende rapporteret, herunder flere ændringer af den oprindelige metode for at forbedre dens ydeevne. En af ændringerne i den oprindelige metode er brugen af et smartphone-kamera som et analytisk instrument. Ved at udnytte de tre former for Coomassie Brilliant Blue-farvestoffet, der findes under betingelserne i Bradford-analysen, beskriver dette papir, hvordan man nøjagtigt kvantificerer protein i prøver ved hjælp af farvedata ekstraheret fra et enkelt billede af en mikroplade. Efter udførelse af analysen i en mikroplade tages et billede ved hjælp af et smartphone-kamera, og RGB-farvedata ekstraheres fra billedet ved hjælp af en gratis og open source billedanalysesoftwareapplikation. Derefter bruges forholdet mellem blå og grøn intensitet (i RGB-skalaen) af prøver med ukendte koncentrationer af protein til at beregne proteinindholdet baseret på en standardkurve. Der observeres ingen signifikant forskel mellem værdier beregnet ved hjælp af RGB-farvedata og dem, der beregnes ved hjælp af konventionelle absorbansdata.
Introduction
Uanset downstream-brugen (f.eks. ELISA, enzymkinetik, western blotting, proteinoprensning og massespektrometri) er proteinkvantificering afgørende for nøjagtig analyse i biovidenskabelige laboratorier. Ud over deres anvendelse som sekundære aflæsninger (dvs. til beregning af relative niveauer af analysander pr. Masse protein) kan proteinniveauer i en prøve også være det ønskede output selv. For eksempel kan man være interesseret i proteinniveauer i fødevareressourcer1 eller i urin2. Der findes mange metoder til måling af proteinkoncentration i prøver3, herunder direkte UV-absorbansaflæsninger4, protein-kobberchelation 5,6, proteinfarvestofbindende kolorimetriske assays7 og proteinfarvestofbindende fluorescerende assays8. Relevansen af proteinkvantificering fremgår af tilstedeværelsen af to artikler, der beskriver proteinmålemetoder 5,7 i top-3 i den mest citerede litteratur 9,10. På trods af at mange forfattere forsømmer deres faktiske citat ved at citere ikke-primære referencer eller slet ikke citere noget, udgør de originale papirer, der beskriver Lowry-proteinanalysen og Bradford-proteinanalysen, > 200.000 citationer hver10.
Populariteten af Bradford-analysen stammer fra dens overkommelighed, enkelhed, hastighed og følsomhed. Analysen er baseret på interaktionen mellem proteiner og farvestoffet Coomassie Brilliant Blue G under sure forhold. Under betingelserne for analysen (dvs. lav pH) findes farvestoffet i tre former: en rød kationisk form med λmax ved 470 nm; en grøn neutral form med λmax ved 650 nm; og en blå anionisk form med λmax ved 590 nm11,12 (figur 1). Den kationiske form dominerer i fravær af proteiner. Når proteiner interagerer med farvestoffet, stabiliserer de den blå anioniske form, hvilket forårsager en mærkbar ændring i opløsningens farve, fra brunlig til blå. Normalt kvantificeres ændringen i koncentrationen af farvestoffets blå form specrophotometrisk, hvis absorbans ved 590-595 nm er proportional med mængden af protein i analysen.
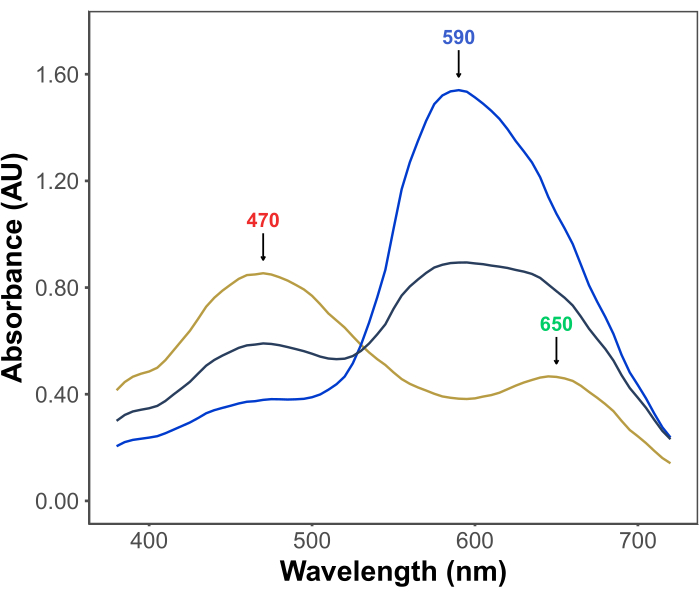
Figur 1: Coomassie brilliant blue G-absorptionsspektre under Bradford-assayets betingelser. De tre hovedtoppe er markeret med pile, der angiver λmax for de røde (470 nm), grønne (650 nm) og blå (590 nm) former af farvestoffet. Spektre blev registreret i fravær af protein (gul linje) og i nærværelse af 3 μg (grå linje) og 10 μg (blå linje) bovint serumalbumin. Klik her for at se en større version af denne figur.
Den udbredte anvendelse af Bradford-analysen har ført til identifikation af flere begrænsninger (f.eks. variable reaktioner på forskellige proteiner11 og interferens fra lipider13 og vaskemidler7) og udvikling af modifikationer for at forbedre dets ydeevne (f.eks. tilsætning af vaskemidler14,15, alkalisering14,16 og anvendelse af forholdet mellem absorbanser17). Ud over ændringer i selve analysen er brugen af alternative enheder, såsom smartphones eller kameraer, til at fange analytiske signaler også blevet beskrevet 18,19,20. Faktisk har udviklingen af metoder, der gør brug af smartphones som bærbare kemiske analysatorer, været et aktivt forskningsområde. Motivationen for brugen af smartphones stammer fra overkommeligheden, bærbarheden, brugervenligheden og den udbredte tilgængelighed af disse enheder.
Dette papir giver en protokol til proteinkvantificering ved hjælp af RGBradford-assay20, der bruger en smartphone som analytisk enhed. I modsætning til den originale RGBradford-publikation20 er der her indført en procedure, der strømliner farveudtrækningsprocessen. Det indebærer brug af en frit tilgængelig softwareapplikation til automatisk at udtrække farveinformation fra hver brønd i et mikropladebillede, hvilket sparer betydelig tid og kræfter. Dette er et alternativ til den tidligere metode til manuelt at erhverve farvedata fra hver brønd en efter en ved hjælp af en grafikredigeringssoftwareapplikation20. I sidste ende kan proteinniveauer i prøver kvantificeres ved hjælp af farvedata ekstraheret fra et billede af en mikroplade taget med en smartphone.
Protocol
Representative Results
Discussion
Dette papir beskriver RGBradford, en metode, der bruger et smartphone-kamera til at registrere data fra et Bradford-proteinassay, ekstrahere farvedata og nøjagtigt kvantificere proteinniveauer i biologiske prøver som oprindeligt beskrevet for nylig20. En forskel fra den originale RGBradford-metode er, at her blev der brugt en procedure til automatisk hentning af farvedata med et ImageJ-plugin22 . Den vigtigste nyhed ved RGBradford-metoden er brugen af RGB-data som analyti…
Declarações
The authors have nothing to disclose.
Acknowledgements
Dette arbejde blev finansieret af National Council for Scientific and Technological Development (CNPq, Brasilien) [bevillingsnumre 428048/2018-8 og 402556/2022-4] og University of Brasilia (Brasilien). Forfatteren takker Dr. Duarte Nuno Carvalho og Dr. Evelyn Santos (i3s, Porto, Portugal) for at give adgang til deres smartphones, der bruges i denne forskning.
Materials
| 96-well flat-bottom polystyrene microtiter plates | Jet Biofil, Guangzhou, China | TCP011096 | Any flat-bottom microplate compativle with optical reading will suffice. |
| Bovine serum albumin | Sigma-Aldrich, St. Louis, MO | A2153 | |
| Coomassie Brilliant Blue G | Sigma-Aldrich, St. Louis, MO | B0770 | |
| Ethyl alcohol | |||
| iPhone 11 | Apple | MWM02BR/A | Can be substituted with other smartphone equiped with a camera |
| iPhone 14 Pro | Apple | N/A | |
| Phosphoric acid | Sigma-Aldrich, St. Louis, MO | 695017 | |
| Redmi Note 9 Pro | XIAOMI | N/A | |
| S22 Ultra | Samsung | N/A | |
| SpectraMax 384 Plus. Microplate reader. | Molecular Devices, San Jose, CA | PLUS 384 | Any microplate reader capable of reading at 450 nm and 590 nm will work. This is optional. The method was actually created to dismiss the need of a microplate reader. |
Referências
- Zaguri, M., Kandel, S., Rinehart, S. A., Torsekar, V. R., Hawlena, D. Protein quantification in ecological studies: A literature review and empirical comparisons of standard methodologies. Methods in Ecology and Evolution. 12 (7), 1240-1251 (2021).
- Koga, T., et al. Mild electrical stimulation and heat shock ameliorates progressive proteinuria and renal inflammation in mouse model of Alport syndrome. PLoS One. 7 (8), e43852 (2012).
- Peterson, G. L. Determination of total protein. Methods in Enzymology. 91, 95-119 (1983).
- Goldfarb, A. R., Saidel, L. J., Mosovich, E. The ultraviolet absorption spectra of proteins. The Journal of Biological Chemistry. 193 (1), 397-404 (1951).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. The Journal of Biological Chemistry. 193 (1), 265-275 (1951).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Analytical Biochemistry. 150 (1), 76-85 (1985).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Datki, Z., et al. Application of BisANS fluorescent dye for developing a novel protein assay. PLoS One. 14 (4), e0215863 (2019).
- Van Noorden, R., Maher, B., Nuzzo, R. The top 100 papers. Nature. 514 (7524), 550-553 (2014).
- . Scopus Available from: https://www.scopus.com/ (2022)
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Analytical Biochemistry. 151 (2), 369-374 (1985).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie Blue G. Analytical Biochemistry. 209 (2), 258-266 (1993).
- Pande, S. V., Murthy, M. S. R. A modified micro-Bradford procedure for elimination of interference from sodium dodecyl sulfate, other detergents, and lipids. Analytical Biochemistry. 220 (2), 424-426 (1994).
- Gogstad, G. O., Krutnes, M. -. B. Measurement of protein in cell suspensions using the Commassie brilliant blue dye-binding assay. Analytical Biochemistry. 126 (2), 355-359 (1982).
- Friedenauer, S., Berlet, H. H. Sensitivity and variability of the Bradford protein assay in the presence of detergents. Analytical Biochemistry. 178 (2), 263-268 (1989).
- Stoscheck, C. M. Increased uniformity in the response of the Coomassie blue G protein assay to different proteins. Analytical Biochemistry. 184 (1), 111-116 (1990).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: Theoretical and experimental studies. Analytical Biochemistry. 236 (2), 302-308 (1996).
- Gee, C. T., Kehoe, E., Pomerantz, W. C. K., Penn, R. L. Quantifying protein concentrations using smartphone colorimetry: A new method for an established test. Journal of Chemical Education. 94 (7), 941-945 (2017).
- de Camargo, C., Vicentini, M., Gobbi, A., Martinez, D., Lima, R. Smartphone for point-of-care quantification of protein by Bradford assay. Journal of the Brazilian Chemical Society. 28 (4), 689-693 (2016).
- Moreira, D. C. RGBradford: Accurate measurement of protein concentration using a smartphone camera and the blue to green intensity ratio. Analytical Biochemistry. 655, 114839 (2022).
- Ernst, O., Zor, T. Linearization of the Bradford Protein Assay. Journal of Visualized Experiments. (38), 1918 (2010).
- Angelani, C. R., et al. A metabolic control analysis approach to introduce the study of systems in biochemistry: the glycolytic pathway in the red blood cell: Metabolic control analysis and the glycolytic pathway. Biochemistry and Molecular Biology Education. 46 (5), 502-515 (2018).

