Profilo fenotipico dei neuroni dopaminergici del mesencefalo derivati da cellule staminali umane
Summary
Questo protocollo descrive la coltura cellulare di neuroni dopaminergici del mesencefalo umano, seguita da colorazione immunologica e generazione di profili fenotipici neuronali da immagini microscopiche acquisite ad alto contenuto che consentono l’identificazione di variazioni fenotipiche dovute a modulazioni genetiche o chimiche.
Abstract
Il morbo di Parkinson (PD) è legato a una serie di processi biologici cellulari che causano la perdita di neuroni dopaminergici del mesencefalo (mDA). Molti attuali modelli cellulari di PD in vitro mancano di complessità e non tengono conto di fenotipi multipli. Il profilo fenotipico nei neuroni mDA derivati da cellule staminali pluripotenti indotte umane (iPSC) può affrontare queste carenze misurando simultaneamente una serie di fenotipi neuronali in un tipo di cellula rilevante per la PD in parallelo. In questo articolo, descriviamo un protocollo per ottenere e analizzare i profili fenotipici da neuroni mDA umani disponibili in commercio. Un pannello di colorazione fluorescente specifico per i neuroni viene utilizzato per visualizzare i fenotipi correlati alla proteina nucleare, alla α-sinucleina, alla tirosina idrossilasi (TH) e alla proteina 2 associata ai microtubuli (MAP2). Il protocollo di profilazione fenotipica descritto è scalabile in quanto utilizza piastre a 384 pozzetti, manipolazione automatica dei liquidi e microscopia ad alto rendimento. L’utilità del protocollo è esemplificata utilizzando neuroni mDA donatori sani e neuroni mDA portatori della mutazione G2019S legata al PD nel gene della chinasi ripetuta 2 ricca di leucina (LRRK2). Entrambe le linee cellulari sono state trattate con l’inibitore della chinasi LRRK2 PFE-360 e sono stati misurati i cambiamenti fenotipici. Inoltre, dimostriamo come i profili fenotipici multidimensionali possano essere analizzati utilizzando metodi di classificazione supervisionati basati sul clustering o sull’apprendimento automatico. Il protocollo descritto interesserà in particolare i ricercatori che lavorano sulla modellazione delle malattie neuronali o che studiano gli effetti dei composti chimici nei neuroni umani.
Introduction
Una varietà di processi biologici cellulari sono disturbati nella malattia di Parkinson (PD). Ad esempio, la disfunzione mitocondriale, lo stress ossidativo, i difetti di degradazione delle proteine, l’interruzione del traffico vescicolare e la funzione endolisosomiale sono stati associati alla perdita di neuroni dopaminergici del mesencefalo (mDA), sono comunemente osservati nella PD1. Pertanto, la malattia di Parkinson sembra coinvolgere molteplici meccanismi patologici che possono interagire e peggiorarsi a vicenda. Un modo utile per studiare questa interazione meccanicistica è la creazione di un’impronta digitale fenotipica completa o di un profilo dei neuroni dopaminergici del mesencefalo (mDA).
La profilazione fenotipica è un approccio che prevede la creazione di un profilo di un campione basato su una raccolta di caratteristiche misurabili e, in secondo luogo, comporta la formulazione di previsioni su un campione basato su questo profilo 2,3. L’obiettivo della profilazione è quello di acquisire una vasta gamma di caratteristiche, alcune delle quali potrebbero non essere state precedentemente associate a una malattia o a un trattamento3. Di conseguenza, la profilazione può rivelare processi biologici inaspettati. La profilazione fenotipica si basa tipicamente su cellule colorate con fluorescenza e saggi standardizzati, come il Cell Painting, sono stati sviluppati per creare profili fenotipici4. Recentemente, la profilazione fenotipica è stata, ad esempio, applicata per la caratterizzazione di piccole molecole o per la predizione accurata di sottotipi di PD basati esclusivamente su fibroblasti derivati da pazienti 5,6. Nonostante questi progressi, il profilo fenotipico è stato raramente applicato a cellule differenziate post-mitotiche, come i neuroni mDA derivati da cellule staminali pluripotenti indotte umane (iPSC) che esprimono mutazioni legate al PD come LRRK2 G2019S. Le sfide significative dei modelli derivati da iPSC includono la presenza di caratteristiche patologiche sottili o variabili tra i lotti di differenziazione o i genotipi e il fatto che i fenotipi isolati di PD non catturano l’intera complessità della malattia. Inoltre, mentre i modelli neuronali di iPSC sono fisiologicamente rilevanti, sono raramente utilizzati nei processi di scoperta di farmaci per la malattia di Parkinson a causa di preoccupazioni sulla complessità tecnica 7,8.
In precedenza abbiamo sviluppato una solida metodologia per misurare più fenotipi fisiopatologici correlati al PD nei neuroni mDA umani che sono sensibili ai cambiamenti fenotipici indotti da composti genetici e chimici9. Questo articolo descrive in dettaglio una versione ulteriormente ottimizzata di questa metodologia per creare profili fenotipici da neuroni mDA (Figura 1). Questo protocollo presenta diversi vantaggi rispetto agli approcci di profilazione fenotipica descritti in precedenza, come l’uso di neuroni mDA di alta qualità e la riproducibilità tecnica. Per la prima volta, questo protocollo consente la profilazione fenotipica nei neuroni mDA post-mitotici fisiologicamente rilevanti dopo perturbazioni chimiche in modo altamente scalabile. I neuroni mDA completamente differenziati e crioconservati sono disponibili in commercio, riducendo significativamente la variabilità della differenziazione da lotto a lotto. In secondo luogo, la variabilità tecnica può essere ulteriormente ridotta utilizzando un disegno sperimentale ben definito (ad esempio, la durata della coltura o evitando i pozzetti di bordo), la manipolazione automatizzata dei liquidi e la microscopia automatizzata. Inoltre, le fasi iniziali dell’analisi del profilo fenotipico utilizzando approcci di clustering non supervisionato o classificazione supervisionata sono descritte qui, indicando come possono essere analizzati i dati di profilazione fenotipica. Questo protocollo sarà utile per i ricercatori interessati ai cambiamenti fenotipici dei neuroni mDA indotti da perturbazioni genetiche o chimiche, in particolare quando è richiesta una configurazione di studio altamente scalabile, ad esempio durante le campagne di screening o quando gli effetti di un numero minore di composti devono essere studiati, ad esempio, per determinare gli effetti tossici. In sintesi, si prevede che l’applicazione del profilo fenotipico dei neuroni umani sia una tecnica preziosa per studiare fenotipi complessi correlati alla malattia e caratterizzare gli effetti cellulari dei farmaci candidati.
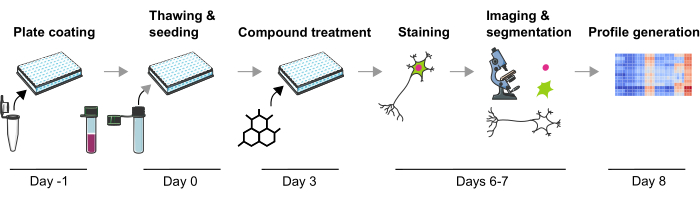
Figura 1: Rappresentazione schematica del protocollo sperimentale per generare profili fenotipici basati su immagini da neuroni mDA umani derivati da iPSC. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
La profilazione fenotipica è una tecnica per misurare un gran numero di fenotipi nelle cellule applicando colorazioni fluorescenti, microscopia e analisi delle immagini3. I profili fenotipici possono essere ottenuti e confrontati tra linee cellulari o altre condizioni sperimentali per comprendere i cambiamenti complessi nella biologia cellulare che potrebbero passare inosservati quando si utilizza una singola lettura. Qui descriviamo l’applicazione del profilo fenotipico ai neuroni mDA umani deri…
Declarações
The authors have nothing to disclose.
Acknowledgements
Gli autori desiderano ringraziare tutti i colleghi di Ksilink per il loro prezioso aiuto e le discussioni che hanno portato alla progettazione del protocollo presentato.
Materials
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse – Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit – Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
Referências
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson’s disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson’s disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson’s Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson’s Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson’s disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson’s disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson’s disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson’s disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).

