बैक्टीरियल के लिए दो घटक नियामक प्रणाली जीन लक्ष्य निर्धारित करने के लिए डीएनए आत्मीयता शुद्ध चिप (डीएपी चिप) विधि
Summary
इस वीडियो लेख दो घटक प्रणाली प्रतिक्रिया नियामकों के लिए जीन लक्ष्य और बाध्यकारी साइटों का निर्धारण करने के लिए इन विट्रो माइक्रोएरे आधारित विधि का वर्णन करता है.
Abstract
ऐसी चिप चिप के रूप में विवो तरीकों में प्रतिलेखन कारकों के लिए वैश्विक जीन लक्ष्य निर्धारित किया अच्छी तरह से स्थापित तकनीक है. हालांकि, वे uncharacterized सक्रियण शर्तों के साथ बैक्टीरियल दो घटक नियामक प्रणाली की खोज में सीमित इस्तेमाल के हैं. अद्वितीय संकेतों की उपस्थिति में सक्रिय जब इस तरह की प्रणाली केवल प्रतिलेखन विनियमित. इन संकेतों को अक्सर अज्ञात होते हैं, इसलिए इस वीडियो लेख में वर्णित इन विट्रो माइक्रोएरे आधारित पद्धति जीन लक्ष्य और प्रतिक्रिया नियामकों के लिए बाध्यकारी साइटों का निर्धारण किया जा सकता है. यह डीएनए आत्मीयता शुद्ध चिप विधि एक अनुक्रम जीनोम के साथ किसी भी जीव में किसी भी शुद्ध नियामक के लिए इस्तेमाल किया जा सकता है. प्रोटोकॉल शुद्ध किया टैग प्रोटीन एक कस्टम टाइलिंग सरणी के लिए डीएनए और संकरण के फ्लोरोसेंट लेबलिंग द्वारा पीछा प्रोटीन वाली डीएनए, sheared जीनोमिक डीएनए के लिए बाध्य है और फिर आत्मीयता सफ़ाई करने के लिए अनुमति शामिल है. Specifi के लिए परख अनुकूलन करने के लिए इस्तेमाल किया जा सकता है कि कदम पूर्ववर्तीसी नियामकों भी वर्णित हैं. सरणी डेटा विश्लेषण द्वारा उत्पन्न चोटियों तो प्रयोगात्मक पुष्टि कर रहे हैं जो बाध्यकारी साइट रूपांकनों, भविष्यवाणी करने के लिए इस्तेमाल कर रहे हैं. आकृति भविष्यवाणियों आगे निकट से संबंधित प्रजातियों में orthologous प्रतिक्रिया नियामकों के जीन लक्ष्य निर्धारित करने के लिए इस्तेमाल किया जा सकता है. हम जीन लक्ष्य निर्धारित करने और साइट बेरी बंधन और इस प्रकार पर्यावरण जीवाणु Desulfovibrio vulgaris Hildenborough में एक sigma54 निर्भर प्रतिक्रिया नियामक DVU3023 के लिए समारोह की भविष्यवाणी के द्वारा इस पद्धति के प्रयोग को प्रदर्शित करता है.
Introduction
जीवित और फूलने के लिए बैक्टीरिया की क्षमता वे देखती है और अपने वातावरण में perturbations के लिए प्रतिक्रिया करने में सक्षम होते हैं कितनी अच्छी तरह पर निर्भर है, और यह बदले में उनके संकेत पारगमन प्रणाली पर निर्भर है. सिगनल सिस्टम एक जीवाणु encodes की संख्या अपने "माइक्रोबियल बुद्धि" बुलाया गया है और अपने पर्यावरण की परिवर्तनशीलता और कई संकेत समझ करने की क्षमता और ठीक धुन अपनी प्रतिक्रिया 1 दोनों का एक संकेत हो सकता है. दो घटक संकेत पारगमन प्रणाली (टीसीएस) बैक्टीरिया द्वारा इस्तेमाल किया सबसे अधिक प्रचलित सिगनल व्यवस्था कर रहे हैं, और वे बाहरी संकेत होश और एक प्रेरक प्रतिक्रिया नियामक (आरआर) 2 के लिए phosphorylation के माध्यम से पहुंचाता है कि एक हिस्टडीन kinase (एच) से मिलकर बनता है. आरआरएस एक उत्पादन डोमेन की विविधता और इस प्रकार विभिन्न प्रेरक मोड में हो सकता है, लेकिन सबसे आम प्रतिक्रिया डोमेन 1 बाध्यकारी एक डीएनए के माध्यम से transcriptional विनियमन है. लगा संकेतों और वीएएस की इसी कार्योंTCSs की टी बहुमत अनजान रहते हैं.
ऐसी चिप चिप के रूप में विवो तरीकों नियमित प्रतिलेखन 3 कारकों के जीनोमिक बाध्यकारी साइटों के निर्धारण के लिए उपयोग किया जाता है सक्रिय शर्तों या संकेतों में जाना जाता है, वे केवल बैक्टीरिया के दो घटक प्रणाली आरआरएस के लिए इस्तेमाल किया जा सकता है. अक्सर एक टीसीएस को सक्रिय कि पर्यावरण cues उनके जीन लक्ष्य से निर्धारित करने के लिए कठिन हैं. इन विट्रो माइक्रोएरे यहाँ वर्णित आधारित परख प्रभावी ढंग से और तेजी से जीन लक्ष्य निर्धारित करने और TCSs के कार्यों की भविष्यवाणी करने के लिए इस्तेमाल किया जा सकता है. यह परख आरआरएस phosphorylated और इस तरह एसिटाइल फॉस्फेट 4 की तरह छोटे अणु दाताओं का उपयोग इन विट्रो में सक्रिय किया जा सकता है कि इस तथ्य का लाभ लेता है.
इस विधि में डीएनए आत्मीयता शुद्ध चिप (चित्रा 1) के लिए डीएपी चिप का नाम है, ब्याज की आरआर जीन ई. में एक उनकी टैग के साथ क्लोन है कोलाई, और एक बाद में शुद्ध किया टैग प्रोटीन द्वि की अनुमति दी हैएन डी जीनोमिक डीएनए sheared लिए. प्रोटीन वाली डीएनए तो आत्मीयता शुद्धि से समृद्ध है, समृद्ध और इनपुट डीएनए परिलक्षित कर रहे हैं, fluorescently एक साथ जमा और कस्टम ब्याज के जीव (चित्रा 1) के लिए किया जाता है कि एक खपरैल का छत सरणी संकरित, लेबल. माइक्रोएरे प्रयोगों कलाकृतियों के अधीन हैं और इसलिए अतिरिक्त कदम परख अनुकूलन करने के लिए कार्यरत हैं. ऐसा ही एक कदम (चित्रा 2 में कार्यप्रवाह देखें) electrophoretic गतिशीलता पारी assays (EMSA) का उपयोग करते हुए अध्ययन के तहत आरआर के लिए एक लक्ष्य निर्धारित करने के लिए प्रयास करने के लिए है. फिर, जीनोमिक डीएनए और डीएपी कदम को बंधन निम्नलिखित, प्रोटीन ही सीमित है और इनपुट डीएनए इस प्रकार के इष्टतम बाध्यकारी शर्तों की पुष्टि, सकारात्मक लक्ष्य इनपुट अंश के सापेक्ष प्रोटीन बाध्य अंश में समृद्ध है यह देखने के लिए qPCR द्वारा जांच कर रहे हैं आरआर (चित्रा 2). सरणी संकरण के बाद, डेटा जीनोमिक loci का संकेत उच्च तीव्रता संकेत की चोटियों को खोजने के लिए विश्लेषण कर रहे हैं जहां प्रोटीन घंटेविज्ञापन के लिए बाध्य. कार्य प्राप्त जीन लक्ष्य पर आधारित आरआर के लिए भविष्यवाणी की जा सकती है. लक्ष्य जीनोमिक loci तो प्रयोगात्मक EMSAs (चित्रा 2) का उपयोग कर पुष्टि कर रहे हैं जो बाध्यकारी साइट रूपांकनों, भविष्यवाणी करने के लिए इस्तेमाल कर रहे हैं. आरआर के लिए कार्यात्मक भविष्यवाणियों और जीन लक्ष्य तो बारीकी इसी तरह बाध्यकारी रूपांकनों के लिए उन जीनोम (चित्रा 2) स्कैनिंग द्वारा orthologous आरआरएस सांकेतिक शब्दों में बदलना है कि प्रजातियों से संबंधित के लिए बढ़ाया जा सकता है. डीएपी चिप विधि पहले जहां कोई नहीं था एक टीसीएस के लिए जानकारी का खजाना प्रदान कर सकते हैं. विधि भी प्रोटीन शुद्ध किया जा सकता है और डीएनए बाध्यकारी शर्तों निर्धारित किया जा सकता है अगर किसी भी transcriptional नियामक के लिए प्रयोग किया जाता है, और एक जीनोम अनुक्रम के साथ ब्याज की किसी भी जीव के लिए उपलब्ध हो सकते हैं.
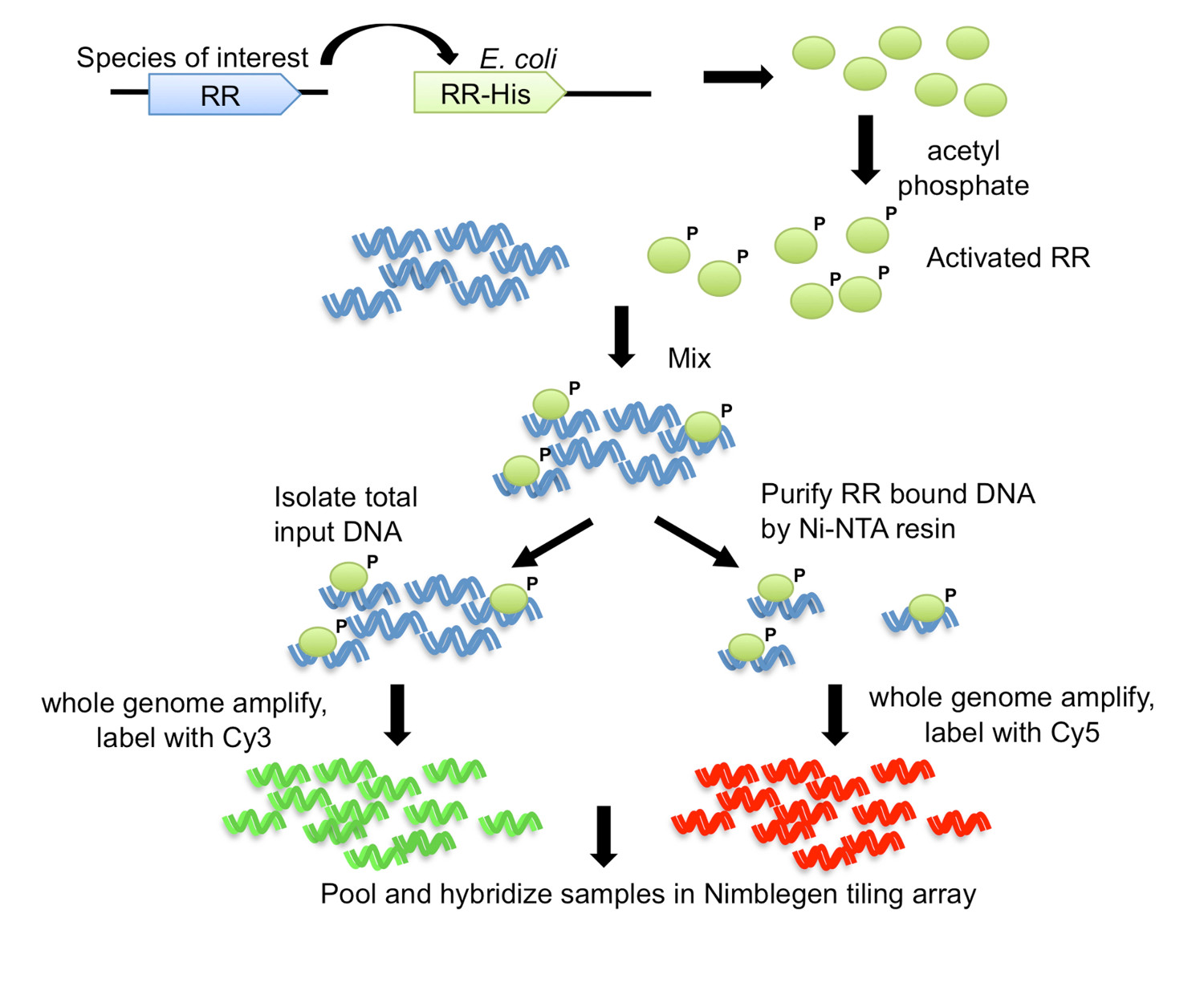
चित्रा 1. डीएनए आत्मीयता शुद्ध चिप (डीएपी-चिप) रणनीति 7. ब्याज के जीव से आरआर जीन एक ई. में एक carboxy टर्मिनल उनकी टैग के साथ क्लोन है कोली अभिव्यक्ति तनाव. शुद्ध किया उसका टैग प्रोटीन एसिटाइल फॉस्फेट के साथ phosphorylation से सक्रिय, और sheared जीनोमिक डीएनए के साथ मिलाया जाता है. बाकी नी NTA राल का उपयोग आत्मीयता शुद्धि के अधीन है, जबकि बाध्यकारी प्रतिक्रिया के एक विभाज्य, इनपुट डीएनए के रूप में सहेजा जाता है. इनपुट और आरआर वाली डीएनए प्रवर्धित पूरे जीनोम हैं, और क्रमशः Cy3 और Cy5, के साथ लेबल. लेबल डीएनए एक साथ जमा और फिर जीन लक्ष्य निर्धारित करने के लिए विश्लेषण किया जाता है जो एक खपरैल का छत सरणी, संकरित है. चित्रा संशोधित और क्रिएटिव 7 से उपयोग कर reprinted.
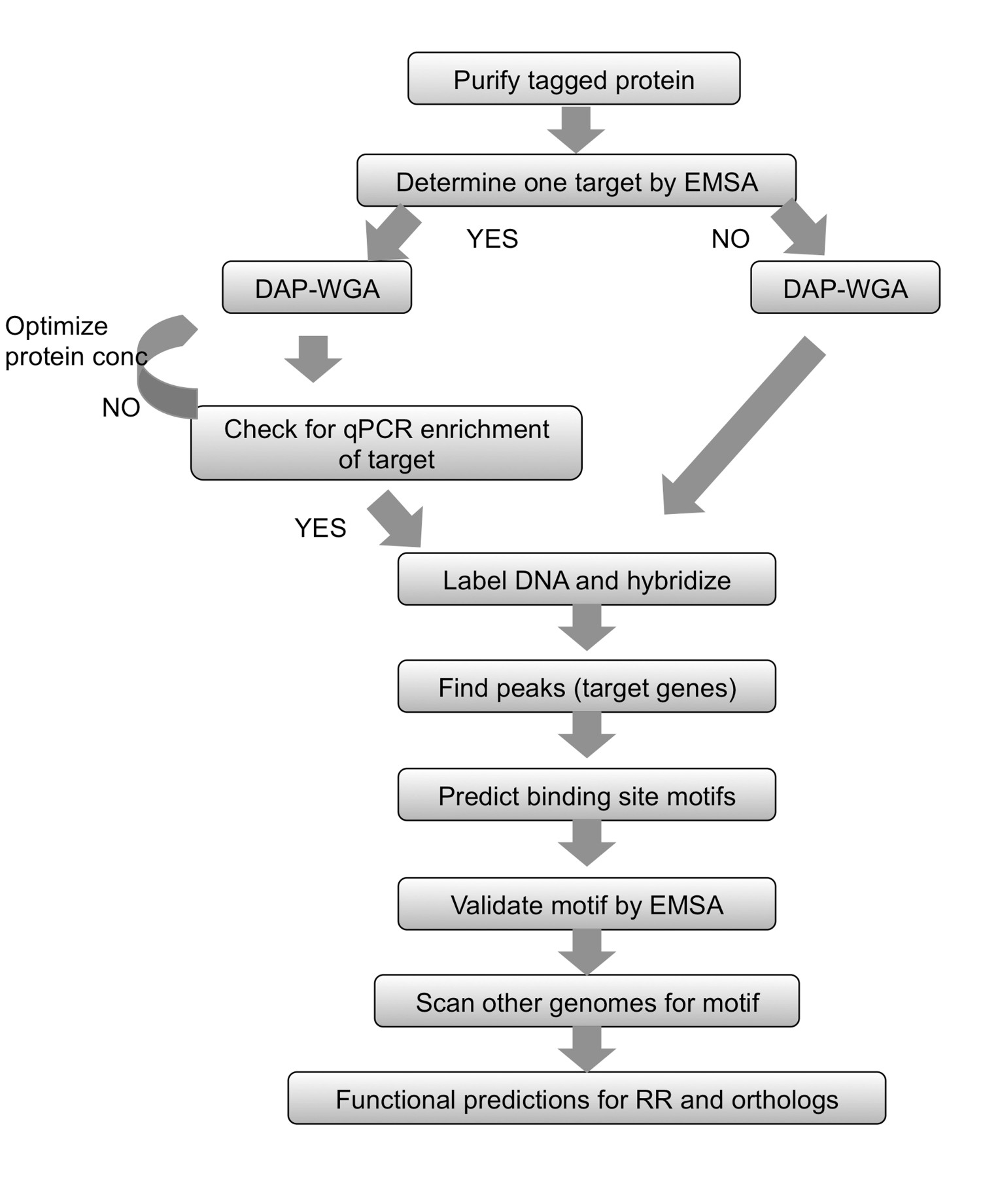
कार्यप्रवाह के चित्रा 2. सारांश. किसी भी शुद्ध किया टैग किया protei के लिएएन, EMSA का उपयोग कर एक लक्ष्य निर्धारित करने से शुरू करते हैं. (WGA) समृद्ध और इनपुट डीएनए बढ़ाना जीनोमिक डीएनए और फिर डीएनए आत्मीयता-शुद्ध (डीएपी) और पूरे जीनोम बाध्य करने के लिए प्रोटीन की अनुमति दें. एक जीन लक्ष्य जाना जाता है, में जाना लक्ष्य प्रोटीन बाध्य अंश में समृद्ध है कि यह सुनिश्चित करने के लिए qPCR का उपयोग करें. कोई लक्ष्य निर्धारित किया जा सकता है, डीएनए लेबलिंग और सरणी संकरण करने के लिए सीधे आगे बढ़ना. QPCR द्वारा संवर्धन देखा नहीं जा सकता है, तो बाध्यकारी प्रोटीन gDNA और विभिन्न प्रोटीन मात्रा का उपयोग डीएपी-WGA चरणों को दोहराएँ. चोटियों पाते हैं और जीनों को लक्ष्य करने के लिए उन्हें नक्शा करने के लिए सरणी विश्लेषण का प्रयोग करें. बाध्यकारी साइट रूपांकनों भविष्यवाणी करने के लिए लक्ष्य जीन के अपस्ट्रीम क्षेत्रों का प्रयोग करें. प्रयोगात्मक EMSAs का उपयोग रूपांकनों मान्य करें. अध्ययन के तहत आरआर के orthologs एन्कोडिंग संबंधित प्रजातियों के जीनोम को स्कैन करने के लिए आकृति का प्रयोग करें, और साथ ही उन प्रजातियों में लक्षित जीन का अनुमान है. प्राप्त जीन लक्ष्य के आधार पर, आरआर और उसके orthologs की शारीरिक समारोह की भविष्यवाणी की जा सकती है. चित्रा संशोधित और रचनात्मक सी का उपयोग पुनर्मुद्रितommons 7 से लाइसेंस.
Protocol
Representative Results
Discussion
यहाँ वर्णित डीएपी चिप विधि सफलतापूर्वक एक एक प्रतिनिधि परिणाम के रूप में यहाँ दिखाया गया है, जिनमें से Desulfovibrio vulgaris Hildenborough 7 में कई आरआरएस के लिए जीन लक्ष्य निर्धारित किया गया था. आरआर DVU3023 के लिए, एक उम्?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
हम वीडियो की शूटिंग के लिए तैयार करने में उसकी मदद के लिए और तकनीक के प्रदर्शन के लिए एमी चेन धन्यवाद. पहेली द्वारा आयोजित इस काम: जीन और आणविक विधानसभाओं (http://enigma.lbl.gov) के साथ पारिस्थितिकी प्रणालियों और नेटवर्क एकीकृत, लॉरेंस बर्कले राष्ट्रीय प्रयोगशाला में एक वैज्ञानिक फोकस क्षेत्र कार्यक्रम, विज्ञान, जैव के कार्यालय के कार्यालय और द्वारा समर्थित किया गया अनुबंध सं डे-AC02-05CH11231 के तहत अमेरिका के ऊर्जा विभाग के पर्यावरण अनुसंधान,.
Materials
| Name of Material/Equipment | Company | Catalog number | Comments |
| HisTrapFF column (Ni-Sepharose column) | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5255-01 | |
| Akta explorer (FPLC instrument) | GE Lfe Sciences, Pittsburgh, PA, USA | ||
| HiPrep 26/10 Desalting column | GE Lfe Sciences, Pittsburgh, PA, USA | 17-5087-01 | |
| Qiaquick Gel extraction kit | Qiagen Inc, Valencia, CA, USA | 28704 | |
| Biotin-labeled oligonucleotides | Integrated DNA Technologies | N/A | |
| 6% polyacrylamide-0.5X TBE precast mini DNA retardation gel | Life Technologies, Grand Island, NY, USA | EC63652BOX | Alternately, you can pour your own gel. |
| Nylon membrane | EMD Millipore, Billerica, MA, USA | INYC00010 | |
| Trans-Blot SD Semi-dry electrophoretic transfer cell | Biorad, Hercules, CA, USA | 170-3940 | |
| Extra thick blot paper, 8 x 13.5 cm | Biorad, Hercules, CA, USA | 170-3967 | |
| UV crosslinker Model XL-1000 | Fisher Scientific | 11-992-89 | |
| Nucleic Acid chemiluminescent detection kit (Pierce) | Thermo fisher Scientific, Rockford, IL, USA | 89880 | |
| Ni-NTA agarose resin | Qiagen Inc, Valencia, CA, USA | 30210 | |
| GenomePlex Whole genome amplification kit (Fragmentation buffer, library preparation buffer, library stabilization solution, library preparation enzyme, 10X amplification master mix, WGA polymerase ) | Sigma-Aldrich, St. Louis, MO, USA | WGA2-50RXN | |
| Nanodrop ND-1000 | Thermo Scientific, Wilmington, DE, USA | For quantitation of DNA | |
| Perfecta Sybr Green SuperMix, with ROX | Quanta biosciences | 95055-500 | Any Sybr Green PCR mix may be used |
| PlateMax Ultra clear heat sealing film for qPCR | Axygen | ||
| 96 well clear low profile PCR microplate | Life Technologies, Grand Island, NY, USA | PCR-96-LP-AB-C | |
| Applied Biosystems StepOne Plus Real time PCR system | Life Technologies, Grand Island, NY, USA | 4376600 | Any real time PCR system may be used |
| Qiaquick PCR purification kit | Qiagen Inc, Valencia, CA, USA | 28104 | Any PCR clean up kit may be used |
| Cy3/Cy5-labeled nonamers | Trilink biotechnologies, San Diego, CA, USA | N46-0001, N46-0002 | |
| Klenow polymerase 50,000U/ml, 3'-5' exo- | New England Biolabs, Ipswich, MA | M0212M | |
| Hybridization system | Roche-Nimblegen, Madison, WI, USA | N/A | This company no longer makes arrays or related items, so alternate sources such as Agilent or Affymetrix will need to be used, |
| Custom printed microarrays and mixers | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Hybridization kit (2X Hybridization buffer, Hybridization component A, Alignment oligo) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| Wash buffer kit (10X Wash buffer I, II, III, 1 M DTT) | Roche-Nimblegen, Madison, WI, USA | N/A | |
| GenePix 4200A microarray scanner | Molecular Devices, Sunnyvale CA, USA | This model has been replaced by superior ones | |
| GenePix Pro microarray software | Molecular Devices, Sunnyvale CA, USA | ||
| Nimblescan v.2.4, ChIP-chip analysis software | Roche-Nimblegen, Madison, WI, USA | N/A | |
References
- Galperin, M. Y. Diversity of structure and function of response regulator output domains. Curr Opin Microbiol. 13, 150-159 (2010).
- Casino, P., Rubio, V., Marina, A. The mechanism of signal transduction by two-component systems. Curr Opin Struct Biol. 20, 763-771 (2010).
- Aparicio, O., Geisberg, J. V., Struhl, K. Chromatin immunoprecipitation for determining the association of proteins with specific genomic sequences in vivo. Curr Protoc Cell Biol. 17 (17), (2004).
- McCleary, W. R., Stock, J. B. Acetyl phosphate and the activation of two-component response regulators. J Biol Chem. 269, 31567-31572 (1994).
- Novichkov, P. S., et al. RegPrecise web services interface: programmatic access to the transcriptional regulatory interactions in bacteria reconstructed by comparative genomics. Nucleic Acids Res. 40, 604-608 (2012).
- Studholme, D. J., Buck, M., Nixon, T. Identification of potential sigma(N)-dependent promoters in bacterial genomes. Microbiology. 146 (12), 3021-3023 (2000).
- Rajeev, L., et al. Systematic mapping of two component response regulators to gene targets in a model sulfate reducing bacterium. Genome Biol. 12, 99 (2011).
- Crooks, G. E., Hon, G., Chandonia, J. M., Brenner, S. E. WebLogo: a sequence logo generator. Genome Res. 14, 1188-1190 (2004).
- Barbieri, C. M., Mack, T. R., Robinson, V. L., Miller, M. T., Stock, A. M. Regulation of response regulator autophosphorylation through interdomain contacts. J Biol Chem. 285, 32325-32335 (2010).
- Bourret, R. B. Receiver domain structure and function in response regulator proteins. Curr Opin Microbiol. 13, 142-149 (2010).
- Gao, R., Stock, A. M. Molecular strategies for phosphorylation-mediated regulation of response regulator activity. Curr Opin Microbiol. 13, 160-167 (2010).
- Hung, D. C., et al. Oligomerization of the response regulator ComE from Streptococcus mutans is affected by phosphorylation. J Bacteriol. 194, 1127-1135 (2012).
- Ladds, J. C., et al. The response regulator Spo0A from Bacillus subtilis is efficiently phosphorylated in Escherichia coli. FEMS Microbiol Lett. 223, 153-157 (2003).
- Wen, Y., Feng, J., Scott, D. R., Marcus, E. A., Sachs, G. Involvement of the HP0165-HP0166 two-component system in expression of some acidic-pH-upregulated genes of Helicobacter pylori. J Bacteriol. 188, 1750-1761 (2006).
- Liu, X., Noll, D. M., Lieb, J. D., Clarke, N. D. DIP-chip: rapid and accurate determination of DNA-binding specificity. Genome Res. 15, 421-427 (2005).
- Gossett, A. J., Lieb, J. D. DNA Immunoprecipitation (DIP) for the Determination of DNA-Binding Specificity. CSH Protoc. 2008, (2008).
- Mascher, T., et al. The Streptococcus pneumoniae cia regulon: CiaR target sites and transcription profile analysis. J Bacteriol. 185, 60-70 (2003).
- Park, P. J. ChIP-seq: advantages and challenges of a maturing technology. Nat Rev Genet. 10, 669-680 (2009).
- Lefrancois, P., Zheng, W., Snyder, M. ChIP-Seq using high-throughput DNA sequencing for genome-wide identification of transcription factor binding sites. Methods Enzymol. 470, 77-104 (2010).
- Ho, J. W., et al. ChIP-chip versus ChIP-seq: lessons for experimental design and data analysis. BMC Genomics. 12, 134 (2011).
- Liu, X., Lee, C. K., Granek, J. A., Clarke, N. D., Lieb, J. D. Whole-genome comparison of Leu3 binding in vitro and in vivo reveals the importance of nucleosome occupancy in target site selection. Genome Res. 16, 1517-1528 (2006).

