Fluorescence Time-lapse avbildning av Complete<em> S. venezuelae</em> Life Cycle använda en mikroflödessystem enhet
Summary
Streptomyces are characterized by a complex life cycle that has been experimentally challenging to study by cell biological means. Here we present a protocol to perform fluorescence time-lapse microscopy of the complete life cycle by growing Streptomyces venezuelae in a microfluidic device.
Abstract
Live-cell imaging of biological processes at the single cell level has been instrumental to our current understanding of the subcellular organization of bacterial cells. However, the application of time-lapse microscopy to study the cell biological processes underpinning development in the sporulating filamentous bacteria Streptomyces has been hampered by technical difficulties.
Here we present a protocol to overcome these limitations by growing the new model species, Streptomyces venezuelae, in a commercially available microfluidic device which is connected to an inverted fluorescence widefield microscope. Unlike the classical model species, Streptomyces coelicolor, S. venezuelae sporulates in liquid, allowing the application of microfluidic growth chambers to cultivate and microscopically monitor the cellular development and differentiation of S. venezuelae over long time periods. In addition to monitoring morphological changes, the spatio-temporal distribution of fluorescently labeled target proteins can also be visualized by time-lapse microscopy. Moreover, the microfluidic platform offers the experimental flexibility to exchange the culture medium, which is used in the detailed protocol to stimulate sporulation of S. venezuelae in the microfluidic chamber. Images of the entire S. venezuelae life cycle are acquired at specific intervals and processed in the open-source software Fiji to produce movies of the recorded time-series.
Introduction
Streptomyces är jordlevande bakterier som kännetecknas av en komplex utvecklingscykel som involverar morfologisk differentiering från en flercellig, närings sophantering mycel vilande, unigenomic sporer 1-3.
Under gynnsamma tillväxtbetingelser, startar en typisk Streptomyces spor att gro genom strängsprutning av en eller två bakterierören (figur 1). Dessa rör avlånga med spets förlängning och växa till en förgrenad hyphal nätverk kallas vegetativt mycel. Polar tillväxt och hyfal förgrening leds av den väsentliga protein DivIVA. Detta coiled-coil-proteinet är en del av en stor cytoplasmisk komplex kallas polarisome, vilket är avgörande för införandet av nya cellhölje material på den utskjutande spetsen 4-7. Under vegetativ tillväxt, de hyfernas trådarna blir fack med ovanliga bildningen av så kallade kors väggar 8. Bildningen av dessa tvärväggar re quires FtsZ, den tubulinliknande cytoskelettala protein som krävs för celldelning i de flesta bakterier 9. I Streptomyces, men dessa vegetativa kors väggar inte leder till sammandragning och cell-cell separation och därför mycelmassan kvarstår som ett nätverk av sammankopplade syncytial fack. Som svar på näringsbegränsning och andra signaler som inte är väl förstådda, specialiserad lufthyfer bryta sig loss från det vegetativa mycelet och växa upp i luften 3. Uppförandet av dessa strukturer initierar den reproduktiva fasen av utveckling, under vilken länge flera genomisk lufthyfer blivit uppdelad i dussintals lika stora unigenomic prespore fack. Denna massiva celldelnings händelse drivs av synkrona sammandragning av flera FtsZ ringar inom enda sporogenic hyfer 2,10. Morfologisk differentiering kompletteras med frisättning av vilande, tjocka väggar, pigmenterade sporer.
t "fo: keep-together.within-page =" 1 ">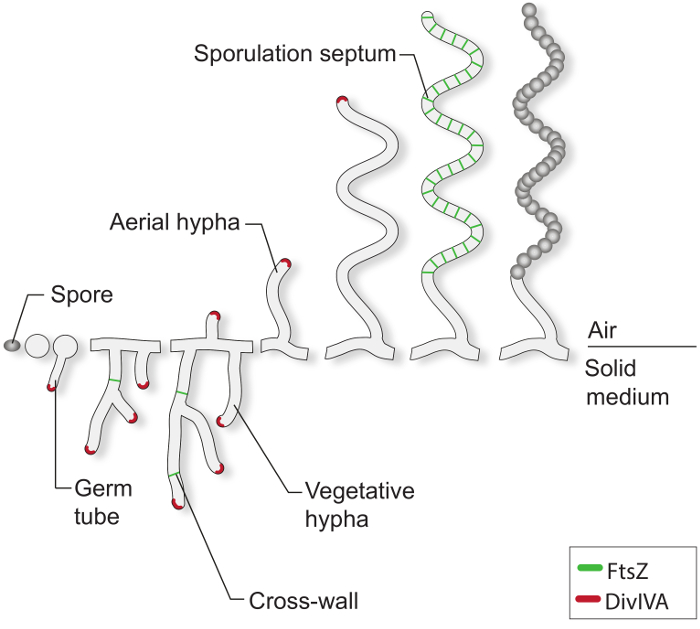
Figur 1:. Streptomyces livscykel på fasta medier Detta är en modell av livscykeln baserad på klassiska studier av S. coelicolor växer på agarplattor. Den cellulära utveckling av en spor börjar med bildningen av en eller två bakterie tuber, som växer genom spets förlängning för att bilda ett nätverk av förgrenande hyfer. Polär tillväxt och förgrening av den vegetativa hyfer riktas av DivIVA (röd). Bildandet av vegetativa tvärväggar kräver FtsZ (grön). Som svar på begränsningar närings och andra signaler är lufthyfer uppfördes. Gripandet av antenn tillväxt tätt samordnas med montering av en stege av FtsZ-ringar, som ger upphov till sporulering septa som compartmentalize den sporogenic hyfer i lådliknande prespore fack. Dessa avdelningar montera en tjock spor vägg och är slutligen Released som mogna pigmente sporer.
De viktigaste utvecklings händelserna i Streptomyces livscykel är väl karakteriserade 1,3. Men vad är fortfarande sällsynta är cellbiologiska studier som använder fluorescenstidsförlopp mikroskopi för att ge insikt i de subcellulära processer som ligger till grund differentiering, såsom protein lokalisering dynamik, kromosom rörelse och utvecklings kontrollerad celldelning. Live-cell imaging av Streptomyces utveckling har varit utmanande på grund av komplexiteten i livscykeln och de fysiologiska egenskaperna hos organismen. Tidigare studier på vegetativ tillväxt och det inledande skedet av sporulering septumbildning har använt syrepermeabla imaging kammare, eller agarosen stödda tillväxten av Streptomyces coelicolor på mikroskop scenen 11-15. Dessa metoder är emellertid begränsad av ett antal faktorer. Vissa system tillåter bara kortsiktiga avbildning av celltillväxt end fluorescerande proteiner före celler lider av otillräcklig syretillförsel eller växa ut ur fokalplanet på grund av den tredimensionella mönster av hyfal utveckling. I de fall där långsiktig avbildning är möjlig, odla cellerna på agaros pads gränser experimentellt flexibilitet eftersom cellerna inte kan utsättas för alternativa tillväxt- eller stressbetingelser, och bakgrundsfluorescens från mediet i agarosen dynor begränsar allvarligt förmågan att övervaka svagare fluorescerande signaler.
Här beskriver vi ett protokoll för levande cell imaging av kompletta Streptomyces livscykel med utmärkt precision och känslighet. Genom att odla Streptomyces i ett mikroflödessystem enhet som är ansluten till en fluorescens widefield mikroskop (Figur 2), kan vi nu att övervaka groning, vegetativ tillväxt och sporbildning septumbildning under en tidsperiod på upp till 30 timmar. Detta underlättas i hög grad genom användning av de nya modellorganism Streptomyces </em> venezuelae eftersom det sporulates till nära färdig i nedsänkt kultur och därmed övervinner begränsningen av den klassiska modellen arten S. coelicolor, som sporulates bara på fasta medier 16-20. För att hjälpa visualisera vegetativ tillväxt och sporulering, vi samar uttrycker fluorescensmärkta versioner av cellens polaritet markören DivIVA och nyckeln celldelning protein FtsZ.
Vi använder en kommersiellt tillgänglig mikroflödessystem enhet som har använts med framgång för mykobakterier, Escherichia coli, Corynebacterium glutamicum, Bacillus subtilis och jäst 21-25. Systemet fångar celler i en enda fokalplan och tillåter kontroll av kontinuerlig perfusion av odlingsmedium från olika reservoarer. I den detaljerade protokollet tar vi fördel av den här funktionen för att exponera S. venezuelae vegetativt mycel till en närings nedväxling för att främja sporbildning.
Protokollet debeskrivs är för levande cell imaging av hela Streptomyces livscykel, men alternativa medier villkor eller mikroskop inställningar kan väljas om specifika utvecklingsstadier är av särskilt intresse.
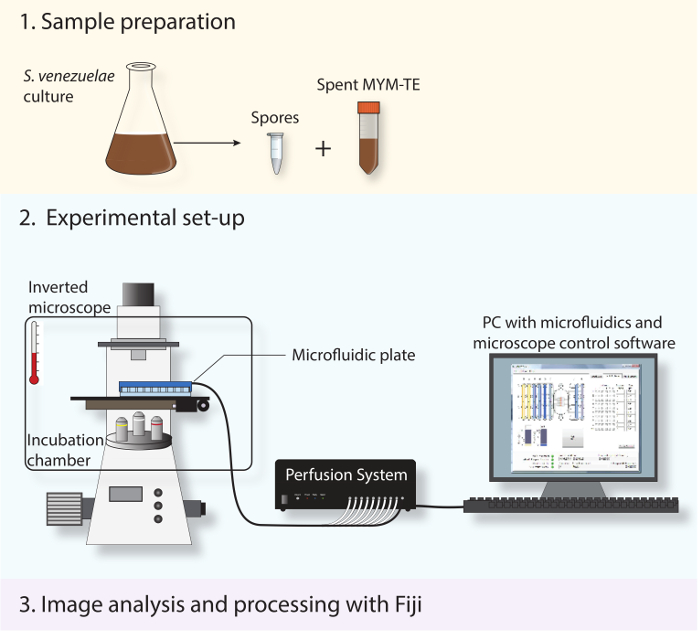
Figur 2: Schematisk skildrar experimentella arbetsflödet. De tre huvudsteg som beskrivs i protokollet visas. Först är sporer och använt medium framställt från en stationär fas kultur. För det andra är de färska sporer laddas in i en mikroflödessystem och S. venezuelae avbildas hela sin utvecklings livscykel med hjälp av en helt automatiserad inverterat mikroskop med en inkubationstid kammare för att upprätthålla en optimal tillväxttemperatur. För det tredje är det tidsförlopp serie erhålls analyseras och bearbetas med hjälp av öppen källkod Fiji.
Protocol
Representative Results
Discussion
Time-lapse mikroskopi av Streptomyces livscykel har varit en teknisk utmaning i det förflutna. Här presenterar vi en robust protokoll för att utföra levande cell imaging av hela livscykel med hjälp av fluorescerande proteinfusioner till cell polaritet markören DivIVA och celldelning proteinet FtsZ att hjälpa visualisera och spåra progression genom utvecklingsprogrammet (Figur 2).
Centralt för denna metod är odling av S. venezuelae i en mikroflödessystem enhet som tillåter konstant medel perfusion och utbyte av normalt odlingsmedium (MYM-TE) med förbrukat medium (bringade MYM-TE). Bytet av odlingsbetingelser är viktigt att det beskrivna protokollet eftersom närings nedväxling som brunnar som någon ännu ej identifierade extracellulära signaler (t.ex. kvorumavkänning signaler) i det använda odlingsmediet, främja sporbildning, medan en kontinuerlig tillförsel av näringsrikt mediet stimulerar vegetativt growth med knappast någon sporbildning (data ej visade). Således, odling av celler i denna mikroflödessystem är överlägset odla celler på agaros eftersom det ger mer experimentell flexibilitet och möjliggör långsiktig uppföljning av förändringar i bakterietillväxt som svar på förändrade odlingsbetingelser. Även om mikroflödesplattorna är konstruerade för engångsbruk, flödes kanaler som inte har ympats med celler kan användas i efterföljande experiment. Vi rekommenderar att du använder alla kanaler i en mikroflödesplatta inom en vecka, eftersom vi har haft problem att täta grenröret plattor som har öppnats under längre tider.
När du ställer in ett experiment, fann vi att nylagad sporer grott inom två timmar av lastas in i flödeskammaren, medan sporer som härrör från en frusen glycerol lager krävs minst 6 timmar före köns rör framkom (data visas ej). Denna fördröjning av groning förlänger längden av experimentet och kan störautrustningens tillgänglighet och experimentella förhållanden. Det är också viktigt att börja experimentera med perfusion av MYM-TE under minst tre timmar för att ge tillräckligt med näringsämnen för utveckling och utväxt av köns rör. Perfusion med MYM-TE kan förlängas utöver den inledande tre timmar om försöket har utformats för att studera vegetativ tillväxt. Medan sporer ge det självklara valet av utgångsmaterial, kan kort hyfernas fragment också laddas in i mikroflödesplattan. Men är signifikant lägre laddningseffektivitet vid användning av klippt mycel och kräver ofta flera last körningar som kan utgöra en begränsning vid prövningen icke-sporbildande mutanter. Oavsett vilken typ av inokulum används är det viktigt att inte överbelasta odlingskammare med sporer eller hyfernas fragment som detta kommer att leda till en snabb överbeläggning som kan störa media diffusion och komplicera bildanalys.
Det är viktigt att planera byggandet av rapportörproteiner carefully för användning i fluorescens tidsförlopp avbildning i Streptomyces. Linkerlängd mellan målproteinet och den fluorescerande reporter och valet av N- eller C-terminala fusioner kan vara kritisk. Dessutom optimala experimentella förhållanden, inklusive avbildning frekvens och exponeringstid, måste fastställas i förväg för varje fluorescerande-märkta proteinet. S. venezuelae hyfer uppvisar auto fluorescens i grön / gul-kanal som kan bli problematisk när avbildning av ett fluorescensmärkt protein som uttrycks vid låga nivåer. Dessutom bör det noteras att, under groning och den initiala vegetativa tillväxtfasen, S. venezuelae är särskilt känslig för kortvågigt ljus (t.ex. vid användning av proteinfusioner till GFP).
Trots de kontinuerliga framsteg inom avbildningstekniker och fluorescerande reportersystem för levande cell imaging av bakterier, de flesta av de programpaket (t.ex. MicrobeTracker, Schnitzelcalnar, CellProfiler) för efterföljande automatisk behandling av dessa typer av dataset stöder inte analys av bilder som härrör från trådbildande bakterier med en flercellig livsstil 27-29. Således finns det ett behov av att utveckla en lämplig algoritm för kvantitativ high-throughput-analys av bilddata från Streptomyces och andra filamentösa bakterier.
Sammanfattningsvis, det arbete som beskrivs här visar den enorma potentialen hos S. venezuelae som en ny modell utvecklingssystem för släktet, på grund av dess förmåga att sporulera i vätska. Den mikroflödesodlande anordning är enkel att använda även för oerfarna användare. Det ger en utmärkt plattform för att studera cellbiologiska processer är centrala för Streptomyces livscykel, inklusive dynamisk protein lokalisering, polariserad tillväxt och morfologisk differentiering av en flercellig mycel i kedjor av unigenomic sporer. Dessutom denna experimentella set up tillhandahåller också en lockande utgångspunkt för att undersöka händelser i utvecklingen som kräver alternerande odlingsbetingelser, eller användning av fluorescerande färgämnen såsom fluorescerande D-aminosyror för att övervaka peptidoglykansyntesen eller propidiumjodid och 4 ', 6-diamidino-2-fenylindol (DAPI) för att visualisera kromosomorganisation 30,31.
Disclosures
The authors have nothing to disclose.
Acknowledgements
The authors thank Grant Calder for technical assistance with the microscope, Matt Bush for comments on the protocol, and the John Innes Centre for purchase of the Zeiss widefield microscope. This work was funded by BBSRC grant BB/I002197/1 (to M.J.B), by BBSRC Institute Strategic Programme Grant BB/J004561/1 to the John Innes Centre, by Swedish Research Council grant 621-2010-4463 (to K.F.), and by a Leopoldina Postdoctoral Fellowship (to S.S.).
Materials
| B04A CellAsic ONIX plate for bacteria cells | Merck-Millipore | B04A-03-5PK | Microfluidic culture plates |
| CellAsic ONIX Microfluidic Perfusion System and ONIX FG (version 5.0.2) | Merck-Millipore | EV-262 | The latest ONIX vesion (July 2015) and instructions on how to use the programme can be found here: http://www.merckmillipore.com |
| Axio Observer.Z1 Microscope | Zeiss | 431007-9902-000 | Fully automated and motorized inverted widefield microscope |
| Incubator XL multi S1 with Temperature Module S1 and Heating unit XL S2 | Zeiss | 411857-9061-000 | Environmental chamber surrounding the microscope |
| Plan-Apochromat 100x/1.46 Oil DIC objective | Zeiss | 420792-9800-000 | |
| Ocra FLASH 4 V2 | Hamamatsu Photonics K.K. | C11440-22CU | |
| Illuminator HXP 120V | Zeiss | 423013-9010-000 | |
| FL Filter Set 46 HE YFP shift free | Zeiss | 489046-9901-000 | Fluorescent filter set, excitation 500/25 nm, emission 535/30 nm |
| FL Filter Set 63 HE RFP shift free | Zeiss | 489063-0000-000 | Fluorescent filter set, excitation 572/25 nm, emission 629/30 nm |
| Mounting frame K-M for multiwell plates | Zeiss | 000000-1272-644 | Stage holder for microfluidic plate |
| ZEN pro 2012 | Zeiss | 410135-1002-120 | Microscope control software |
| ZEN Module Time Lapse | Zeiss | 410136-1031-110 | Software module to set up time-lapse microscopy experiments |
| ZEN Module Tiles/Positions | Zeiss | 410136-1025-110 | Software module to save specific stage positions (xzy) |
| Fiji | open-source software package | http://fiji.sc/Fiji | Generation of time-lapse movies |
| Maltose-Yeast Exctract-Malt Extract (MYM) 4 g Maltose 4 g Yeast extract 10 g Malt extract add 1 L H2O using 50 % tap water and 50 % reverse osmosis water and supplement with 200 ml of R2 trace element solution per 100 ml after autoclaving |
Sporulation medium used to culture S. venezuelae SV60 | ||
| R2 Trace element solution (TE) 8 mg ZnCl2 40 mg FeCl3-6H2O 2 mg CuCl2-2H2O 2 mg MnCl2-4H2O 2 mg Na2B4O7-10H2O 2 mg (NH4)6Mo7O24-4H2O add 200 ml H2O Autoclave and store at 4 oC |
Add 0.002 volumes to MYM | ||
| PBS (phosphate buffered saline) | Sigma | P4417-100TAB | Used to refill inlet wells of unused lanes in B04A plates in order to prepare plate for short-term storage. |
| 0.22 µm syringe filters | Satorius stedim | 16532-K | Preparation of spent MYM-TE |
| SV60 | John Innes Centre strain collection | S. venezuelae strain expressing divIVA-mcherry and ftsZ-ypet |
References
- Bush, M. J., Tschowri, N., Schlimpert, S., Flärdh, K., Buttner, M. J. c-di-GMP signalling and the regulation of developmental transitions in streptomycetes. Nat. Rev. Microbiol. 13, 749-760 (2015).
- Jakimowicz, D., van Wezel, G. P. Cell division and DNA segregation in Streptomyces.: how to build a septum in the middle of nowhere?. Mol. Microbiol. 85, 393-404 (2012).
- McCormick, J. R., Flärdh, K. Signals and regulators that govern Streptomyces development. FEMS Microbiol. Rev. 36, 206-231 (2012).
- Flärdh, K., Richards, D. M., Hempel, A. M., Howard, M., Buttner, M. J. Regulation of apical growth and hyphal branching in Streptomyces. Curr. Opin. Microbiol. 15, 737-743 (2012).
- Hempel, A. M., et al. The Ser/Thr protein kinase AfsK regulates polar growth and hyphal branching in the filamentous bacteria Streptomyces. Proc. Natl. Acad. Sci. U S A. 109, E2371-E2379 (2012).
- Fuchino, K., et al. Dynamic gradients of an intermediate filament-like cytoskeleton are recruited by a polarity landmark during apical growth. Proc. Natl. Acad. Sci. U S A. 110, E1889-E1897 (2013).
- Holmes, N. A., et al. Coiled-coil protein Scy is a key component of a multiprotein assembly controlling polarized growth in Streptomyces. Proc. Natl. Acad. Sci. U S A. 110, E397-E406 (2013).
- McCormick, J. R., Su, E. P., Driks, A., Losick, R. Growth and viability of Streptomyces coelicolor. mutant for the cell division gene ftsZ. Mol. Microbiol. 14, 243-254 (1994).
- Margolin, W. FtsZ and the division of prokaryotic cells and organelles. Nat. Rev. Mol. Cell Biol. 6, 862-871 (2005).
- Grantcharova, N., Lustig, U., Flärdh, K. Dynamics of FtsZ assembly during sporulation in Streptomyces coelicolor A3(2). J. Bacteriol. 187, 3227-3237 (2005).
- Richards, D. M., Hempel, A. M., Flärdh, K., Buttner, M. J., Howard, M. Mechanistic basis of branch-site selection in filamentous bacteria. PLoS Comput. Biol. 8, e1002423 (2012).
- Hempel, A. M., Wang, S. B., Letek, M., Gil, J. A., Flärdh, K. Assemblies of DivIVA mark sites for hyphal branching and can establish new zones of cell wall growth in Streptomyces coelicolor. J. Bacteriol. 190, 7579-7583 (2008).
- Wolanski, M., et al. Replisome trafficking in growing vegetative hyphae of Streptomyces coelicolor A3(2). J. Bacteriol. 193, 1273-1275 (2011).
- Jyothikumar, V., Tilley, E. J., Wali, R., Herron, P. R. Time-lapse microscopy of Streptomyces coelicolor.growth and sporulation. Appl. Environ. Microbiol. 74, 6774-6781 (2008).
- Willemse, J., Borst, J. W., de Waal, E., Bisseling, T., van Wezel, G. P. Positive control of cell division: FtsZ is recruited by SsgB during sporulation of Streptomyces. Genes. Dev. 25, 89-99 (2011).
- Glazebrook, M. A., Doull, J. L., Stuttard, C., Vining, L. C. Sporulation of Streptomyces venezuelae. in submerged cultures. J. Gen. Microbiol. 136, 581-588 (1990).
- Bibb, M. J., Domonkos, A., Chandra, G., Buttner, M. J. Expression of the chaplin and rodlin hydrophobic sheath proteins in Streptomyces venezuelae.is controlled by sigma(BldN) and a cognate anti-sigma factor, RsbN. Mol. Microbiol. 84, 1033-1049 (2012).
- Al-Bassam, M. M., Bibb, M. J., Bush, M. J., Chandra, G., Buttner, M. J. Response regulator heterodimer formation controls a key stage in Streptomyces development. PLoS Genet. 10, e1004554 (2014).
- Bush, M. J., Bibb, M. J., Chandra, G., Findlay, K. C., Buttner, M. J. Genes required for aerial growth, cell division, and chromosome segregation are targets of WhiA before sporulation in Streptomyces venezuelae. MBio. 4, e00684-e00613 (2013).
- Tschowri, N., et al. Tetrameric c-di-GMP mediates effective transcription factor dimerization to control Streptomyces development. Cell. 158, 1136-1147 (2014).
- Mirouze, N., Ferret, C., Yao, Z., Chastanet, A., Carballido-Lòpez, R. MreB-Dependent Inhibition of Cell Elongation during the Escape from Competence in Bacillus subtilis. PLoS Genet. 11, e1005299 (2015).
- Zopf, C. J., Maheshri, N. Acquiring fluorescence time-lapse movies of budding yeast and analyzing single-cell dynamics using GRAFTS. J. Vis. Exp. (e50456), (2013).
- Meniche, X., et al. Subpolar addition of new cell wall is directed by DivIVA in mycobacteria. Proc. Natl. Acad. Sci. U S A. 111, E3243-E3251 (2014).
- Donovan, C., Schauss, A., Kramer, R., Bramkamp, M. Chromosome segregation impacts on cell growth and division site selection in Corynebacterium glutamicum. PLoS One. 8, e55078 (2013).
- Rojas, E., Theriot, J. A., Huang, K. C. Response of Escherichia coli. growth rate to osmotic shock. Proc. Natl. Acad. Sci. U S A. 111, 7807-7812 (2014).
- Flärdh, K. Essential role of DivIVA in polar growth and morphogenesis in Streptomyces coelicolor A3(2). Mol. Microbiol. 49, 1523-1536 (2003).
- Sliusarenko, O., Heinritz, J., Emonet, T., Jacobs-Wagner, C. High-throughput subpixel precision analysis of bacterial morphogenesis and intracellular spatio-temporal dynamics. Mol. Microbiol. 80, 612-627 (2011).
- Young, J. W., et al. Measuring single-cell gene expression dynamics in bacteria using fluorescence time-lapse microscopy. Nat. Protoc. 7, 80-88 (2012).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7, R100 (2006).
- Kuru, E., Tekkam, S., Hall, E., Brun, Y. V., Van Nieuwenhze, M. S. Synthesis of fluorescent D-amino acids and their use for probing peptidoglycan synthesis and bacterial growth in situ. Nat. Protoc. 10, 33-52 (2015).
- Szafran, M., et al. Topoisomerase I (TopA) is recruited to ParB complexes and is required for proper chromosome organization during Streptomyces coelicolor. sporulation. J. Bacteriol. 195, 4445-4455 (2013).

