GeNemo का उपयोग कर Epigenomic डेटा के पैटर्न आधारित खोज
Summary
डीएनए अनुक्रम डेटा के विपरीत, epigenomic डेटा आसानी से पाठ आधारित खोजों के अधीन नहीं हैं । यहां प्रस्तुत प्रक्रियाओं GeNemo, एक वेब आधारित bioinformatics उपकरण के एक उंनत संस्करण का उपयोग करने के लिए कर रहे हैं, epigenomic के साथ डीएनए तत्वों के विश्वकोश सहित उपलब्ध ऑनलाइन डाटाबेस की तुलना डेटा में समानता के लिए पैटर्न आधारित खोजों आचरण उपयोगकर्ता का डेटा ।
Abstract
जीनोमिक या आरएनए अनुक्रमण डेटा के लिए मजबूत पाठ आधारित खोज उपकरणों के साथ तुलना में, epigenomic और अन्य कार्यात्मक जीनोमिक डेटा के पैटर्न आधारित खोजों के लिए वर्तमान तरीके बहुत सीमित हैं । GeNemo पहली ऑनलाइन खोज उपकरण है जो इस लक्ष्य को पूरा करता है । उपयोगकर्ताओं को ब्राउज़र एक्सटेंसिबल डेटा (बिस्तर), चोटियों, और bigWig स्वरूपों में अपने कार्यात्मक जीनोमिक डेटा इनपुट, और किसी भी तीन स्वरूपों में डेटा के लिए खोज कर सकते हैं । उपयोगकर्ता निर्दिष्ट कर सकते है कि किस प्रकार के डेटासेट के विरुद्ध खोज करने के लिए, विभिन्न epigenomic चिह्नों का प्रतिनिधित्व करने वाले डीएनए तत्वों के विश्वकोश (सांकेतिक शब्दों में), transcriptional फ़ैक्टर बाइंडिंग साइट्स, और क्रोमेटिन के साथ ऑनलाइन डेटासेट के विभिंन प्रकारों में से चुनना विशिष्ट कक्ष प्रकारों, और विकासात्मक अवस्थाओं या प्रजातियों (माउस या मानव) में hypersensitivities या accessibilities । GeNemo इनपुट डेटा, जो ब्राउज़र में देखा जा सकता है और साथ ही बिस्तर फ़ाइल प्रारूप में डाउनलोड के लिए पैटर्न मिलान के साथ जीनोमिक क्षेत्रों की एक सूची देता है । उंनत GeNemo चित्रमय प्रदर्शन में सुधार हुआ है, और अधिक मजबूत इंटरफेस है, और अब कैलिफोर्निया, सांता क्रूज़ (UCSC) जीनोम ब्राउज़र के विश्वविद्यालय में परिवर्तन के कारण त्रुटियों के लिए प्रवण है । सामांय समस्याओं के लिए समस्या निवारण चरण चर्चा है । कार्यात्मक जीनोमिक डेटा की मात्रा के रूप में तेजी से विस्तार हो रहा है, वहां एक महत्वपूर्ण को विकसित करने और डेटा विश्लेषण और व्याख्या के लिए GeNemo जैसे नए bioinformatic उपकरण परिष्कृत की जरूरत है ।
Introduction
हाल ही में तकनीकी प्रगति epigenomic या कार्यात्मक जीनोमिक डेटा खजाने, जो प्रासंगिक विश्लेषणात्मक उपकरण के विकास से आगे निकल गया है जैविक अंतर्दृष्टि निकालने के एक तेजी से विस्तार के लिए अनुमति दी है । एक महत्वपूर्ण तरीका है epigenomic डेटा का विश्लेषण करने के लिए उपयोगकर्ता खोज डेटा खजाने के खिलाफ डेटा उत्पंन और विशेष रूप से डीएनए तत्वों के विश्वकोश से उन (सांकेतिक शब्दों में बदलना) पैटर्न है कि नए ज्ञान के लिए नेतृत्व कर सकते है मिलान के लिए1 परियोजनाओं है । उदाहरण के लिए, जीनोम भर में परिभाषित loci पर दो अलग epigenomic निशान के पैटर्न में समानता की पहचान क्रोमेटिन अनुरूप और transcriptional विनियमन2 पर विभिंन आणविक खिलाड़ियों द्वारा समंवित कार्रवाई का संकेत हो सकता है ,3,4.
पारंपरिक पाठ आधारित खोज इंजन इस संबंध में अप्रभावी क्योंकि, डीएनए अनुक्रम के विपरीत, epigenomic डेटा मुख्यतः तीव्रता या कार्यात्मक जीनोमिक क्षेत्रों के स्वरूप में मौजूद हैं । GeNemo, जीन निमो के लिए खड़े (निमो खोजने में के रूप में), इस unmet पैटर्न आधारित खोजों5का उपयोग करने की आवश्यकता को संबोधित करने के लिए विकसित किया गया था । इसके एल्गोरिथ्म एक मार्कोव चेन मोंटे कार्लो अधिकतम प्रक्रिया का इस्तेमाल करता है5. उपयोगकर्ताओं को अपने डेटा या एक खजाने से डाउनलोड डेटासेट ले और पैटर्न में समानता की पहचान करने के लिए ऑनलाइन epigenomic डेटा की एक सरणी खोज ।
GeNemo के वर्तमान संस्करण एक अद्यतन प्रदर्शन किया है, कैलिफोर्निया विश्वविद्यालय के साथ और अधिक मजबूत इंटरफेस, सांता क्रूज़ (UCSC) जीनोम ब्राउज़र6, और कम बाद में परिवर्तन की वजह से मुद्दों के लिए अतिसंवेदनशील है । विशेष रूप से, जबकि है GeNemo परिणाम पृष्ठ पर UCSC जीनोम ब्राउज़र इंटरफेस के आधार पर इस्तेमाल किया, GeNemo के वर्तमान संस्करण अपने परिणाम पृष्ठ का समर्थन करता है और फलस्वरूप अब कोई प्रतिकूल UCSC जीनोम ब्राउज़र को संरचनात्मक परिवर्तन से प्रभावित है । GeNemo किसी भी जीनोमिक संकेत का उपयोग कर सकते हैं, जिसमें प्रोटीन-बाइंडिंग, citrullinated संशोधन, क्रोमेटिन पहुंच, टोपोलॉजिकल डोमेन, इत्यादि, जैसे बड़े colocalized से ज्ञात डेटा सेट के बीच consortia/समान सेगमेंट ढूंढने के लिए क्वेरी । इसलिए, यह ब्याज और बड़े पैमाने पर जीनोमिक परियोजनाओं में उत्पंन ज्ञात डेटा के विभिंन epigenomic डेटा के बीच संबंधों का अध्ययन करने के लिए एक महत्वपूर्ण उपकरण है ।
Protocol
नोट: प्रोटोकॉल कहीं भी ठहर सकता है ।
1. मूल सेटअप एक बिस्तर, चोटियों प्रारूप, या BigWig 7 फ़ाइल के जीनोम में इनपुट होने के लिए डेटा युक्त । फ़ाइल का एक्सटेंशन नाम होना चाहिए & #34; bed & #34;, & #34; broadpeaks & #34; & #34; narrowpeaks & #34;, या & #34; bigWig & #34; क्रमश. & #8203; नोट: इन प्रकार की फ़ाइलों के ज़िप्ड संस्करण भी काम करेंगे । genemo.org जाने के लिए एक इंटरनेट ब्राउज़र का उपयोग करें । सबसे आम इंटरनेट ब्राउज़र चलाने में सक्षम किसी भी ऑपरेटिंग सिस्टम GeNemo. का उपयोग करने में सक्षम होना चाहिए ड्रॉपडाउन मेनू का उपयोग कर के खिलाफ खोज करने के लिए जो प्रजातियों चुनें । वर्तमान में उपलब्ध प्रजातियों में मानव और माउस शामिल हैं । अपलोड उपयोगकर्ता एक यूआरएल या एक प्रत्यक्ष अपलोड का उपयोग कर फ़ाइल । BigWig फ़ाइलें केवल url अपलोड विधि के साथ काम करते हैं । बिस्तर और चोटियों प्रारूप फ़ाइलें दोनों विधियों के साथ काम करते हैं (अब के रूप में मुख्य डेटा के रूप में चढ़ाव फ़ाइलें अपलोड नहीं किया जा सकता).
2. वैकल्पिक सेटअप खोज किया जाता है जब ई-मेल द्वारा खोज परिणाम प्राप्त करने के लिए संबंधित बॉक्स में एक ईमेल पता प्रदान करें । & #8203; नोट: जब जीनोम का एक बड़ा हिस्सा खोज और/या पटरियों की एक बड़ी संख्या के खिलाफ (नीचे देखें), यह अनुशंसित है कि उपयोगकर्ता उसके ईमेल प्रदान करता है, क्योंकि खोज एक लंबा समय लग सकता है । उदाहरण के लिए, एक १०० megabase खोज 15 एस के आसपास लेता है । खोज के पूर्ण होने पर प्रदान किए गए ई-मेल पते पर एक लिंक भेजा जाएगा । लिंक एक खोज के पूरा होने के बाद 7 दिनों में समाप्त हो जाएगा । प्रदान एक bigwig फ़ाइल या चढ़ाव प्रदर्शन फ़ाइल किसी url से हो सकता है । यह प्रदर्शन फ़ाइल परिणामों को प्रभावित नहीं करेगी; यह केवल परिणामों के साथ दिखाया जाएगा. संबंधित बॉक्स में (गुणसूत्र और आधार जोड़ी पदों सहित) एक खोज रेंज निर्दिष्ट करें । गुणसूत्र की सूची, आधार युग्म, और अंत आधार जोड़ी शुरू करते हैं । उपयोग & #39; chrN & #39; गुणसूत्र प्रारूप के लिए, जहां & #39; N & #39; गुणसूत्र संख्या/पत्र (1, 2, & #8230; X, या Y) है । आधार जोड़े के लिए, बस संख्या में टाइप करें । सभी तीन प्रविष्टियों के बीच रिक्त स्थान शामिल हैं, या एक बृहदांत्र (गुणसूत्र संख्या और पहली आधार जोड़ी के बीच:), और/ उदाहरणार्थ: chr1:1000000-2000000, chr1 १०००००० २००००००, chr1 1000000-2000000, chr1:1000000 २००००००. नोट: चरण २.१-२.३ वैकल्पिक है.
3. डाटा चयन 4. खोज और परिणाम डेटा चयन के बाद & #34; Search & #34; बटन क्लिक करें । खोज में कुछ समय लग सकता है । खोज पूरी होने के बाद, उपयोगकर्ताओं को परिणाम पृष्ठ में विभिंन बक्से दिखाई देंगे । प्रत्येक बॉक्स जीनोम के एक खंड का प्रतिनिधित्व करता है, जहां उपयोगकर्ता & #39; s डेटा फ़ाइल में एक या अधिक ट्रैक के साथ एक निकट मिलान प्रतिमान है उपयोगकर्ता ने क्वेरी की है । यदि कोई बॉक्स दृश्यमान नहीं हैं, तो अधिक प्रकार के ट्रैक खोजने या खोज श्रेणी को समान इनपुट फ़ाइल से बड़ा करने का प्रयास करें । सब कुछ के बिना यह करने के लिए एक आसान तरीका क्लिक कर रहा है & #34; & #9776; & #34; बटन लोगो के बगल में । यह एक साइडबार है कि उपयोगकर्ता खोज को संशोधित करने की अनुमति देता है खुल जाएगा । परिणाम पृष्ठ के तल पर & #34;D ownload bed file & #34; बटन पर क्लिक करके एक बेड फाइल के रूप में निर्यात किया जा सकता है । परिणामों को विज़ुअलाइज़ करने के लिए प्रत्येक बॉक्स के ऊपर दाईं ओर विज़ुअलाइज़ करें बटन पर क्लिक करे. दाईं तरफ विज़ुअलाइज़ेशन पैनल में, एकाधिक चीज़ें डेटा सहित प्रदर्शित की जाती हैं, जो उपयोगकर्ता इनपुट फ़ाइल को शामिल करती है, प्रदर्शन फ़ाइल अगर एक से बाहर किया गया था, पटरियों मिलान, और कुछ डिफ़ॉल्ट पटरियों । परिणाम से, उपयोगकर्ता ज्ञात एन्कोडेड dataset के विरुद्ध आगे की जाँच के लिए दिए गए डेटासेट की तुलना कर सकते हैं । उपयोगकर्ता क्वेरी परिणामों के संदर्भ को देखने के लिए UCSC जीन का उल्लेख भी कर सकते हैं । यदि एकाधिक सेल लाइनों/ऊतकों से पटरियों का चयन किया जाता है, तो उपयोगकर्ता दिए गए डेटासेट और एनकोडिंग डेटासेट के बीच समानता की ऊतक विशिष्टता के बारे में अंतर्दृष्टि प्राप्त करने के लिए ऐसे परिणामों का उपयोग कर सकते हैं. परिणाम पृष्ठ पर , उपयोगकर्ता किसी भी पटरियों पर खींचें के लिए ऊपर या जीनोम के बहाव को स्थानांतरित कर सकते हैं; जब माउस कर्सर निर्देशांक पर है, तो उपयोगकर्ता माउस व्हील का उपयोग करें और/या में और बाहर ज़ूम कर सकते हैं । 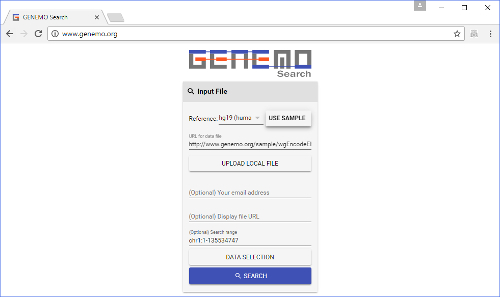 चित्रा 1 : GeNemo & #39; एस के आवश्यक क्षेत्रों के साथ सामने पृष्ठ । & #160; एक उपयोगकर्ता को प्रजातियों, खोज फ़ाइल और खोज रेंज इनपुट की जरूरत है, और पटरियों का चयन करें वह के खिलाफ खोज करने के लिए चाहता है । ईमेल पता और प्रदर्शन फ़ाइल वैकल्पिक हैं । & #160; इस फिगर का बड़ा वर्जन देखने के लिए यहां क्लिक करें ।
चित्रा 1 : GeNemo & #39; एस के आवश्यक क्षेत्रों के साथ सामने पृष्ठ । & #160; एक उपयोगकर्ता को प्रजातियों, खोज फ़ाइल और खोज रेंज इनपुट की जरूरत है, और पटरियों का चयन करें वह के खिलाफ खोज करने के लिए चाहता है । ईमेल पता और प्रदर्शन फ़ाइल वैकल्पिक हैं । & #160; इस फिगर का बड़ा वर्जन देखने के लिए यहां क्लिक करें । 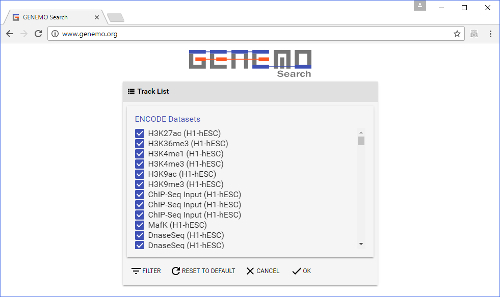 चित्रा 2 : ट्रैक चयन खिड़की. & #160; यह सामने पृष्ठ पर & #34;D ata चयन & #34; बटन पर क्लिक करके लाया जाता है । यहाँ, उपयोगकर्ता इनपुट फ़ाइल के विरुद्ध खोज करने के लिए ट्रैक का चयन करें । कुछ ट्रैक्स पहले से डिफॉल्ट रूप से चयनित हैं । & #160; इस फिगर का बड़ा वर्जन देखने के लिए यहां क्लिक करें । डेटा चयन बटन क्लिक करने के बाद, ( यानी , क्वेरी में जोड़ने के लिए) के विरुद्ध खोज करने के लिए ट्रैक के किस प्रकार का चयन करें । ट्रैक संग्रह दुनिया भर में प्रयोगशालाओं से कई विभिन्न डेटासेट शामिल हैं. पटरियों की सूची के रूप में काफी लंबा है, उपयोगकर्ताओं को ट्रैक चयन की सुविधा के लिए फ़िल्टर बटन (शीर्ष पर) का उपयोग करना चाहते हो सकता है । पटरियों प्रयोग, ऊतक, सेल लाइन और/ द्वारा फ़िल्टर किया जा सकता है ट्रैक चयन निष्पादित करने में मदद करने के लिए नीचे दिए गए पांच बटन हैं: सभी का चयन करें, कोई नहीं का चयन करें, जोड़ें, फ़िल्टर, बाहर । का चयन सभी & #34; र & #34; चय क कोई भी & #34; य है । & #34; Add & #34; बटन वर्तमान में चयनित ट्रैक्स को क्वेरी में जोड़ता है । यह तर्क गेट & #34 के रूप में कार्य करता है; या & #34;. ध्यान दें कि उपरोक्त फ़िल्टर ( उदा. , कुछ प्रयोग, ऊतक, सेल लाइन या लैब्स) का चयन करने से खोज क्वेरी में स्वचालित रूप से संगत ट्रैक नहीं जुड़ते हैं । उपयोगकर्ताओं को पहले पटरियों ( जैसे , मस्तिष्क, जिगर के नीचे ऊतक) का चयन करना होगा, और फिर उन्हें क्वेरी में जोड़ने के लिए & #34; Add & #34; बटन पर क्लिक करें. ट्रैक का चयन करते समय, नोट करें कि केवल फ़िल्टर विंडो में खोले गए टैब में निर्दिष्ट फ़िल्टर्स खोज क्वेरी पर लागू किए जाएंगे. अंय टैब्स पर चयनों को फ़िल्टर विंडो में सहेजा जाएगा, लेकिन खोज क्वेरी पर लागू नहीं होगा । & #34; फ़िल्टर & #34; बटन केवल वर्तमान में क्वेरी में फ़िल्टर विंडो में चयनित ट्रैक के प्रकार बनाए रखता है और अन्य सभी प्रकार की ट्रैक्स को निकालता है. यह तर्क गेट & #34 के रूप में कार्य करता है; और & #34;. मूलतः, & #34; फिल् & #34; पटरियों की दो श्रेणियों ( जैसे , कुछ प्रयोगशालाओं के साथ कुछ ऊतकों) के बीच बातचीत के चयन की अनुमति देता है. ध्यान दें कि & #34; फ़िल्टर & #34; यदि वे पहले से ही क्वेरी में नहीं हैं, तो चयनित प्रकार के ट्रैक्स को क्वेरी में न जोड़ें. & #34; छोडी & #34; बटन सभी प्रकार की ट्रैक्स निकालता है जो वर्तमान में क्वेरी से फ़िल्टर विंडो में चयनित हैं । यह तर्क gate & #34 के रूप में कार्य करता है; न & #34;, विरोध में & #34; फ़िल्टर & #34; फ़ंक्शन. नु, त & #34; छोडी & #34; कोई भी ट्रैक्स वर्तमान में फ़िल्टर विंडो में क्वेरी करने के लिए चयनित नहीं जोड़ नहीं है ।
चित्रा 2 : ट्रैक चयन खिड़की. & #160; यह सामने पृष्ठ पर & #34;D ata चयन & #34; बटन पर क्लिक करके लाया जाता है । यहाँ, उपयोगकर्ता इनपुट फ़ाइल के विरुद्ध खोज करने के लिए ट्रैक का चयन करें । कुछ ट्रैक्स पहले से डिफॉल्ट रूप से चयनित हैं । & #160; इस फिगर का बड़ा वर्जन देखने के लिए यहां क्लिक करें । डेटा चयन बटन क्लिक करने के बाद, ( यानी , क्वेरी में जोड़ने के लिए) के विरुद्ध खोज करने के लिए ट्रैक के किस प्रकार का चयन करें । ट्रैक संग्रह दुनिया भर में प्रयोगशालाओं से कई विभिन्न डेटासेट शामिल हैं. पटरियों की सूची के रूप में काफी लंबा है, उपयोगकर्ताओं को ट्रैक चयन की सुविधा के लिए फ़िल्टर बटन (शीर्ष पर) का उपयोग करना चाहते हो सकता है । पटरियों प्रयोग, ऊतक, सेल लाइन और/ द्वारा फ़िल्टर किया जा सकता है ट्रैक चयन निष्पादित करने में मदद करने के लिए नीचे दिए गए पांच बटन हैं: सभी का चयन करें, कोई नहीं का चयन करें, जोड़ें, फ़िल्टर, बाहर । का चयन सभी & #34; र & #34; चय क कोई भी & #34; य है । & #34; Add & #34; बटन वर्तमान में चयनित ट्रैक्स को क्वेरी में जोड़ता है । यह तर्क गेट & #34 के रूप में कार्य करता है; या & #34;. ध्यान दें कि उपरोक्त फ़िल्टर ( उदा. , कुछ प्रयोग, ऊतक, सेल लाइन या लैब्स) का चयन करने से खोज क्वेरी में स्वचालित रूप से संगत ट्रैक नहीं जुड़ते हैं । उपयोगकर्ताओं को पहले पटरियों ( जैसे , मस्तिष्क, जिगर के नीचे ऊतक) का चयन करना होगा, और फिर उन्हें क्वेरी में जोड़ने के लिए & #34; Add & #34; बटन पर क्लिक करें. ट्रैक का चयन करते समय, नोट करें कि केवल फ़िल्टर विंडो में खोले गए टैब में निर्दिष्ट फ़िल्टर्स खोज क्वेरी पर लागू किए जाएंगे. अंय टैब्स पर चयनों को फ़िल्टर विंडो में सहेजा जाएगा, लेकिन खोज क्वेरी पर लागू नहीं होगा । & #34; फ़िल्टर & #34; बटन केवल वर्तमान में क्वेरी में फ़िल्टर विंडो में चयनित ट्रैक के प्रकार बनाए रखता है और अन्य सभी प्रकार की ट्रैक्स को निकालता है. यह तर्क गेट & #34 के रूप में कार्य करता है; और & #34;. मूलतः, & #34; फिल् & #34; पटरियों की दो श्रेणियों ( जैसे , कुछ प्रयोगशालाओं के साथ कुछ ऊतकों) के बीच बातचीत के चयन की अनुमति देता है. ध्यान दें कि & #34; फ़िल्टर & #34; यदि वे पहले से ही क्वेरी में नहीं हैं, तो चयनित प्रकार के ट्रैक्स को क्वेरी में न जोड़ें. & #34; छोडी & #34; बटन सभी प्रकार की ट्रैक्स निकालता है जो वर्तमान में क्वेरी से फ़िल्टर विंडो में चयनित हैं । यह तर्क gate & #34 के रूप में कार्य करता है; न & #34;, विरोध में & #34; फ़िल्टर & #34; फ़ंक्शन. नु, त & #34; छोडी & #34; कोई भी ट्रैक्स वर्तमान में फ़िल्टर विंडो में क्वेरी करने के लिए चयनित नहीं जोड़ नहीं है । 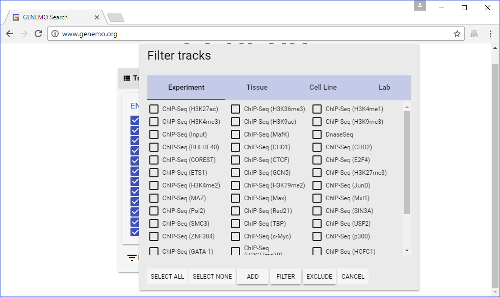 चित्रा 3 : फ़िल्टर विंडो . इस पर क्लिक करके लाया जाता है & #34; फ़िल्टर & #34; बटन ट्रैक चयन विंडो पर । यहाँ, उपयोगकर्ताओं को एक ही समय में कई पटरियों का चयन कर सकते हैं, रिश्तेदार आसानी के साथ. & #160; इस फिगर का बड़ा वर्जन देखने के लिए यहां क्लिक करें ।
चित्रा 3 : फ़िल्टर विंडो . इस पर क्लिक करके लाया जाता है & #34; फ़िल्टर & #34; बटन ट्रैक चयन विंडो पर । यहाँ, उपयोगकर्ताओं को एक ही समय में कई पटरियों का चयन कर सकते हैं, रिश्तेदार आसानी के साथ. & #160; इस फिगर का बड़ा वर्जन देखने के लिए यहां क्लिक करें । 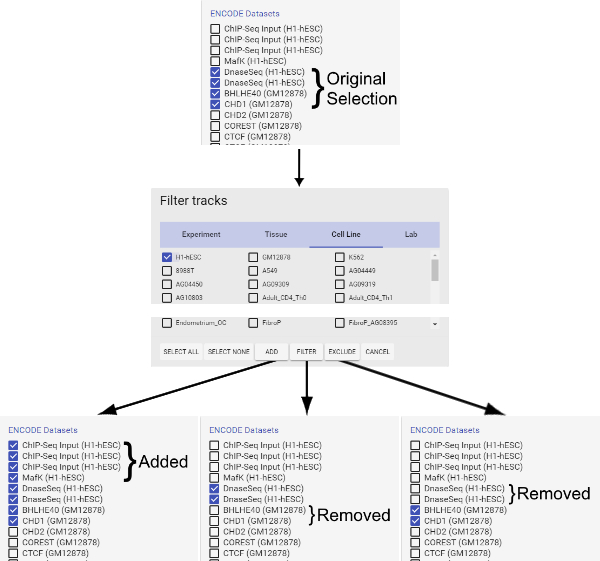 चित्रा ४ : फ़िल्टर फ़ंक्शन का उपयोग कैसे करें. & #160; इस फिगर का बड़ा वर्जन देखने के लिए कृपया यहां क्लिक करें । क्वेरी के लिए इच्छित ट्रैक जोड़ने के बाद, नीचे दाईं ओर & #34; Update & #34; बटन क्लिक करें । यह डेटा का चयन करने के लिए दो तरीके को समायोजित करने के लिए आवश्यक है: व्यक्तिगत डेटा पटरियों का चयन या फ़िल्टरिंग/ द & #34; रिसेट दृ & #34; बटन मानव/माउस भ्रूण स्टेम कोशिकाओं में जीन अभिव्यक्ति विनियमन से संबंधित डिफ़ॉल्ट ट्रैक करने के लिए क्वेरी रीसेट करता है । नोट: ट्रैक का चयन via & #34 के खिलाफ खोजा जा;D ata चयन & #34; वैकल्पिक है लेकिन अनुशंसितकारण डिफ़ॉल्ट खोज ट्रैक सबसे अधिक संभावना उपयोगकर्ता & #39; s आवश्यकताओं के लिए अनुकूल नहीं हैं ।
चित्रा ४ : फ़िल्टर फ़ंक्शन का उपयोग कैसे करें. & #160; इस फिगर का बड़ा वर्जन देखने के लिए कृपया यहां क्लिक करें । क्वेरी के लिए इच्छित ट्रैक जोड़ने के बाद, नीचे दाईं ओर & #34; Update & #34; बटन क्लिक करें । यह डेटा का चयन करने के लिए दो तरीके को समायोजित करने के लिए आवश्यक है: व्यक्तिगत डेटा पटरियों का चयन या फ़िल्टरिंग/ द & #34; रिसेट दृ & #34; बटन मानव/माउस भ्रूण स्टेम कोशिकाओं में जीन अभिव्यक्ति विनियमन से संबंधित डिफ़ॉल्ट ट्रैक करने के लिए क्वेरी रीसेट करता है । नोट: ट्रैक का चयन via & #34 के खिलाफ खोजा जा;D ata चयन & #34; वैकल्पिक है लेकिन अनुशंसितकारण डिफ़ॉल्ट खोज ट्रैक सबसे अधिक संभावना उपयोगकर्ता & #39; s आवश्यकताओं के लिए अनुकूल नहीं हैं । 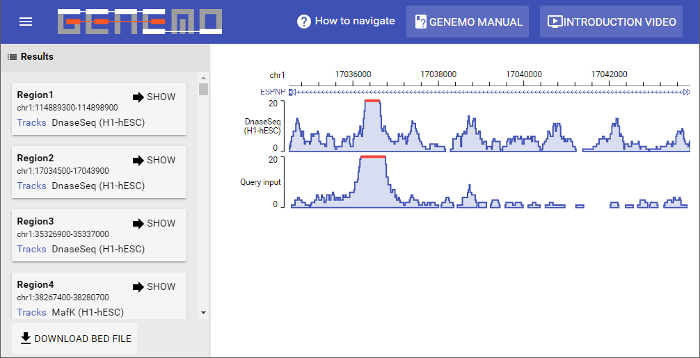 चित्रा ५ : परिणाम पृष्ठ . इस विशेष खोज ३६३ मिलान क्षेत्रों लौट आए । प्रथम मिलान क्षेत्र को प्रदर्शित करके, प्रत्येक परिणामी क्षेत्र बॉक्स के नीचे बाईं ओर & #34; SHOW & #34; बटन क्लिक करके किया जा सकता है । प्रदर्शन विंडो के बाएं भाग पर यह देखा जा सकता है कि दो डेटा फ़ाइलें (इनपुट और चयनित ट्रैक) संकेत शक्ति पैटर्न में समान हैं । & #160; कृपया click यहां इस आंकड़े का एक बड़ा संस्करण देखने के लिए ।
चित्रा ५ : परिणाम पृष्ठ . इस विशेष खोज ३६३ मिलान क्षेत्रों लौट आए । प्रथम मिलान क्षेत्र को प्रदर्शित करके, प्रत्येक परिणामी क्षेत्र बॉक्स के नीचे बाईं ओर & #34; SHOW & #34; बटन क्लिक करके किया जा सकता है । प्रदर्शन विंडो के बाएं भाग पर यह देखा जा सकता है कि दो डेटा फ़ाइलें (इनपुट और चयनित ट्रैक) संकेत शक्ति पैटर्न में समान हैं । & #160; कृपया click यहां इस आंकड़े का एक बड़ा संस्करण देखने के लिए ।
Representative Results
Discussion
epigenome की एक पूरी तरह से समझ के लिए नई जैविक अंतर्दृष्टि8प्रदान करने में मानव जीनोम अनुक्रमण की पूरी क्षमता प्राप्त करने की आवश्यकता है । वर्तमान में केवल अपने डेटा वर्णन और शीर्षक (जैसे, मेटाडेटा)1द्वारा ऑनलाइन epigenomic डेटासेट खोज करने के तरीके हैं. यह गंभीर रूप से एक खोज के प्रकार सीमा epigenomic डेटा के साथ कर सकते हैं । epigenomic डेटा के लिए प्रतिमान आधारित खोज उपकरण विभिंन epigenomic चिह्नों के बीच संबंध तलाशने के लिए आवश्यक हैं, जो नई जैविक अंतर्दृष्टि को जंम दे सकते हैं । GeNemo, जो डेटा और नहीं मेटाडाटा की सामग्री द्वारा खोजों, अपनी तरह की पहली सेवा है एक उपयोगकर्ता के साथ सांकेतिक शब्दों में बदलना डेटाबेस के रूप में प्रकाशित खजाने से epigenomic डेटा में पैटर्न की तुलना-उत्पंन या डाउनलोड किया गया डेटासेट5। यह एक epigenomic खोज उपकरण की उपलब्धता की शुरुआत है कि व्यापक रूप से बस के रूप में दुनिया भर के शोधकर्ताओं के लिए सुलभ है पाठ आधारित अनुक्रम खोज उपकरण के 1990 के दशक में व्यापक रूप से उपलब्ध हो गया निशान । वर्तमान में, GeNemo के अलावा अन्य epigenomic डेटा के लिए प्रतिमान आधारित ऑनलाइन खोज उपकरण के लिए कोई विकल्प नहीं हैं ।
GeNemo का उपयोग कर के एक संभावित उदाहरण के लिए खोज करने के लिए है सह citrullinated संशोधनों और मानव भ्रूण स्टेम कोशिकाओं में transcriptional कारक E2F6 के साथ अंय epigenetic के निशान (एक उदाहरण E2F6 बाध्यकारी संकेत फ़ाइल सांकेतिक शब्दों में बदलना डेटा पोर्टल पर उपलब्ध है या https://sysbio.ucsd.edu/public/xcao3/ENCODESample/ENCFF001UBC.bed) । क्वेरी के रूप में इस फ़ाइल का उपयोग करके H1-hESC में सभी एन्कोड datasets के विरुद्ध खोज करने के लिए, GeNemo दिखाएगा कि E2F6 बाइंडिंग संकेत भारी H3K4me1, H3K4me2, H3K4me3, और H3K27me3, जो मौजूदा दिखा रहा है कि E2F6 के माध्यम से कुछ जीनों को नियंत्रित करता है कि अनुसंधान के साथ सहमत से समृद्ध है H3K27 का मिथाइल9. दूसरी ओर, वहां E2F6 और CtBP2 बंधन साइटों की colocalization प्रतीत होता है, जो एक ही परिवार में एक कारक के साथ बातचीत करने के लिए जाना जाता है, E2F710। epigenetic के निशान की एक बड़ी संख्या के खिलाफ पूरे जीनोम के लिए इन परिणामों, transcriptional कारक बाध्यकारी संकेतों, और अंय संकेतों में शामिल सांकेतिक शब्दों में बदलना काफी आसानी से GeNemo, जो आगे विश्लेषण के लिए सभी संभावित लक्ष्य प्रदान कर सकते है के साथ प्राप्त कर सकते हैं ।
के बाद से पहली प्रकाशन5 GeNemo के एक वेब आधारित epigenomic डेटा खोज उपकरण के रूप में, GeNemo के परिणाम अनुभाग है GeNemo सामने पृष्ठ के साथ एक मेल उपस्थिति है अद्यतन किया गया है । पुराने परिणाम अनुभाग बारीकी से UCSC जीनोम ब्राउज़र परिणाम अनुभाग को प्रतिबिंबित किया, और काफी हद तक प्रदर्शन के लिए दूरदराज के UCSC सर्वर पर निर्भर था । नए इंटरफेस के साथ, GeNemo अधिक उपयोगकर्ता के अनुकूल है और अब UCSC जीनोम सर्वर पर निर्भर है (भले ही डेटा अभी भी दूर से दिलवाया हैं) । यह GeNemo और अधिक मजबूत बनाता है और कम UCSC सर्वर पर कोड परिवर्तन के कारण समस्याओं के लिए अतिसंवेदनशील । इसके अलावा, GeNemo के नए, तेजी से बहुलक इंटरफेस उपयोगकर्ता अधिक उपकरण को कल्पना और डेटा में पैटर्न का विश्लेषण देता है ।
महत्वपूर्ण कदम उपयुक्त इनपुट फ़ाइल प्रदान करने और के खिलाफ खोज के लिए डेटा पटरियों का चयन शामिल है । उपयोगकर्ताओं को दृढ़ता से विभिन्न ट्रैक चयन कार्यों के साथ प्रयोग करने के लिए प्रोत्साहित किया जाता है चयन प्रक्रिया से परिचित हो और कैसे अलग आदेशों उद्देश्य परिणाम प्राप्त करने के लिए जोड़ा जा सकता है । विशेष रूप से, ध्यान दें कि “जोड़ें” समारोह वांछित पटरियों क्वेरी के लिए चयनित जोड़ने के लिए आवश्यक है, जबकि “फ़िल्टर” या “बाहर” तर्क गेट कमानों के रूप में इस्तेमाल किया जा सकता है “और” और “या”, क्रमशः. “अद्यतन” फ़ंक्शन खोज को लागू करने से पहले सभी चयनों को प्रभावित करने के लिए आवश्यक है । जब कोई परिणाम नहीं दिए जाते हैं, तो उपयोगकर्ता इनपुट डेटा फ़ाइल की जांच, अधिक ट्रैक खोज या खोज श्रेणी बढ़ा सकता है । जब भी वहां एक त्रुटि है, वहां एक को परिभाषित क्या वास्तव में त्रुटि है खिड़की हो जाएगा । वहां कुछ अस्पष्ट त्रुटियां हैं, हालांकि । उदाहरण के लिए, जब विंडो कहता है कि ‘ कोई फ़ाइल अपलोड की गई थी, ‘ या तो कोई फ़ाइल अपलोड नहीं की गई थी, या अपलोड की गई फ़ाइल किसी स्वीकार्य स्वरूप की नहीं थी और फलस्वरूप, प्रोग्राम इसे सही ढंग से पढ़ नहीं पा रहा था । फ़ाइल अपलोड के लिए स्वीकार्य फ़ाइल स्वरूपों में दोनों अपलोड विधियों के लिए बिस्तर और चोटियों प्रारूप फ़ाइल, और ऑनलाइन लिंक केवल अपलोड के लिए bigWig शामिल हैं । इन फ़ाइल स्वरूपों के ज़िपित संस्करण भी स्वीकार्य हैं ।
इस दृष्टिकोण की वर्तमान सीमाएं अभी तक-होना करने के लिए अनुकूलित एल्गोरिदम और GeNemo में कार्यरत कार्यों में शामिल हैं । GeNemo अभी तक दिए गए किसी भी डेटासेट की व्याख्या पर कोई मार्गदर्शन प्रदान नहीं कर सकता । यह कार्य उन उपयोगकर्ताओं पर निर्भर है, जिनके जीनोम और epigenome के जीवविज्ञान में महत्वपूर्ण ज्ञान और विशेषज्ञता की आवश्यकता है. इसके अलावा, एक और मौजूदा सीमा है कि प्रयोक्ताओं की संवेदनशीलता और खोजों के शोर स्तर को बदल नहीं सकते । हम सुधार और इसके पैटर्न खोज क्षमताओं और भविष्य में डेटासेट संग्रह पर GeNemo विस्तार जारी रखने की उंमीद है ।
Disclosures
The authors have nothing to disclose.
Acknowledgements
यह काम NHGRI से niched, R01HG008135 से DP1HD087990 सहित NIH अनुदान द्वारा समर्थित किया गया था । हम बहुमूल्य प्रतिक्रिया के लिए Zhong लैब के सदस्यों को धंयवाद ।
लेखक योगदान:
X.C. और A.T.Z. नए इंटरफेस और सुविधाओं कोडिंग द्वारा GeNemo अद्यतन; A.T.Z. में घर नमूना वीडियो का उत्पादन किया; A.T.Z., एक्स. सी और S.Z. ने पेपर लिखे ।
References
- The ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature. 489, 57-74 (2012).
- Barski, A., et al. High-Resolution Profiling of Histone Methylations in the Human Genome. Cell. 129 (4), 823-837 (2007).
- Meaney, M. J., Ferguson-Smith, A. C. Epigenetic regulation of the neural transcriptome: the meaning of the marks. Nature Neuroscience. 13, 1313-1318 (2010).
- Roh, T. -. Y., Cuddapah, S., Cui, K., Zhao, K. The genomic landscape of histone modifications in human T cells. PNAS. 103 (43), 15782-15787 (2006).
- Zhang, Y., Cao, X., Zhong, S. GeNemo: a search engine for web-based functional genomic data. Nucleic Acids Res. 44, W122-W127 (2016).
- Fujita, P. A., Rhead, B., Zweig, A. S., Hinrichs, A. S., Karolchik, D., Cline, M. S., Goldman, M., Barber, G. P., Clawson, H., Coelho, A., et al. The UCSC Genome Browser database: update 2011. Nucleic Acids Res. 39, 876-882 (2011).
- Neph, S., Vierstra, J., Stergachis, A. B., Reynolds, A. P., Haugen, E., Vernot, B., Thurman, R. E., John, S., Sandstrom, R., Johnson, A. K., et al. An expansive human regulatory lexicon encoded in transcription factor footprints. Nature. 489, 83-90 (2012).
- Sarda, S., Hannenhalli, S. Next-generation sequencing and epigenomics research: a hammer in search of nails. Genomics Inform. 12 (1), 2-11 (2014).
- Storre, J., et al. Silencing of the Meiotic Genes SMC1β and STAG3 in Somatic Cells by E2F6. J Biol Chem. 280, 41380-41386 (2005).
- Liu, B., Shats, I., Angus, S. P., Gatza, M. L., Nevins, J. R. Interaction of E2F7 Transcription Factor with E2F1 and C-terminal-binding Protein (CtBP) Provides a Mechanism for E2F7-dependent Transcription Repression. J Biol Chem. 288, 24581-24589 (2013).

