Traducción de la purificación de afinidad de ribosoma (TRAP) para investigar el desarrollo de la raíz de Arabidopsis thaliana a una escala específica de tipo de célula
Summary
La traducción de la purificación de afinidad ribosoma (TRAP) ofrece la posibilidad de diseccionar programas de desarrollo con un procesamiento mínimo de órganos y tejidos. El protocolo produce ARN de alta calidad a partir de células dirigidas con una subunidad ribosomal etiquetada con proteína fluorescente verde (GFP). Las herramientas de análisis aguas abajo, como qRT-PCR o RNA-seq, revelan perfiles de expresión específicos del tejido y del tipo de célula.
Abstract
En este artículo, damos instrucciones prácticas para obtener datos de traducción de diferentes tipos de células raíz de Arabidopsis thaliana a través del método de traducción de purificación de afinidad ribosoma (TRAP) y la preparación de bibliotecas de baja entrada optimizadas consecutivas.
Como material de partida, empleamos líneas vegetales que expresan la proteína ribosomal etiquetada por GFP RPL18 de una manera específica del tipo de célula mediante el uso de promotores adecuados. Antes de la inmunopurificación y la extracción de ARN, el tejido se congela a presión, lo que preserva la integridad del tejido y simultáneamente permite la ejecución de estudios de series temporales con alta resolución temporal. En particular, las estructuras de las paredes celulares permanecen intactas, lo que es un inconveniente importante en procedimientos alternativos como enfoques basados en la clasificación celular activado por fluorescencia que se basan en el protoduro de tejidos para aislar poblaciones celulares distintas. Además, no es necesaria ninguna fijación tisular como en las técnicas basadas en microdisección de captura láser, lo que permite obtener ARN de alta calidad.
Sin embargo, el muestreo de subpoblaciones de células y solo el arn asociado al poliso limita gravemente los rendimientos del ARN. Por lo tanto, es necesario aplicar métodos de preparación de bibliotecas suficientemente sensibles para la adquisición exitosa de datos por ARN-seq.
TRAP ofrece una herramienta ideal para la investigación de plantas, ya que muchos procesos de desarrollo implican vías de señalización mecánica y relacionadas con la pared celular. El uso de promotores para dirigirse a poblaciones celulares específicas está cerrando la brecha entre el nivel de órgano y de una sola célula que a su vez sufren de poca resolución o costos muy altos. Aquí, aplicamos TRAP para estudiar la comunicación célula-célula en la formación de raíces laterales.
Introduction
Impulsada por la creciente aplicación de técnicas de secuenciación de próxima generación, se podría aumentar la resolución espacial en la biología del desarrollo. Los estudios contemporáneos tienen como objetivo disuajar tejidos hasta tipos de células especializadas, si no de un solo nivel de células1,,2,3,4. Para ello, en los últimos cincuenta años se ha ideado una plétora de métodos diferentes (véase la Figura 1A)5,6,7,8,9,10,11,12,13,14,15.
Muchas herramientas en la ciencia vegetal han sido adaptaciones de técnicas que fueron pioneras en la investigación animal. Este no es el caso del método que estamos introduciendo en detalle aquí. En 2005, equipado con una sólida experiencia en traducción de proteínas, el Bailey-Serres Lab se propuso diseñar proteínas ribosomales para la posterior purificación de afinidad16. Por lo tanto, podrían evitar el perfilado de polisomas que consume mucho tiempo y requiere mucha mano de obra, que se basa en la ultracentrifugación con un gradiente de sacarosa y se utilizó para evaluar la traducción de ribosomas desde la década de 196017,18. Desde entonces, el método se ha denominado purificación de afinidad ribosoma traslacional (TRAP)16. Después de estudios de traducción exitosos en plantas, Heiman et al. adaptó TRAP para animales19 y otros ampliaron su aplicación a la levadura20, Drosophila21, Xenopus22 y zebrafish23,,24.
Aunque la modificación genética del sistema modelo es un requisito previo para trap, que limita su aplicación a especies susceptibles a la transformación genética, se puede aprovechar simultáneamente esta objeción a los subconjuntos objetivo de células que son de especial interés y, por lo tanto, extremadamente difíciles de aislar del tejido/órgano intacto25 (por ejemplo, células dendríticas altamente ramificadas en un cerebro de ratón o hifas fúngicas en el tejido vegetal infectado). En las plantas, todas las células se mantienen en su lugar a través de paredes celulares que forman la base del esqueleto hidrostático26. Para liberar una célula vegetal de esta matriz, los científicos han cortado físicamente la célula de su tejido circundante a través de la microdisección de captura láser (LCM)27 o han realizado digestión enzimática de las paredes celulares28. Entre estas últimas células, las llamadas protoplastias, la población de interés está etiquetada fluorescentemente y se puede separar mediante la clasificación celular activada por fluorescencia (FACS)7. LCM generalmente requiere que una muestra sea fija e incrustada en la cera, lo que en última instancia deteriora la calidad de su ARN29. Los métodos basados en FACS producen ARN de alta calidad, pero el proceso de protoplasting en sí mismo introduce diferencias en la expresión génica30 y los tejidos con paredes celulares secundarias modificadas y gruesas son notoriamente difíciles de tratar. Además, se supone que muchos procesos de desarrollo en las plantas dependen de señales transmitidas mecánicamente y, por lo tanto, la integridad de la pared celular es de suma importancia31. Dos métodos, que utilizan un acceso directo para eludir el aislamiento celular operando en el nivel de nucleii, son la clasificación nuclear activada por fluorescencia (FANS) y el aislamiento de núcleos etiquetados en tipos de células específicos (INTACT). Al igual que en TRAP, utilizan promotores específicos del tipo de celda para marcar los núcleos, que posteriormente se enriquecen a través de la clasificación o el tirón hacia abajo, respectivamente8,15. Un desafío importante para todos estos enfoques es obtener suficiente material de ARN de subconjuntos de células en un tejido. Como TRAP captura solo una fracción de los ARN celulares, la recolección de muestras es un cuello de botella considerable. Por lo tanto, se necesitan protocolos de preparación de bibliotecas especialmente sensibles para producir datos de alta calidad a partir de cantidades de entrada bajas.
Desde su creación, TRAP se ha utilizado en combinación con microarrays de ADN o, como los costos de secuenciación disminuyeron significativamente en los últimos años, ARN-seq10,32,33. Una multitud de preguntas de investigación ya se ha aclarado como se revisó en Sablok et al.34. Estamos convencidos de que seguirán más informes en los próximos años, ya que la técnica es muy versátil a la hora de combinar diferentes promotores para apuntar a tipos de células específicas. Eventualmente, esto se hará incluso de una manera inducible, y puede combinarse con el sondeo de la reacción de la planta a muchos factores de estrés biótico y abiótico. Además, cuando no se dispone de líneas transgénicas estables, los sistemas de expresión de raíces peludas también se han utilizado con éxito para realizar TRAP en tomate y medicago35,,36.
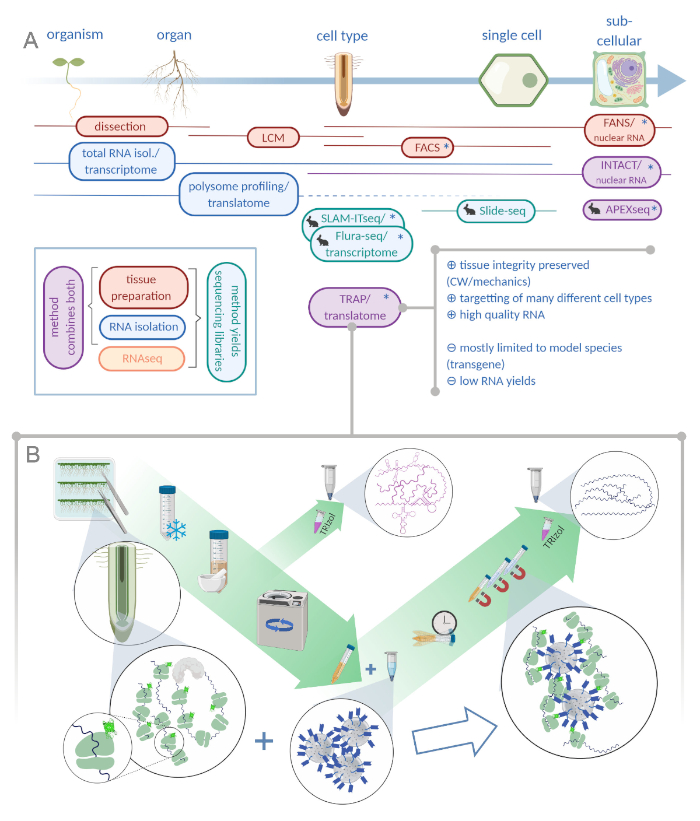
Figura 1: La traducción de la purificación de afinidad ribosoma (TRAP) complementa la cartera de análisis “omics”. A. El aumento de los niveles de precisión analítica, hasta una sola célula o incluso la resolución subcelular se puede lograr mediante una plétora de métodos o combinaciones de los mismos. El esquema ofrece una visión general de las herramientas disponibles actualmente en el campo vegetal y animal. La recolección de tejidos a resolución celular se puede lograr mediante protocolos como LCM o FACS, que luego se acoplan al transcriptoma estándar o al análisis de perfiles/traducción de polisoma. TRAP e INTACT integran la captura de tejido y el aislamiento de ARN, ya que se basan en el etiquetado de epítopos. Sin embargo, INTACT sólo toma muestras de núcleos celulares y constituye, por lo tanto, un caso especial de análisis de transcriptoma. Un pequeño icono de conejo marca los métodos recientemente desarrollados en el campo animal: Mientras que SLAM-ITseq y Flura-seq se basan en el objetivo metabólico de ARN nacientes con bases de uracilo modificadas en células que expresan la enzima permisiva, Slide-seq hace uso de un portaobjetos de vidrio recubierto con códigos de barras de ADN que proporcionan información posicional en el rango celular. Se sigue un enfoque de etiquetado de proximidad en APEX-seq para muestrear ARN en compartimentos subcelulares específicos. En particular, el aumento de la resolución a menudo requiere la generación de material transgénico (asteriscos) y, por lo tanto, estos métodos se utilizan predominantemente para las especies modelo. TRAP es especialmente adecuado para estudios de ciencias vegetales que involucran pared celular (CW) o señalización mecánica, así como especies celulares que son difíciles de liberar de su matriz CW. B. Pasos detallados de laboratorio húmedo del procedimiento TRAP: Las plántulas que expresan proteína ribosomal etiquetada con GFP en distintos tipos de células (por ejemplo, endodermis de raíz) se cultivan en platos De Petri durante siete días y el material radicular cosechado por congelación rápida. Se recoge una muestra de control total de ARN del extracto crudo homogeneizado antes de peletizar los escombros a través de la centrifugación. Las perlas magnéticas anti-GFP se añaden al extracto transparente para realizar la inmunoprecipitación. Después de la incubación y tres pasos de lavado, el ARN asociado al polisoma (ARN TRAP/polisoma) se obtiene directamente a través de la extracción de fenol-cloroformo. LCM: microdisección de captura láser, FACS/FANS: clasificación celular/nuclear activada por fluorescencia, APEX-seq: método basado en ascorbación peroxidasa diseñada, INTACT: aislamiento de núcleos etiquetados en tipos de células específicas, alquilación vinculada a SLAM-ITseq: thiol(SH) para la secuenciación metabólica de ARN en tejido, Flura-seq: secuenciación de ARN con etiqueta de fluorouracilo (Creado con Biorender.com) Haga clic aquí para ver una versión más grande de esta figura.
El objetivo de este artículo es proporcionar una descripción detallada del método TRAP, resaltar los pasos críticos y proporcionar orientación para un posible método de preparación de biblioteca.
Un experimento trap genérico consistirá esencialmente en los siguientes pasos (véase también la figura 1B):(1) Preparación de material vegetal, incluida la clonación de la construcción de etiquetado de ribosoma, producción y selección de líneas transgénicas, cultivo y abultamiento de semillas, esterilización y chapado, y aplicación/tratamiento de tensión (opcional) y recolección de tejidos; 2) inmunopurificación, incluida la homogeneización tisular y la limpieza del extracto crudo, el lavado de cuentas y la inmunopurificación, y las etapas de lavado; 3) extracción de ARN y evaluación de la calidad; y (4) la preparación de la biblioteca.
La raíz de Arabidopsis ha sido un sistema modelo para estudiar el desarrollo de la planta desde su introducción como planta modelo37,,38. Aquí, la aplicación de TRAP se muestra en el contexto del desarrollo de la raíz lateral de la planta. En las plantas, la acumulación de todo el sistema radicular se basa en la ejecución de este programa y por lo tanto es muy importante para la supervivencia del organismo39. En Arabidopsis,las raíces laterales se originan a partir de tejido periciclo que reside junto a vasos xilemas y, por lo tanto, se denomina periciclo de polo xilem (XPP; véase la figura 2C)40. Algunas células XPP, que se encuentran en lo profundo de la raíz, adquieren una identidad celular fundadora y, tras un desencadenante hormonal local, comienzan a proliferar por hinchazón y división anticlinalmente41. Sin embargo, debido a la presencia de una matriz de pared celular rígida, este proceso ejerce tensión mecánica en los tejidos circundantes. En particular, la endodermis superpuesta se ve afectada, ya que se encuentra en el camino del eje de crecimiento de la raíz lateral42,,43,44. De hecho, el primordium recién formado tendrá que crecer a través de la célula de endodermis superpuesta(Figura 2C2),mientras que las células de corteza y epidermis simplemente se apartan para que el primordium finalmente surja45,46. El trabajo reciente en nuestro laboratorio ha demostrado que la endodermis está contribuyendo activamente a acomodar la proliferación en el periciclo. El bloqueo dirigido de la señalización hormonal endodérmica es suficiente para inhibir incluso la primera división en las células XPP47. Por lo tanto, la comunicación periciclo-endodermis constituye un punto de control muy temprano para el desarrollo de la raíz lateral en Arabidopsis. Sin embargo, no se sabe cómo se realiza esta charla cruzada. Para desentrañar este misterio, elegimos el enfoque TRAP-seq para apuntar a las células XPP y endodeérmicas. Para enriquecer las células en el programa de raíz lateral, imitamos el desencadenante hormonal aplicando exógenamente un análogo de auxina (ácido 1-naftalencia, NAA)48,que al mismo tiempo permitió resolver temporalmente la fase inicial de la formación de la raíz lateral.
Protocol
Representative Results
Discussion
Verificación del patrón de localización RPL18
El patrón de expresión adecuado de la subunidad ribosomal etiquetada es fundamental para evitar la interpretación errónea de los datos de cualquier experimento TRAP. Por lo tanto, la incorporación de GFP como etiqueta de epítope a RPL18 permite muy elegantemente la verificación del patrón de expresión deseado y, consecutivamente, la extracción de la fracción de polisoma del mismo tejido. Los enfoques más invasivos para asegurar patrones de p…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Nos gustaría dar las gracias a Jean-Claude Walser, del Centro de Diversidad Genética de Zúrich, por su asesoramiento experto crucial en la fase inicial de este proyecto. El trabajo en el laboratorio de Vermeer fue apoyado por una beca de cátedra SNF (PP00P3_157524) y una subvención de equipo R’EQUIP (316030_164086) de la Fundación Nacional Suiza de Ciencias (SNSF) otorgada a JEMV.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

