Oversette ribosom affinitet rensing (TRAP) for å undersøke Arabidopsis thaliana Root Development på en celle type-spesifikk skala
Summary
Oversette ribosome affinitet rensing (TRAP) gir mulighet til å dissekere utviklingsprogrammer med minimal behandling av organer og vev. Protokollen gir høy kvalitet RNA fra celler målrettet med en grønn fluorescerende protein (GFP)-merket ribosomal subunit. Nedstrøms analyseverktøy, for eksempel qRT-PCR eller RNA-seq, avslører vevs- og celletypespesifikke uttrykksprofiler.
Abstract
I denne artikkelen gir vi praktiske instruksjoner for å få translatome data fra forskjellige Arabidopsis thaliana rotcelletyper via den oversette ribosome affinitetsrensing (TRAP) metoden og påfølgende optimalisert lav-input bibliotek forberedelse.
Som startmateriale bruker vi plantelinjer som uttrykker GFP-merket ribosomalt protein RPL18 på en celletypespesifikk måte ved bruk av tilstrekkelige arrangører. Før immunrensing og RNA-ekstraksjon blir vevet frosset, noe som bevarer vevsintegritet og samtidig tillater gjennomføring av tidsseriestudier med høy temporal oppløsning. Spesielt forblir celleveggstrukturer intakte, noe som er en stor ulempe i alternative prosedyrer som fluorescensaktiverte cellesorteringsbaserte tilnærminger som er avhengige av vevsomforsynt for å isolere forskjellige cellepopulasjoner. I tillegg er ingen vevfiksering nødvendig som i laserfangst mikrodisseksjonsbaserte teknikker, noe som gjør at høykvalitets RNA kan oppnås.
Prøvetaking fra underpopulasjoner av celler og bare isolere polysom-assosiert RNA begrenser rna-utbytter alvorlig. Det er derfor nødvendig å anvende tilstrekkelig sensitive bibliotekforberedelsesmetoder for vellykket datainnhenting av RNA-seq.
TRAP tilbyr et ideelt verktøy for planteforskning, da mange utviklingsprosesser involverer celleveggrelaterte og mekaniske signalveier. Bruken av arrangører for å målrette spesifikke cellepopulasjoner er å bygge bro mellom organ- og encellenivå som igjen lider av liten oppløsning eller svært høye kostnader. Her bruker vi TRAP for å studere cellecellekommunikasjon i lateral rotdannelse.
Introduction
Drevet av den økende anvendelsen av neste generasjons sekvenseringsteknikker, kan romlig oppløsning i utviklingsbiologi økes. Moderne studier tar sikte på å dissekere vev ned til spesialiserte celletyper, om ikke encellede nivå1,2,3,4. For dette formål har en mengde forskjellige metoder blitt utarbeidet i løpet av de siste femti årene (se figur 1A)5,6,7,8,9,10,11,12,13,14,15.
Mange verktøy i plantevitenskap har vært tilpasninger av teknikker som var pionerer i dyreforskning. Dette er ikke tilfelle for metoden vi introduserer i detalj her. I 2005, utstyrt med sterk bakgrunn i proteinoversettelse, satte Bailey-Serres Lab ut for å konstruere ribosomale proteiner for påfølgende affinitetsrensing16. Dermed kunne de unngå tidkrevende og arbeidskrevende polysom profilering, som er basert på ultracentrifugering med en sukrosegradient og ble brukt til å vurdere å oversette ribosomer siden 1960-tallet17,18. Metoden har siden blitt referert til som translasjonell ribosom affinitet rensing (TRAP)16. Etter vellykkede translatome studier i planter, Heiman et al. tilpasset TRAP for dyr19 og andre utvidet sin søknad til gjær20, Drosophila21, Xenopus22 og sebrafisk23,24.
Selv om genetisk modifisering av modellsystemet er en forutsetning for TRAP, som begrenser anvendelsen til arter som er egnet til genetisk transformasjon, kan man samtidig utnytte denne innvendingen mot undergrupper av celler som er av spesiell interesse og ellers ekstremt vanskelig å isolere fra det intakte vevet/orgelet25 (f.eks. svært forgrenede dendrittiske celler i en mushjerne eller sopphyphae i infisert plantevev). I planter holdes alle celler på plass via cellevegger som danner grunnlaget for det hydrostatiske skjelettet26. For å frigjøre en plantecelle fra denne matrisen, har forskere enten fysisk kuttet cellen ut av sitt omkringliggende vev gjennom laserfangstmikrodisseksjon (LCM)27 eller utført enzymatisk fordøyelse av celleveggene28. Blant de sistnevnte cellene, såkalte protoplasts, er befolkningen av interesse fluorescerende merket og kan skilles via fluorescensaktivert cellesortering (FACS)7. LCM krever vanligvis en prøve som skal festes og bygges inn i voks, noe som til slutt forverrer kvaliteten på RNA29. FACS-baserte metoder gir høy kvalitet RNA, men prosessen med å protopelastere i seg selv introduserer forskjeller i genuttrykk30 og vev med modifiserte og tykke sekundære cellevegger er notorisk vanskelig å behandle. Videre antas mange utviklingsprosesser i planter å stole på mekanisk overførte signaler, og derfor er integriteten til celleveggen av avgjørende betydning31. To metoder, som bruker en snarvei til å omgå celleisolasjon ved å operere på nivået av kjerner, er fluorescensaktivert kjernefysisk sortering (FANS) og isolering av kjerner merket i bestemte celletyper (INTAKT). Som i TRAP bruker de celletypespesifikke arrangører for å markere kjerner, som senere blir beriket via sortering eller pull down, henholdsvis8,15. En stor utfordring for alle disse tilnærmingene er å få tilstrekkelig RNA-materiale fra undergrupper av celler i et vev. Som TRAP fanger bare en brøkdel av cellulære RNAs, prøvesamling er en betydelig flaskehals. Spesielt sensitive bibliotekforberedelsesprotokoller er derfor nødvendig for å produsere data av høy kvalitet fra lave inngangsbeløp.
Siden etableringen har TRAP enten blitt brukt i kombinasjon med DNA-mikroarrayer eller, som sekvenseringskostnader falt betydelig de siste årene, RNA-seq10,32,33. En rekke forskningsspørsmål har allerede blitt belyst som gjennomgått i Sablok et al.34. Vi er overbevist om at flere rapporter vil følge i de kommende årene som teknikken er svært allsidig når du kombinerer ulike arrangører for å målrette bestemte celletyper. Til slutt vil dette bli gjort selv på en utilbørlig måte, og kan kombineres med å undersøke anleggets reaksjon på mange biotiske og abiotiske stressfaktorer. I tillegg, der stabile transgene linjer ikke er tilgjengelige, har hårete rotuttrykkssystemer også blitt brukt til å utføre TRAP i tomat og medicago35,36.
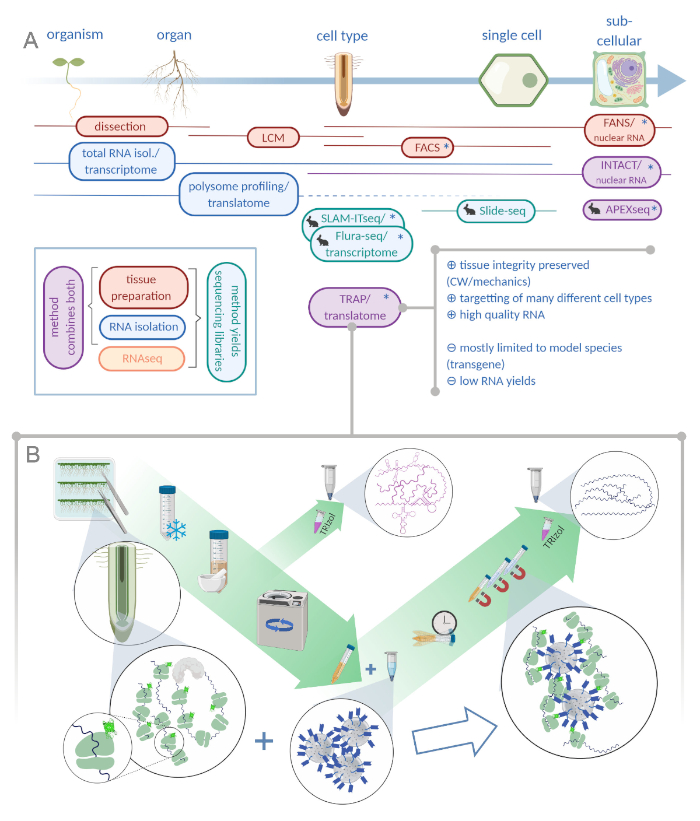
Figur 1: Translating ribosome affinity purification (TRAP) utfyller “omics” analyseporteføljen. A. Økende nivåer av analytisk presisjon, ned til encellede eller til og med subcellulær oppløsning kan oppnås ved en mengde metoder eller kombinasjoner av disse. Ordningen gir en oversikt over tilgjengelige verktøy i plante- og dyrefeltet. Vevsinnsamling ved cellulær oppløsning kan oppnås ved protokoller som LCM eller FACS, som deretter kobles til standard transkripsjon eller polysome profilering / translatome analyse. TRAP og INTAKT integrerer både vevsfangst og RNA-isolasjon som de er basert på epitop-tagging. Intakte prøver bare cellekjerner og utgjør derfor et spesielt tilfelle av transkripsjonsanalyse. Et lite kaninikon markerer nyutviklede metoder i dyrefeltet: Mens SLAM-ITseq og Flura-seq er avhengige av metabolsk targetting av nascent RNAs med modifiserte uracilbaser i celler som uttrykker det tillatte enzymet, bruker Slide-seq et belagt glasslysbilde med DNA-strekkoder som gir posisjonsinformasjon i cellulærområdet. En nærhetsmerkingstilnærming følges i APEX-seq for å prøve RNAer i bestemte undercellulære rom. Spesielt krever økt oppløsning ofte generering av transgen materiale (stjerner) og disse metodene brukes dermed hovedsakelig til modellarter. TRAP er spesielt egnet for plantevitenskapsstudier som involverer cellevegg (CW) eller mekanikersignalering samt cellearter som er vanskelige å frigjøre fra cw-matrisen. B. Detaljerte våtlabtrinn i TRAP-prosedyren: Frøplanter som uttrykker GFP-merket ribosomalt protein i forskjellige celletyper (f.eks. rotendodermis) dyrkes på Petri-retter i syv dager og rotmateriale høstet ved snap frysing. En total RNA-kontrollprøve samles inn fra det homogeniserte råoljeekstraktet før ruskene blir avstøtende via sentrifugering. Magnetiske anti-GFP perler legges til det klarerte ekstraktet for å utføre immunnedbør. Etter inkubasjon og tre vasketrinn oppnås polysomassosiert RNA (TRAP/polysome RNA) direkte via fenol-kloroformekstraksjon. LCM: laser fangst mikrodisseksjon, FACS / FANS: fluorescens-aktivert celle / kjernefysisk sortering, APEX-seq: metode basert på konstruert ascorbat peroksidans, INTAKT: isolering av kjerner merket i spesifikke celletyper, SLAM-ITseq: tiol (SH)-koblet alkylation for metabolsk sekvensering av RNA i vev, Flura-seq: fluorouracil-merket RNA sekvensering (Opprettet med Biorender.com) Vennligst klikk her for å se en større versjon av denne figuren.
Målet med denne artikkelen er å gi en detaljert beskrivelse av TRAP-metoden, for å markere kritiske trinn og gi veiledning for en mulig bibliotekklargjøringsmetode.
Et generisk TRAP-eksperiment vil i hovedsak bestå av følgende trinn (se også figur 1B): (1) Tilberedning av plantemateriale, inkludert kloning av ribosom-tagging konstruksjon, transgen linjeproduksjon og valg, voksende og bulking opp av frø, sterilisering og plating, og stress søknad / behandling (valgfritt) og vev høsting; (2) immunrensing inkludert vevhomogenisering og rydding av råekstrakt, perlevask og immunrensing, og vasketrinn; (3) RNA-ekstraksjon og kvalitetsvurdering; og (4) bibliotekforberedelse.
Arabidopsis roten har vært et modellsystem for å studere planteutvikling helt siden introduksjonen som modellanlegg37,38. Her vises anvendelsen av TRAP i sammenheng med plantesiderotutvikling. I planter er oppbyggingen av hele rotsystemet avhengig av utførelsen av dette programmet og er derfor svært viktig for organismens overlevelse39. I Arabidopsisstammer laterale røtter fra pericycle vev som ligger ved siden av xylem fartøy og derfor kalles xylem pole pericycle (XPP; se figur 2C)40. Noen XPP-celler, som ligger dypt inne i roten, får en grunnleggercelleidentitet og, på en lokal hormonell utløser, begynner å spre seg ved hevelse og dele anticlinally41. Men på grunn av tilstedeværelsen av en stiv celleveggmatrise, utøver denne prosessen mekanisk stress på det omkringliggende vevet. Spesielt påvirkes den overliggende endodermis, da den er i veien for den laterale rotvekstaksen42,43,44. Faktisk må det nylig dannede primordium vokse gjennom den overliggende endodermiscellen (Figur 2C2) mens cortex- og epidermis-celler bare skyves til side for primordium å endelig dukke opp45,46. Nylig arbeid i laboratoriet vårt har vist at endodermis aktivt bidrar til å imøtekomme spredningen i perisyklusen. Målrettet blokkering av endodermal hormonell signalisering er tilstrekkelig til å hemme selv den aller første divisjonen i XPP-cellene47. Derfor utgjør pericycle-endodermis kommunikasjon et svært tidlig kontrollpunkt for lateral rotutvikling i arabidopsis. Det er imidlertid ikke kjent hvordan denne crosstalk utføres. For å løse dette mysteriet valgte vi TRAP-seq-tilnærmingen for å målrette XPP og endodermale celler. For å berike for celler i siderotprogrammet, etterlignet vi den hormonelle utløseren ved å eksogene bruke en auxin analog (1-naftaleneddiksyre, NAA)48, som samtidig lov til å midlertidig løse den første fasen av lateral rotdannelse.
Protocol
Representative Results
Discussion
Verifisering av RPL18 lokaliseringsmønster
Avgjørende for å unngå feiltolkning av data fra et TRAP-eksperiment er riktig uttrykksmønster for den kodede ribosomale underenheten. Derfor tillater inkorporering av GFP som en epitoptag til RPL18 svært elegant verifisering av ønsket uttrykksmønster og fortløpende, nedtrekking av polysome fraksjonen fra samme vev. Mer invasive tilnærminger for å sikre riktig promotor mønstre etterfølges av Jiao og Mayerowitz 2010, som krever GUS-farging og i Tia…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi vil gjerne takke Jean-Claude Walser fra Genetic Diversity Center Zurich for avgjørende ekspertråd i den tidlige fasen av dette prosjektet. Arbeidet i Vermeer-laboratoriet ble støttet av et SNF Professorship-stipend (PP00P3_157524) og et R’EQUIP-utstyrsstipend (316030_164086) fra Swiss National Science Foundation (SNSF) tildelt JEMV.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

