Översätta Ribosom Affinity Rening (TRAP) för att undersöka Arabidopsis thaliana Root Development på en celltyp-specifik skala
Summary
Translating ribosomaffinitet rening (TRAP) erbjuder möjligheten att dissekera utvecklingsprogram med minimal bearbetning av organ och vävnader. Protokollet ger högkvalitativt RNA från celler riktade med ett grönt fluorescerande protein (GFP)-märkt ribosomal underavdelning. Nedströms analysverktyg, till exempel qRT-PCR eller RNA-seq, avslöjar vävnads- och celltypspecifika uttrycksprofiler.
Abstract
I den här artikeln ger vi praktiska instruktioner för att erhålla översättningsdata från olika Arabidopsis thaliana rotcellstyper via metoden översätta ribosomaaffinitetsrening (TRAP) och på varandra följande optimerade biblioteksförberedelser med låg ingång.
Som utgångsmaterial använder vi växtlinjer som uttrycker GFP-märkt ribosomalt protein RPL18 på ett celltypspecifikt sätt med hjälp av lämpliga initiativtagare. Före immunorening och RNA-extraktion är vävnaden snäppfryst, vilket bevarar vävnadsintegriteten och samtidigt möjliggör utförande av tidsseriestudier med hög temporal upplösning. Framför allt förblir cellväggsstrukturerna intakta, vilket är en stor nackdel i alternativa förfaranden såsom fluorescensaktiverade cellsorteringsbaserade metoder som är beroende av vävnadsprotoplasting för att isolera olika cellpopulationer. Dessutom är ingen vävnad fixering nödvändig som i laser fånga mikrodissection-baserade tekniker, vilket gör att högkvalitativa RNA som skall erhållas.
Provtagning från subpopulationer av celler och endast isolera polysome-associerade RNA begränsar dock kraftigt RNA avkastning. Det är därför nödvändigt att tillämpa tillräckligt känsliga metoder för förberedelse av bibliotek för framgångsrik datainsamling av RNA-seq.
TRAP erbjuder ett idealiskt verktyg för växtforskning eftersom många utvecklingsprocesser involverar cellväggsrelaterade och mekaniska signalvägar. Användningen av initiativtagare för att rikta in sig på specifika cellpopulationer överbryggar klyftan mellan organ- och encellig nivå som i sin tur lider av små upplösningar eller mycket höga kostnader. Här tillämpar vi TRAP för att studera cellcellskommunikation i lateral rotbildning.
Introduction
Driven av den ökande tillämpningen av nästa generations sekvenseringstekniker skulle rumslig upplösning i utvecklingsbiologi kunna utökas. Samtida studier syftar till att dissekera vävnader ner till specialiserade celltyper, om inte encellig nivå1,,2,,3,4. I detta syfte har en uppsjö av olika metoder utarbetats under de senaste femtio åren (se figur 1A)5,,6,,7,,8,,9,,10,,11,,12,,13,,14,,15.
Många verktyg inom växtvetenskap har varit anpassningar av tekniker som var pionjärer inom djurforskning. Detta är inte fallet för den metod som vi inför i detalj här. Under 2005, utrustad med en stark bakgrund inom proteinöversättning, Bailey-Serres Lab syftar till att ingenjör ribosomala proteiner för efterföljande affinitet rening16. Således kunde de undvika tidskrävande och arbetsintensiv polysome profilering, som bygger på ultracentrifugering med en sackaros gradient och användes för att bedöma översätta ribosomer sedan 1960-talet17,18. Metoden har sedan dess kallat translationell ribosomaffinitetsrening (TRAP)16. Efter framgångsrika översättarstudier på växter, Heiman et al. anpassade TRAP för djur19 och andra utvidgas dess tillämpning till jäst20, Drosophila21, Xenopus22 och zebrafisk23,24.
Även om genetisk modifiering av modellsystemet är en förutsättning för TRAP, som begränsar dess tillämpning till arter som kan användas för genetisk omvandling, kan man samtidigt utnyttja denna invändning mot målundergrupper av celler som är av särskilt intresse och i övrigt extremt svårt att isolera från intakt vävnad/organ25 (t.ex. höggrenade dendritiska celler i en mushjärna eller svamphyfa i infekterad växtvävnad). I växter hålls alla celler på plats via cellväggar som ligger till grund för det hydrostatiska skelettet26. För att frigöra en växtcell från denna matris, forskare har antingen fysiskt skära cellen ur sin omgivande vävnad genom laser fånga microdissection (LCM)27 eller utförs enzymatisk matsmältning av cellväggarna28. Bland de senare cellerna, så kallade protoplaster, är populationen av intresse fluorescerande märkt och kan separeras via fluorescensaktiverad cellsortering (FACS)7. LCM kräver vanligtvis ett prov som skall fastställas och inbäddad i vax, vilket i slutändan försämrar kvaliteten på dess RNA29. FACS-baserade metoder ger högkvalitativa RNA, men processen att protoplasting själv introducerar skillnader i genuttryck30 och vävnader med modifierade och tjocka sekundära cellväggar är notoriskt svåra att behandla. Dessutom antas många utvecklingsprocesser i anläggningar förlita sig på mekaniskt överförda signaler och därför är cellväggens integritet av största vikt31. Två metoder, som använder en genväg för att kringgå cellisolering genom att arbeta på nivån av kärnor, är fluorescensaktiverad kärnsortering (FANS) och isolering av kärnor taggade i specifika celltyper (INTAKT). Liksom i TRAP använder de celltypspecifika promotorer för att markera kärnor, som därefter berikas via sortering eller pull down,respektive 8,15. En stor utmaning för alla dessa metoder är att få tillräckligt RNA-material från delmängder av celler i en vävnad. Som TRAP fångar bara en bråkdel av den cellulära RNAs, prov insamling är en betydande flaskhals. Därför behövs särskilt känsliga biblioteksförberedelseprotokoll för att producera högkvalitativa data från låga indatamängder.
Sedan starten har TRAP antingen använts i kombination med DNA-mikroarrayer eller, som sekvensering kostnader minskat betydligt under de senaste åren, RNA-seq10,32,33. En mängd forskningsfrågor har redan klarlagts som granskats i Sablok et al.34. Vi är övertygade om att fler rapporter kommer att följa under de kommande åren eftersom tekniken är mycket mångsidig när man kombinerar olika initiativtagare för att rikta specifika celltyper. Så småningom, Detta kommer att ske även på ett inducerande sätt, och kan kombineras med sondering av anläggningens reaktion på många biotiska och abiotiska stressfaktorer. Dessutom, där stabila transgena linjer inte finns tillgängliga, håriga rot uttryckssystem har också framgångsrikt använts för att utföra TRAP i tomat och medicago35,36.
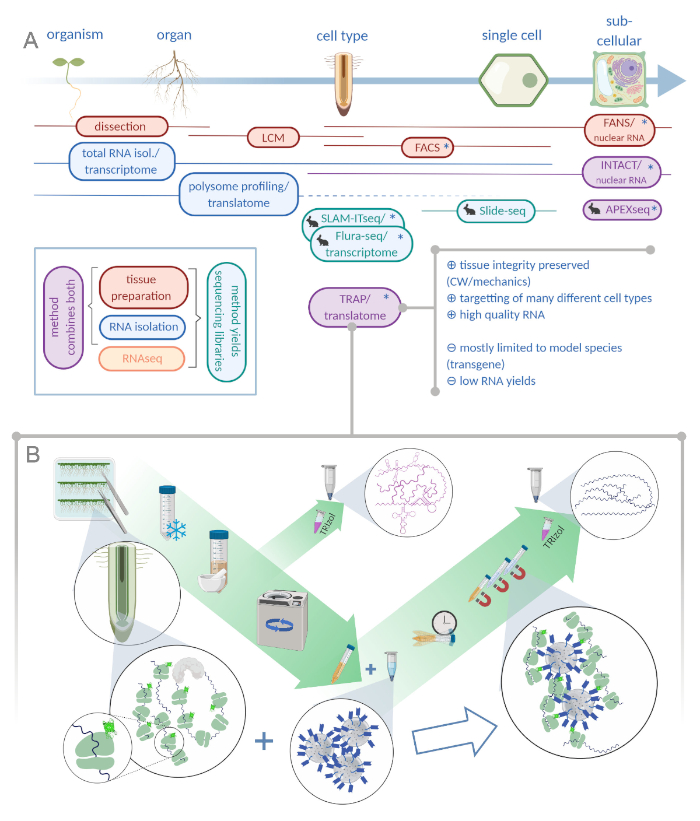
Figur 1: Omräkning av ribosomaffinitetsrening (TRAP) kompletterar analysportföljen “omics”. A. Ökande nivåer av analytisk precision, ner till encellig eller till och med subcellulär upplösning kan uppnås genom en uppsjö av metoder eller kombinationer av dessa. Systemet ger en översikt över de verktyg som för närvarande finns tillgängliga på växt- och djurområdet. Vävnadssamling vid cellupplösning kan uppnås genom protokoll som LCM eller FACS, som sedan kopplas till standard transkriptom eller polysome profilering / översättaranalys. TRAP och INTACT integrerar både vävnadsfångst och RNA-isolering eftersom de är baserade på epitop-märkning. Intakt prover endast cellkärnor och utgör därför ett specialfall av transkriptom analys. En liten kanin ikon markerar nyutvecklade metoder inom djurområdet: Medan SLAM-ITseq och Flura-seq förlita sig på metabolisk targetting av begynnande RNAs med modifierade uracil baser i celler som uttrycker tillåtande enzym, Slide-seq använder sig av en bestruken glas bild med DNA-streckkoder som ger positionsinformation i cellområdet. En närhetsmärkning följs i APEX-seq för att prova RNA i specifika subcellulära fack. Framför allt kräver ökad upplösning ofta generering av transgent material (asterisker) och dessa metoder används därför främst för modellarter. TRAP är särskilt lämpad för växtvetenskapliga studier med cellvägg (CW) eller mekaniker signalering samt cellarter som är svåra att släppa från sin CW matris. B. Detaljerade våt-lab steg i TRAP förfarandet: Plantor som uttrycker GFP-märkt ribosomalt protein i olika celltyper (t.ex. rot endodermis) odlas på petriskålar i sju dagar och rotmaterial skördas genom snap frysning. Ett totalt RNA-kontrollprov samlas in från det homogeniserade råextraktet innan skräpet pelleteras via centrifugering. Magnetiska anti-GFP pärlor läggs till rensade extrakt för att utföra immunprecipitation. Efter inkubation och tre tvättsteg erhålls det polysome-associerade RNA (TRAP/polysome RNA) direkt via fenol-kloroformextraktion. LCM: mikroavskiljning av laseravskiljning, FACS/FLÄKTAR: fluorescensaktiverad cell-/kärnsortering. APEX-seq: metod baserad på konstruerad askorbatperoxidas, INTAKT: isolering av kärnor taggade i specifika celltyper, SLAM-ITseq: thiol(SH)-linked alkylation för metabolisk sekvensering av RNA i vävnad, Flura-seq: fluorouracil-märkt RNA-sekvensering (Skapad med Biorender.com) Klicka här för att se en större version av denna figur.
Målet med den här artikeln är att tillhandahålla en detaljerad beskrivning av TRAP-metoden, att markera kritiska steg och att ge vägledning för en möjlig biblioteksförberedelsemetod.
Ett generiskt TRAP-experiment kommer huvudsakligen att bestå av följande steg (se även figur 1B):(1) Beredning av växtmaterial, inklusive kloning av ribosommärkningskonstruktion, transgen linjeproduktion och val, odling och sammanslagning av frön, sterilisering och plätering samt stressapplicering/behandling (frivillig uppgift) och vävnadsskörd. (2) immunrening inklusive vävnad homogenisering och clearing av rå extrakt, pärla tvätt och immunrening, och tvätta steg; (3) RNA-utvinning och kvalitetsbedömning. och (4) biblioteksförberedelser.
Den Arabidopsis roten har varit en modell system för att studera växtutveckling ända sedan den infördes som en modell anläggning37,38. Här visas tillämpningen av TRAP upp i samband med växt lateral rot utveckling. I växter, uppbyggnaden av hela rotsystemet bygger på genomförandet av detta program och är därför mycket viktigt för överlevnaden av organismen39. I Arabidopsiskommer laterala rötter från pericycle vävnad som finns bredvid xylem fartyg och därför kallas xylem pole pericycle (XPP; se figur 2C)40. Vissa XPP celler, som ligger djupt inne i roten, förvärva en grundare cell identitet och, på en lokal hormonell trigger, börjar föröka sig genom svullnad och dela anticlinally41. Men på grund av närvaron av en styv cellvägg matris, utövar denna process mekanisk stress på de omgivande vävnaderna. I synnerhet påverkas den överliggande endodermis, som det är i vägen för den laterala rottillväxtaxeln42,43,44. Faktum är att den nybildande primordium måste växa genom den överliggande endodermis cellen (Figur 2C2) medan cortex och epidermis celler bara skjuts åt sidan för primordium att äntligen dyka upp45,46. Senaste arbetet i vårt labb har visat att endodermis aktivt bidrar till att tillgodose spridningen i pericycle. Riktad blockering av endodermal hormonell signalering är tillräcklig för att hämma även den allra första divisionen i XPP-cellerna47. Därför utgör pericycle-endodermis kommunikation en mycket tidig kontrollpunkt för lateral rot utveckling i Arabidopsis. Det är dock inte känt hur denna överhörning utförs. För att reda ut detta mysterium valde vi TRAP-seq-metoden för att rikta in sig på XPP och endodermala celler. För att berika för celler i det laterala rotprogrammet, härmade vi den hormonella utlösaren genom att exogent tillämpa en auxin analog (1-naftalenetiksyra, NAA)48, som samtidigt tillåtet att tidsmässigt lösa den inledande fasen av laterala rotbildning.
Protocol
Representative Results
Discussion
Verifiering av RPL18-lokaliseringsmönster
Avgörande för att undvika feltolkning av data från alla TRAP-experiment är det korrekta uttrycksmönstret för den taggade ribosomala underenheten. Därför tillåter införlivandet av GFP som en epitoptagg till RPL18 mycket elegant kontroll av det önskade uttrycksmönstret och i följd, pulldown av polysomefraktionen från samma vävnad. Mer invasiva metoder för att säkerställa korrekt promotor mönster följs av Jiao och Mayerowitz 2010, vilket krä…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi vill tacka Jean-Claude Walser från Genetic Diversity Center Zürich för viktiga expertråd i den tidiga fasen av detta projekt. Arbetet i Vermeer labbet stöddes av en SNF professur stipendium (PP00P3_157524) och en R’EQUIP utrustning bidrag (316030_164086) från Swiss National Science Foundation (SNSF) tilldelas JEMV.
Materials
| Sterilization | |||
| bleach, 13% | Sigma | 71696 | |
| beaker | VWR | 214-1172/74/75 | |
| desiccator with porcelaine plate (DURAN) | Sigma/Merck | Z317454-1EA/Z317594-1EA | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| HCl, 37% | Roth | 4625.1 | |
| Tween 20 | Sigma | P9416 | |
| Plate growth + harvesting | |||
| MS salts, basal salt mixture, incl. MES buffer | Duchefa | M0254 | |
| agar plant for cell culture | Applichem/Panreac | A2111.1000 | |
| DMSO | Sigma | D4540 | |
| forcepts | Rubis Switzerland | 5-SA model | |
| KOH | Fluka | 60370 | |
| micropore/surgical tape | 3M | 1530-0 | |
| NAA | Duchefa | N0903 | |
| petri dishes 120×120 mm | Greiner bio-one | 688102 | |
| scalpel | VWR/Swann-Morton | 233-5454 | |
| tissues, neutral, two-layered | any supplier of your choice | ||
| Immunoprecipitation | |||
| GFP-beads: gtma-100 GFP-Trap_MA | Chromotek | e.g. gtma-100 | |
| Brij-35 | Sigma | P1254-500G | |
| centrifuge tubes (in accordance with centrifuge) | Beckman Coulter | 357001 | |
| Chloramphenicol | Applichem | C0378-25G | |
| cotton gloves | VWR | 113-7355 | |
| Cycloheximide, HPLC grade | Sigma | 01810-1G | |
| DEPC | VWR | E174 | might have long delivery times |
| DTT | Fluka | 43815 | |
| EGTA | Sigma | 3054.3 | |
| homogenizers DUALL 23 | KONTES GLASS CO (via VWR) | SCERSP885450-0023 (set) | SCERSP885451-0023 pestle only – SCERSP885452-0023 cylinder only; long delivery times |
| Igepal CA-360 | Sigma | I3021-100ml | |
| KCl | Sigma | 60130 | |
| MgCl2 hexahydrat | Roth | 2189.2 | |
| mortar and pestle | VWR | 470148-960 & 470019-978 | |
| PMSF | Roche | 10 837 091 001 | |
| Polyoxyethylene-(10)-tridecylether/PTE | Sigma | P2393-500G | |
| RNase-free water | Roth | T143.3 | |
| RNAZap | Thermo Fisher | AM9780/AM9782 | for cleaning surfaces |
| Tris, >99.3% | Roth | AE15.3 | |
| Triton X-100 | Fluka | T8787-250ml | |
| Tween 20 | Sigma | P9416-100ml | |
| RNA extraction | |||
| 2-Propanol, p.a. | Sigma | 33539-1L-GL-R | |
| Chloroform, HPLC grade | Scharlau | CL02181000 | |
| EtOH, p.a. | Honeywell | 02860-1L | |
| low-retention microcentrifuge tubes, 1.5 ml | Eppendorf/Sigma | Z666548-250EA | LoBind |
| RNase-free DNase set | Qiagen | 79254 | |
| RNeasy MiniElute Cleanup Kit | Qiagen | 74204 | |
| TRIzol reagent | ThermoFisher/Ambion | 15596018 | |
| Library preparation | |||
| 15/50 mL Tube Magnetic Separator | Abraxis | PN 472250 | |
| AMPure beads | Beckman Coulter | A63881 | |
| Index Kit A | Illumina | FC-131-2001 | |
| Index Kit D | Illumina | FC-131-2004 | |
| neodymium magnets | Amazon/other | 6 x 1.5 mm range: N42 (NdFeB) | |
| Nextera XT kit | Illumina | FC-131-1024/1096 | https://emea.support.illumina.com/ |
| PCR strips | ThermoScientific | AB-0266 | |
| SMARTer v4 kit | Takara Bioscience | 634892 | https://www.takarabio.com/ |
| Bioanalyzer | Agilent | 2100 Bioanalyzer Instrument | specialized equipment for RNA/DNA quality control |
| Tapestation | Agilent | 4200 Tapestation Instrument | specialized equipment for RNA/DNA quality control |
| Fragment Analyzer | Agilent | 5400 Fragment Analyzer System | specialized equipment for RNA/DNA quality control (high throughput) |
| LabChip | PerkinElmer | LabChip GX Touch Nucleic Acid Analyzer | specialized equipment for RNA/DNA quality control (high throughput) |
| Qubit 4 Fluorometer | ThermoFisher | Q33239 | specialized equipment for RNA/DNA concentration determination |
| qRT-PCR | |||
| GATA23 | Microsynth | fwd: AGTGAGAATGAA AGAAGAGAAGGG; rev: GTGGCTGCGAAT AATATGAATACC |
|
| GH3.3 | Microsynth | fwd: CAAACCAATCCT CCAAATGAC; rev: ACTTATCCGCAA CCCGACT |
|
| LBD29 | Microsynth | fwd: TCTCCAACAACA GGTTGTGAAT; rev: AAGGAGCCTTAG TAGTGTCTCCA |
|
| UBC21 | Microsynth | fwd: TGCGACTCAGGG AATCTTCT; rev: TCATCCTTTCTT AGGCATAGCG |
|
| SsoAdvanced Universal SYBR Green | Bio-Rad | #172-5270 | |
| iScript Adv cDNA Kit | Bio-Rad | #172-5038 | |
| miscellaneous | |||
| Falcon tubes 15 ml, Cellstar | Greiner bio-one | 188261 | |
| Falcon tubes 50 ml, Cellstar | Greiner bio-one | 210261 | |
| filter tips 1 ml | Axygen | TF-1000-R-S | |
| filter tips 10 µl | Axygen | TF-10-R-S | |
| filter tips 100 µl | Axygen | TF-100-R-S | |
| filter tips 20 µl | Axygen | TF-20-R-S | |
| filter tips 200 µl | Axygen | TF-200-R-S | |
| microcentrifuge tubes 1.5 ml | SARSTEDT | 72.690.001 | |
| Propidium iodide | Sigma | P4170-100MG | |
| sequencing company | Novogene | en.novogene.com |
References
- Van Verk, M. C., Hickman, R., Corné, M. J., Pieterse, M., Van Wees, S. C. RNA-Seq: Revelation Of The Messengers. Trends In Plant Science. 18 (4), 175-179 (2013).
- Libault, M., Pingault, L., Zogli, P., Schiefelbein, J. Plant Systems Biology At The Single-Cell Level. Trends In Plant Science. 22 (11), 949-960 (2017).
- Mustroph, A., et al. Profiling Translatomes Of Discrete Cell Populations Resolves Altered Cellular Priorities During Hypoxia In Arabidopsis. Proceedings Of The National Academy Of Sciences Of The United States Of America. 106 (44), 18843-18848 (2009).
- Karve, R., Iyer-Pascuzzi, A. S. Digging Deeper: High-Resolution Genome-Scale Data Yields New Insights Into Root Biology. Current Opinion In Plant Biology. 24, 24-30 (2015).
- Warner, J. R., Knopf, P. M., Rich, A. A Multiple Ribosomal Structure In Protein Synthesis. Proceedings of The National Academy of Sciences of The United States of America. 49 (1), 122-129 (1963).
- Gautam, V., Sarkar, A. K. Laser Assisted Microdissection, An Efficient Technique To Understand Tissue Specific Gene Expression Patterns And Functional Genomics In Plants. Molecular Biotechnology. 57 (4), 299-308 (2015).
- Bargmann, B. O. R., Birnbaum, K. D. Fluorescence Activated Cell Sorting Of Plant Protoplasts. Journal of Visualized Experiments. (36), e1673 (2010).
- Deal, R. B., Henikoff, S. The Intact Method For Cell Type-Specific Gene Expression And Chromatin Profiling In Arabidopsis Thaliana. Nature Protocols. 6 (1), 56-68 (2011).
- Dougherty, J. D. The Expanding Toolkit Of Translating Ribosome Affinity Purification. The Journal of Neuroscience: The Official Journal Of The Society For Neuroscience. 37 (50), 12079-12087 (2017).
- Mustroph, A., Juntawong, P., Bailey-Serres, J. Isolation Of Plant Polysomal mRNA By Differential Centrifugation And Ribosome Immunopurification Methods. Methods in Molecular Biology. 553, 109-126 (2009).
- Matsushima, W., et al. SLAM-ITseq: Sequencing Cell Type-Specific Transcriptomes Without Cell Sorting. Development. 145 (13), (2018).
- Basnet, H., et al. Flura-Seq Identifies Organ-Specific Metabolic Adaptations During Early Metastatic Colonization. Elife. 8, (2019).
- Rodriques, S. G., et al. Slide-Seq: A Scalable Technology For Measuring Genome-Wide Expression At High Spatial Resolution. Science. 363 (6434), 1463-1467 (2019).
- Fazal, F. M., et al. Atlas Of Subcellular RNA Localization Revealed By Apex-Seq. Cell. 178 (2), 473-490 (2019).
- Slane, D., Bayer, M., Kaufmann, K., Mueller-Roeber, B. Cell Type-Specific Gene Expression Profiling Using Fluorescence-Activated Nuclear Sorting. Plant Gene Regulatory Networks: Methods And Protocols. , 27-35 (2017).
- Zanetti, M. E., Chang, I. F., Gong, F., Galbraith, D. W., Bailey-Serres, J. Immunopurification Of Polyribosomal Complexes Of Arabidopsis For Global Analysis Of Gene Expression. Plant Physiology. 138 (2), 624-635 (2005).
- King, H. A., Gerber, A. P. Translatome Profiling: Methods For Genome-Scale Analysis Of mRNA Translation. Briefings In Functional Genomics. 15 (1), 22-31 (2016).
- Mašek, T., Valášek, L., Pospíšek, M., Nielsen, H. Polysome Analysis And RNA Purification From Sucrose Gradients. RNA: Methods And Protocols. , 293-309 (2011).
- Heiman, M., et al. A Translational Profiling Approach For The Molecular Characterization Of Cns Cell Types. Cell. 135 (4), 738-748 (2008).
- Halbeisen, R. E., Scherrer, T., Gerber, A. P. Affinity Purification Of Ribosomes To Access The Translatome. Methods. 48 (3), 306-310 (2009).
- Thomas, A., et al. A Versatile Method For Cell-Specific Profiling Of Translated mRNAs In Drosophila. Plos One. 7 (7), e40276 (2012).
- Watson, F. L., et al. Cell Type-Specific Translational Profiling In The Xenopus Laevis Retina. Developmental Dynamics. 241 (12), 1960-1972 (2012).
- Lam, P. Y., Harvie, E. A., Huttenlocher, A. Heat Shock Modulates Neutrophil Motility In Zebrafish. Plos One. 8 (12), e84436 (2013).
- Fang, Y., et al. Translational Profiling Of Cardiomyocytes Identifies An Early Jak1/Stat3 Injury Response Required For Zebrafish Heart Regeneration. Proceedings Of The National Academy Of Sciences Of The United States Of America. 110 (33), 13416-13421 (2013).
- Mustroph, A., Zanetti, M. E., Girke, T., Bailey-Serres, J. Isolation And Analysis Of mRNAs From Specific Cell Types Of Plants By Ribosome Immunopurification. Methods In Molecular Biology. 959, 277-302 (2013).
- Monshausen, G. B., Gilroy, S. Feeling Green: Mechanosensing In Plants. Trends In Cell Biology. 19 (5), 228-235 (2009).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be More Specific! Laser-Assisted Microdissection Of Plant Cells. Trends In Plant Science. 10 (8), 397-406 (2005).
- Sheen, J. Signal Transduction In Maize And Arabidopsis Mesophyll Protoplasts. Plant Physiology. 127 (4), 1466-1475 (2001).
- Datta, S., et al. Laser Capture Microdissection: Big Data From Small Samples. Histology And Histopathology. 30 (11), 1255-1269 (2015).
- Birnbaum, K., et al. A Gene Expression Map Of The Arabidopsis Root. Science. 302 (5652), 1956 (2003).
- Hamant, O., Haswell, E. S. Life Behind The Wall: Sensing Mechanical Cues In Plants. BMC Biology. 15 (1), 1354 (2017).
- Vragović, K., et al. Translatome Analyses Capture Of Opposing Tissue-Specific Brassinosteroid Signals Orchestrating Root Meristem Differentiation. Proceedings of The National Academy of Sciences of the United States of America. 112 (3), 923-928 (2015).
- Wang, Y., Jiao, Y. Translating Ribosome Affinity Purification (Trap) For Cell-Specific Translation Profiling In Developing Flowers. Methods In Molecular Biology. 1110, 323-328 (2014).
- Sablok, G., Powell, J. J., Kazan, K. Emerging Roles And Landscape Of Translating mRNAs In Plants. Frontiers in Plant Science. 8, 1443 (2017).
- Ron, M., et al. Hairy Root Transformation Using Agrobacterium Rhizogenes As A Tool For Exploring Cell Type-Specific Gene Expression And Function Using Tomato As A Model. Plant Physiology. 166 (2), 455-469 (2014).
- Reynoso, M. A., et al. Evolutionary Flexibility In Flooding Response Circuitry In Angiosperms. Science. 365 (6459), 1291-1295 (2019).
- Dolan, L., et al. Cellular Organisation Of The Arabidopsis Thaliana Root. Development. 119 (1), 71 (1993).
- Ristova, D., Barbez, E. . Root Development. , (2018).
- Shekhar, V., Stӧckle, D., Thellmann, M., Vermeer, J. E. M. The Role Of Plant Root Systems In Evolutionary Adaptation. Current Topics in Developmental Biology. 131, 55-80 (2019).
- Malamy, J. E., Benfey, P. N. Down And Out In Arabidopsis: The Formation Of Lateral Roots. Trends in Plant Science. 2 (10), 390-396 (1997).
- de Smet, I., et al. Bimodular Auxin Response Controls Organogenesis In Arabidopsis. Proceedings of the National Academy of Sciences of The United States of America. 107 (6), 2705-2710 (2010).
- Péret, B., et al. Arabidopsis Lateral Root Development: An Emerging Story. Trends In Plant Science. 14 (7), 399-408 (2009).
- Vilches-Barro, A., Maizel, A. Talking Through Walls: Mechanisms Of Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 23, 31-38 (2015).
- Porco, S., et al. Lateral Root Emergence In Arabidopsis Is Dependent On Transcription Factor Lbd29 Regulation Of Auxin Influx Carrier Lax3. Development. 143 (18), 3340-3349 (2016).
- Stoeckle, D., Thellmann, M., Vermeer, J. E. Breakout-Lateral Root Emergence In Arabidopsis Thaliana. Current Opinion in Plant Biology. 41, 67-72 (2018).
- Banda, J., et al. Lateral Root Formation In Arabidopsis: A Well-Ordered Lrexit. Trends in Plant Science. 24 (9), 826-839 (2019).
- Vermeer, J. E. M., et al. A Spatial Accommodation By Neighboring Cells Is Required For Organ Initiation In Arabidopsis. Science. 343 (6167), 178-183 (2014).
- Vanneste, S., et al. Cell Cycle Progression In The Pericycle Is Not Sufficient For Solitary Root/Iaa14-Mediated Lateral Root Initiation In Arabidopsis Thaliana. The Plant Cell. 17 (11), 3035-3050 (2005).
- Marques-Bueno, M. M., et al. A Versatile Multisite Gateway-Compatible Promoter And Transgenic Line Collection For Cell Type-Specific Functional Genomics In Arabidopsis. The Plant Journal : For Cell and Molecular Biology. 85 (2), 320-333 (2016).
- Shimada, T. L., Shimada, T., Hara-Nishimura, I. A Rapid And Non-Destructive Screenable Marker, Fast, For Identifying Transformed Seeds Of Arabidopsis Thaliana. The Plant Journal : For Cell and Molecular Biology. 61 (3), 519-528 (2010).
- Clough, S. J., Bent, A. F. Floral Dip: A Simplified Method For Agrobacterium Mediated Transformation Of Arabidopsis Thaliana. The Plant Journal. 16 (6), 735-743 (1998).
- Lindsey, B. E., Rivero, L., Calhoun, C. S., Grotewold, E., Brkljacic, J. Standardized Method For High-Throughput Sterilization Of Arabidopsis Seeds. Journal Of Visualized Experiments. (128), e56587 (2017).
- Andersen, T. G., et al. Diffusible Repression Of Cytokinin Signalling Produces Endodermal Symmetry And Passage Cells. Nature. 555, 529-533 (2018).
- Schroeder, A., et al. The Rin: An Rna Integrity Number For Assigning Integrity Values To Rna Measurements. BMC Molecular Biology. 7, 3 (2006).
- Vragović, K., Bartom, E., Savaldi-Goldstein, S. Quantitation Of Cell Type-Specific Responses To Brassinosteroid By Deep Sequencing Of Polysome-Associated Polyadenylated RNA. Methods in Molecular Biology. 1564, 81-102 (2017).
- Bertin, B., Renaud, Y., Aradhya, R., Jagla, K., Junion, G. Trap-Rc, Translating Ribosome Affinity Purification From Rare Cell Populations Of Drosophila Embryos. Journal Of Visualized Experiments. (103), e52985 (2015).
- Livak, K. J., Schmittgen, T. D. Analysis Of Relative Gene Expression Data Using Real-Time Quantitative PCR And The 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Jiao, Y., Meyerowitz, E. M. Cell-Type Specific Analysis Of Translating Rnas In Developing Flowers Reveals New Levels Of Control. Molecular Systems Biology. 6, 419 (2010).
- Tian, C., et al. A Gene Expression Map Of Shoot Domains Reveals Regulatory Mechanisms. Nature Communications. 10 (1), 141 (2019).
- Townsley, B. T., Covington, M. F., Ichihashi, Y., Zumstein, K., Sinha, N. R. Brad-Seq: Breath Adapter Directional Sequencing: A Streamlined, Ultra-Simple And Fast Library Preparation Protocol For Strand Specific mRNA Library Construction. Frontiers in Plant Science. 6, 366 (2015).
- Song, Y., et al. A Comparative Analysis Of Library Prep Approaches For Sequencing Low Input Translatome Samples. BMC Genomics. 19 (1), 696 (2018).
- Basu, D., Haswell, E. S. Plant Mechanosensitive Ion Channels: An Ocean Of Possibilities. Current Opinion in Plant Biology. 40, 43-48 (2017).
- Brady, S. M., et al. A High-Resolution Root Spatiotemporal Map Reveals Dominant Expression Patterns. Science. 318 (5851), 801 (2007).

