Лазер-Захват Микродиссеция РНК-секвенирования для пространственной и временной ткани-специфический анализ экспрессии генов в растениях
Summary
Здесь представлен протокол для лазерного захвата микродиссекции (LCM) тканей растений. LCM является микроскопическим методом для изоляции областей ткани в без загрязнения образом. Процедура включает фиксацию тканей, встраивание парафина, секциоирование, LCM и экстракцию РНК. РНК используется в ниже по течению ткани конкретных, временно решенный анализ транскриптомов.
Abstract
Развитие сложного многоклеточного организма регулируется отдельными типами клеток, которые имеют различные транскрипционные профили. Для выявления транскрипционных регуляторных сетей, регулирующих процессы развития, необходимо измерить пространственные и временные профили экспрессии генов этих отдельных типов клеток. Таким образом, понимание пространственно-временного контроля экспрессии генов имеет важное значение для получения понимания того, как регулируются биологические процессы и процессы развития. Здесь мы описываем метод микродиссекции лазерного захвата (LCM), чтобы изолировать небольшое количество клеток из трех органов эмбриона ячменя в течение времени во время прорастания с последующим профилированием стенограммы. Метод состоит из фиксации тканей, обработки тканей, встраивания парафина, секций, LCM и извлечения РНК с последующим ПЦР или РНК-сек в режиме реального времени. Этот метод позволил нам получить пространственные и временные профили транскрипций органов семян из различных количеств клеток (десятки и сотни), обеспечивая гораздо большую специфичность тканей, чем типичные анализы навалочных тканей. На основе этих данных мы смогли определить и сравнить транскрипционные регуляторные сети, а также предсказать факторы транскрипции кандидатов для отдельных тканей. Метод должен применяться к другим тканям растений с минимальной оптимизацией.
Introduction
Развитие и рост растений связаны с скоординированным действием транскрипционных регуляторных сетей в различных клетках, которые существуют в сложной клеточной среде. Чтобы понять активность этих регулятивных сетей, нам необходимы знания пространственной и временной экспрессии генов в различных типах клеток на разных стадиях развития. Однако анализ экспрессии генов чаще проводится в образцах целых органов или навалочных тканей из-за технической проблемы изоляции и анализа небольшого количества клеток. Метод, который мы описываем здесь, позволил получить анализ транскриптома пространственной и височной ткани путем соединения LCM с РНК-сек.
LCM был разработан два десятилетия назад Эммерт-Бак и его коллеги1. Техника позволила исследователям точно изолировать однокамеры или кластеры клеток из окружающей среды с помощью прямой микроскопической визуализации и манипуляции с помощью узкого лучалазера 1. С тех пор метод широко используется в биологии рака и патологии2,3. Многие исследовательские группы растений также адаптировали LCM для использования с различными видамирастений и различными типами тканей 4,,5,,6,,7,,8,,9,,10,,11. В последнее время несколько работ также использовали LCM на eudicot и семена монокота для изучения эмбрионов, эндоспермов и других структур семян во время развития семяни прорастания 10,12,13. Большинство других широко используемых одноклеточных методов изоляции, таких как микро-пипеттинг, сортировка клеток, магнитное разделение и микрофлюидные платформы, зависят от энзиматического пищеварения или механической гомогенизации для диссоциации клеток. Это может возмутить экспрессию генов, вводя технические артефакты, которые путаютинтерпретацию данных 14,,15. Эти методы также требуют, чтобы предыдущие знания маркерных генов для каждого типа клеток соотносить диссоциированные клетки с их пространственным расположением и истинным типом клеток. Дальнейшая группа методов зависит от сродства на основе изоляции подклеточных структур, а не целые клетки, например INTACT (Изоляция ядер, отмеченных в типах клеток) и TRAP (Перевод Рибосома Аффинити Очистка)16,17. Тем не менее, сродство маркировки и очистки ядер или рибосом являются технически сложными в видов растений, которые не имеют устоявшихся протоколов трансформации. LCM использует быструю фиксацию тканей для сохранения уровней транскрипции и обычной гистологической идентификации путем прямой визуализации клеток в пределах их нормальной ткани / органа контексте, что позволяет дискретных клеток, которые будут изолированы в течение короткогопериода времени 18,19.
Протокол, представленный здесь, представляет 100-й метод изоляции конкретных клеток или типов клеток от тканевых секций семян зерновых, которые могут быть применены к большинству клеток, которые могут быть гисто логически идентифицированы. LCM предоставляет бесконтактный метод изоляции клеток, значительно снижая загрязнение и повышая целостность восстановленной РНК. Кроме того, метод иллюстрирует силу LCM на крупномасштабных исследованиях генома, начиная с небольших количеств биологических материалов. Мы также описываем линейное усиление РНК для генерации достаточного входного материала для анализа стенограммы/транскриптома ниже по течению.
Есть десять основных шагов в этом LCM РНК-сек протокол для пространственной и височной ткани конкретных транскриптомов, в том числе фиксации образцов тканей, обезвоживание, парафин инфильтрации, встраивания, секционирования, LCM, РНК экстракции, РНК усиления, РНК количественной оценки и qRT-PCR и / или РНК-сек (Рисунок 1).
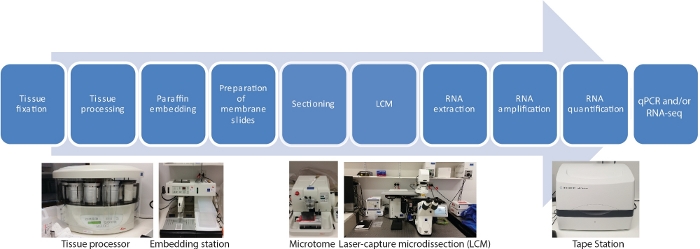
Рисунок 1: Flowchart LCM следуют РНК-сек или qRT-PCR. LCM является пространственно точным и бесконтактной техникой для сбора клеток из фиксированных секций тканей с помощью лазерного луча под микроскопической визуализацией. Процесс начинается с фиксации образцов тканей, а затем обезвоживания с использованием градиента серии этанола и ксилена, и закончил с парафином инфильтрации. Процесс может быть полностью автоматизирован с помощью процессора ткани. После того, как ткань проникли с парафином, он встроен в форму с расплавленным парафином с помощью встраивания станции. Раздел осуществляется с использованием микротома, установленного до нужной толщины. Слайды готовятся и LCM проводится непосредственно перед РНК должна быть извлечена из захваченных клеток. Добыча РНК сопровождается непосредственно двумя раундами усиления РНК до qRT-PCR и/или РНК-сек. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
Protocol
Representative Results
Discussion
Многие исследования экспрессии генов, специфичные для тканей, были ограничены вскрытием образцов вручную, что является трудоемким, трудоемким, имеет высокий риск заражения и может использовать только образцы, которые оперативник человека достаточно ловкий, чтобы собрать урожай. LCM яв?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Эта работа была поддержана Австралийский научно-исследовательский совет Центр передового опыта в области биологии энергии растений (CE140100008) в JW. M.G.L была поддержана стартовым грантом Университета Ла Троб. Мы благодарим платформу La Trobe Genomics platform за поддержку в последовательности с высокой пропускной способностью и анализе данных. Мы благодарим доцента Мэтью Такера за экспертные советы по созданию LCM в нашей лаборатории.
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
References
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

