Laser-Capture Microdissection RNA-sekvensering för rumsliga och temporala vävnadsspecifika genuttryck analys i växter
Summary
Presenteras här är ett protokoll för laser-capture microdissection (LCM) av växtvävnader. LCM är en mikroskopisk teknik för att isolera områden av vävnad på ett kontaminationsfritt sätt. Förfarandet omfattar vävnad fixering, paraffin inbäddning, snittning, LCM och RNA extraktion. RNA används i den nedströms vävnadsspecifika, tidsmässigt löst analys av transkriptomer.
Abstract
Utvecklingen av en komplex flercellig organism styrs av distinkta celltyper som har olika transkriptionella profiler. För att identifiera transkriptionella regleringsnätverk som styr utvecklingsprocesser är det nödvändigt att mäta dessa enskilda celltypers rumsliga och tidsmässiga genuttrycksprofiler. Därför är insikt i den spatio-temporala kontrollen av genuttryck avgörande för att få förståelse för hur biologiska och utvecklingsmässiga processer regleras. Här beskriver vi en laser-capture microdissection (LCM) metod för att isolera litet antal celler från tre korn embryo organ under en tid-kurs under grobarhet följt av avskrift profilering. Metoden består av vävnadsfixering, vävnadsbearbetning, paraffininbäddning, snittning, LCM- och RNA-extraktion följt av realtids-PCR eller RNA-seq. Denna metod har gjort det möjligt för oss att få rumsliga och tidsmässiga profiler av utsäde organ transkriptomer från varierande antal celler (tiotals till hundratals), vilket ger mycket större vävnad-specificitet än typiska bulk-vävnad analyser. Från dessa data kunde vi definiera och jämföra transkriptionella regulatoriska nätverk samt förutsäga kandidatreglerande transkriptionsfaktorer för enskilda vävnader. Metoden bör vara tillämplig på andra växtvävnader med minimal optimering.
Introduction
Växtutveckling och tillväxt innebär en samordnad åtgärd av transkriptionella regleringsnätverk inom olika celler som finns i en komplex cellulär miljö. För att förstå aktiviteten i dessa regleringsnätverk kräver vi kunskap om rumsliga och tidsmässiga genuttryck inom olika celltyper över utvecklingsstadier. Analyser av genuttryck utförs dock oftare i hela organ eller massvävnadsprover på grund av den tekniska utmaningen att isolera och analysera ett litet antal celler. Den metod vi beskriver här har tillåtit erhålla rumsliga och tidsmässiga vävnadsspecifika transkriptom analys genom koppling LCM med RNA-seq.
LCM utvecklades för två decennier sedan av Emmert-Buck och kollegor1. Tekniken gjorde det möjligt för forskare att exakt isolera enceller eller kluster av celler från sin omgivning med hjälp av direkt mikroskopisk visualisering och manipulation med en smal strållaser1. Sedan dess har metoden använts flitigt inom cancerbiologi och patologi2,3. Många växtforskningsgrupper har också anpassat LCM för användning med olika växtarter och olika vävnadstyper4,5,6,7,8,9,10,11. Nyligen har flera papper också använt LCM på eudikot och monocot frön att studera embryo, frövita och andra strukturer utsäde under utsäde utveckling och grobarhet10,12,13. De flesta av de andra vanligt förekommande encelliga isoleringsmetoderna som mikro-pipettering, cellsortering, magnetisk separation och mikrofluidiska plattformar är beroende av den enzymatiska matsmältningen eller mekanisk homogenisering för att dissociate celler. Detta kan genomsyra genuttryck, införa tekniska artefakter som förvirrar datatolkning14,15. Dessa metoder kräver också tidigare kunskaper om markörgener för varje celltyp för att relatera de dissocierade cellerna till deras rumsliga placering och sanna cell-typ. Ytterligare en grupp tekniker beror på affinitetsbaserad isolering av subcellulära strukturer istället för hela celler, till exempel INTAKT (Isolering av kärnor Taggade i celltyper) och TRAP (Translating Ribosome Affinity Purification)16,17. Affinitetsmärkning och rening av atomkärnor eller ribosomer är dock tekniskt utmanande hos växtarter som inte har väletablerade transformationsprotokoll. LCM drar fördel av snabb vävnad fixering för att bevara transkription nivåer och konventionella histologiska identifiering genom direkt visualisering av celler inom deras normala vävnad / organ sammanhang, som gör att diskreta celler isoleras på kort tid18,19.
Det protokoll som presenteras här är en optimerad metod för isolering av specifika celler eller celltyper från vävnadssektionerna i spannmålsfrön, som kan appliceras på de flesta av de celler som kan histologiskt identifieras. LCM ger en kontaktfri metod för cellisolering, kraftigt minska kontaminering och öka integriteten av återvunnet RNA. Vidare illustrerar metoden LCM:s kraft på storskaliga genomsvida studier som börjar med små mängder biologiska material. Vi beskriver också linjär förstärkning av RNA för att generera tillräckligt med inmatningsmaterial för nedströms avskrift/transkriptomanalyser.
Det finns tio huvudsteg i detta LCM RNA-seq-protokoll för rumsliga och tidsmässiga vävnadsspecifika transkriptomer, inklusive fixering av vävnadsprover, uttorkning, paraffinininfiltration, inbäddning, snittning, LCM, RNA-extraktion, RNA-amplifiering, RNA-kvantifiering och qRT-PCR och/eller RNA-seq (Figur 1).
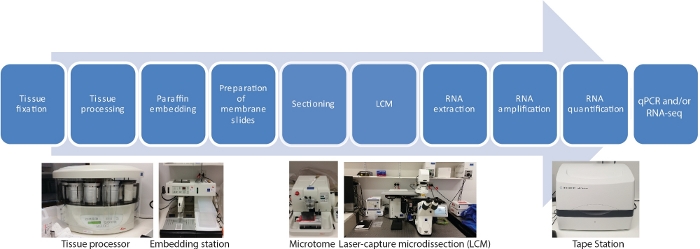
Bild 1: Flödesschema av LCM följt av RNA-seq eller qRT-PCR. LCM är en rumsligt exakt och kontaktfri teknik för att samla in celler från fasta vävnadssektioner med hjälp av en laserstråle under mikroskopisk visualisering. Processen börjar med fixering av vävnadsprover, följt av uttorkning med hjälp av en gradient serie av etanol och xylen, och slutade med paraffin infiltration. Processen kan automatiseras helt genom att använda en vävnadsprocessor. När vävnaden är infiltrerad med paraffin, är det inbäddat i en form med smält paraffin med hjälp av en inbäddningsstation. Snittning utförs med hjälp av mikrotom inställd på önskad tjocklek. Diabilder bereds och LCM genomförs omedelbart innan RNA ska extraheras från fångade celler. RNA-extraktion följs direkt av två omgångar av RNA-amplifiering före qRT-PCR och/eller RNA-seq. Vänligen klicka här för att visa en större version av denna figur.
Protocol
Representative Results
Discussion
Många vävnadsspecifika genuttryck studier har begränsats genom hand dissekering av prover, som är tidskrävande, arbetsintensiva, har en hög risk för kontaminering och kan bara utnyttja prover som en mänsklig operativ är tillräckligt fingerfärdig att skörda. LCM är en exakt och kontaktfri teknik för att samla in celler från fasta vävnadssektioner med hjälp av en mekaniskt manövrerad laserstråle under mikroskopisk visualisering.
Bra provpreparering är avgörande för LCM. Pro…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Detta arbete stöddes av Australian Research Council Centre of Excellence in Plant Energy Biology (CE140100008) till JW. M.G.L stöddes av ett la Trobe-universitet som började ge bidrag. Vi tackar La Trobe Genomics-plattformen för deras stöd inom sekvensering med hög genomströmning och dataanalys. Vi tackar docent Matthew Tucker för expertråd om att etablera LCM i vårt labb.
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
References
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

