オークリッジ国立研究所の高フラックス同位体原子炉における中性子ラジオグラフィーと生体システムのコンピューター断層撮影
Summary
この原稿では、高フラックス同位体リアクター(HFIR)CG-1Dビームラインを使用して、ラット大腿骨、マウス肺、および草本植物の根/土壌システムの金属インプラントを測定するための中性子ラジオグラフィーおよび生体サンプルのコンピューター断層撮影のプロトコルについて説明しています。
Abstract
中性子は歴史的に、小角中性子散乱、中性子スピンエコー、回折、非弾性散乱などの技術を採用した幅広い生物学的用途に使用されてきました。逆数空間で情報を得る中性子散乱技術とは異なり、減衰ベースの中性子イメージングは、数十マイクロメートルオーダーで分解された実空間で信号を測定します。中性子イメージングの原理は、ランベルトベールの法則に従い、サンプルを通るバルク中性子減衰の測定に基づいています。より大きな減衰は、生物学的サンプルの主要な成分であるいくつかの軽元素(特に水素)によって示されます。重水素、ガドリニウム、またはリチウム化合物などの造影剤は、光学画像、磁気共鳴画像法、X線、陽電子放出断層撮影法などの技術を含む医用画像で行われるのと同様の方法でコントラストを高めるために使用できます。生物学的システムでは、地下の植物の根ネットワークの複雑さ、土壌との相互作用、およびその場での水フラックスのダイナミクスを調査するために、中性子ラジオグラフィーとコンピューター断層撮影がますます使用されています。さらに、軟部組織や骨などの動物サンプルのコントラストの詳細を理解するための努力が検討されています。本稿では、試料調製、計測、データ取得戦略、高フラックス同位体炉CG-1D中性子イメージングビームラインを用いたデータ解析など、中性子バイオイメージングの進歩に焦点を当てています。前述の能力は、植物生理学(草本植物/根/土壌系)および生物医学的用途(ラット大腿骨およびマウス肺)における例の選択を使用して説明されます。
Introduction
中性子ラジオグラフィー(nR)の原理は、中性子が通過する物質を介した中性子の減衰に基づいています。原子の電子雲によって散乱されるX線とは異なり、中性子はその原子核によって吸収または散乱することができます。中性子は水素(H)などの軽元素に敏感であり、その結果、動物1,2,3,4,5,6,7またはヒト組織8,9および地下土壌/根系10,11,12,13,14などの生物学的用途をX線撮影するために使用できます。、15。中性子イメージングは、重元素を検出することができるX線イメージングを補完する技術です16,17,18。減衰ベースのnRは、透過ビームが材料の量と材料を通る経路長に正比例するというベール・ランベルトの法則で説明されているように、サンプル内の材料の線形減衰係数とサンプルの厚さによって支配されます。したがって、透過率Tは次のように計算できます。
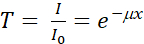 (1)
(1)
ここで、I 0およびIは、それぞれ、入射ビーム強度および透過ビーム強度である。μとxはそれぞれ線形減衰係数と均質なサンプルの厚さです。減衰係数μ次式で与えられます。
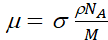 (2)
(2)
ここで、σはサンプルの中性子減衰断面積(散乱と吸収の両方)、ρはその密度、NA はアボガドロ数、Mはそのモル質量です。
低エネルギー中性子(すなわち、0.5eV未満のエネルギー)を用いた生物学的試料のX線撮影におけるコントラストは、主にHの密度の変化によるものである(一定の試料厚さの場合)。これは、中性子とH原子核との相互作用の確率が生物学的サンプルに存在する他の原子核よりも大きいこと、およびH原子の密度が生物学的サンプル中に最も豊富な原子であるため最も重要であるという事実によるものです。
初期の段階から、nRおよび中性子コンピュータ断層撮影(nCT)は、材料および工学用途に広く使用されてきました19,20,21,22,23。生物学的サンプル中のHに対する中性子感受性の最初の実証実験は、1950年代半ばに始まりました24植物標本の測定から。この研究は、例えば、酸化ガドリニウム(Gd2O3)などの造影剤の使用が探求されたヒト胸部25またはラット26のX線撮影で1960年代まで続いた。さらに、ヒト腫瘍組織と正常組織のコントラストは、H含有量の局所的な増加によるものであるという仮説が立てられました。これらの最初の試験では、中性子束と空間分解能の増加によりnRの品質が向上し、産業または生物医学的用途の補完的な技術としての人気が高まる可能性が高いと結論付けられました。最新の研究は、生物医学的および法医学的用途のために、癌組織標本1および動物臓器の切片2、3、27に対して実施されたnRおよびnCT測定で構成されています。
テネシー州オークリッジのオークリッジ国立研究所にある高フラックス同位体原子炉(HFIR)は、核分裂反応によって中性子を生成する強力な中性子源です。これらの中性子は2MeV程度のエネルギーを持ち、重水との運動反応によって原子炉プール内で「冷却」され、100〜300eVのオーダーのエネルギーに達します。散乱であれイメージングであれ、中性子実験の最適化は、中性子源とそのビーム強度、エネルギー分布、バックグラウンド(高速中性子、遅延中性子、ガンマ線)の影響などのビームラインの特性を理解することから始まります。イメージングビームラインが配置されているHFIRコールドガイドホールでは、液体H減速材との運動学的相互作用によって中性子がさらに「冷却」されます。その後、それらは線源の視線から離れた湾曲したガイドシステムで輸送され、高速中性子とガンマ汚染を排除します。図1に示すように、CG-1D中性子イメージングビームライン28,29はコールドガイド上に置かれており、中性子エネルギー範囲は数meVから数十eV(この場合、対応する使用可能な中性子波長は0.8〜10Åの範囲)の範囲で変化し、磁束は107 n /(cm2∙s)をサンプル位置に置く。電動アパーチャ/ディフューザーシステムは、イメージング機器のピンホール形状を定義します。中性子は、両端にアルミニウム(Al)窓を備えたヘリウム(He)で満たされた飛行管内で6.59mの距離を移動します。フライトチューブは、ビーム強度の損失が最小限になるように空気散乱を制限しながら中性子を輸送するために使用されます。この原稿に記載されている測定のために、ディフューザーは、Al容器に包まれた厚さ1mmの50nmの酸化アルミニウム(Al2O3)ナノ粉末でできている。ディフューザーは、中性子ガイド(イメージングビームラインのピンホール形状によって拡大される)から来るビームアーチファクトを低減し、そうでなければ、X線写真に鋭い水平および垂直強度の変動が見られ、データの正規化が困難になります。 ここに示す実験では、厚さ25μmのフッ化リチウム6リチウム/硫化亜鉛蛍光体(6LiF/ZnS:Ag)を使用して中性子を光に変換します。
コリメーションの最適化は、サンプルから検出器までの位置、必要な空間分解能、および取得時間によって異なります。サンプルがシンチレータから数cm離れた場所にある場合、高いコリメーション(L / Dが800を超える場合、Lは直径Dのピンホール開口部と検出器からの距離)により、中性子束を犠牲にして空間分解能が向上します。低いコリメーション(L/D 800未満)は、時間分解能が空間分解能よりも優先される場合のin situダイナミックスタディに適しています。本稿で紹介した測定では、L/Dと空間分解能はそれぞれ約355μmと75μmでした。時間分解能は、信号対雑音比(SNR)に基づいて変化しました。サンプルは、ぼやけなどの幾何学的歪みを低減するために、シンチレータにできるだけ近づけて配置しました。平行移動ステージと回転ステージを使用して、サンプルを検出器の近くにセットし、コンピューター断層撮影(CT)を実行できます。CG-1Dは、画素ピッチ13.5μmの2048画素×2048画素の電荷結合素子(CCD)、画素ピッチ6.5μmの2560画素×2160画素の科学用相補型金属酸化膜半導体(sCMOS)検出器、画素サイズ55μmの512画素×512画素のマイクロチャンネルプレート(MCP)検出器30,31の3種類の検出器を提供しています。散乱した中性子は~5mm厚のホウ素ゴムで吸収され、検出器チップが中性子を見るのを防ぎます。この吸収は、ホウ素ゴムと検出器の間に配置された鉛(Pb)によって止めることができるガンマ線を生成します。各検出器は、異なる視野(FOV)と空間分解能、時間分解能に最適化されています。ラット大腿骨とマウス肺の測定には、CCD検出器がその大きなFOV能力(~7 cm x 7 cm)と約75μmの合理的な空間分解能のために利用されました。植物の根/土壌システムのnCTは、FOV(~5 cm x 4.2 cmに制限)を犠牲にしてできるだけ早くnCTを取得することが目標であったため、sCMOSで実行されました。したがって、空間分解能は明らかに損なわれました。これらの検出器では、中性子は検出目的で光またはアルファ粒子に変換されます。サンプルを垂直軸を中心に回転させ、連続した回転角度でX線写真を取得することで、nCTを取得できます。調査中のサンプルの3次元体積レンダリングモデルは、社内のiMARS3D PythonベースのJupyterフィルターバックプロジェクション(FBP)ノートブック、pyMBIR、または商用ソフトウェアを利用して取得されます。
最後に、試料や検出器と相互作用していない中性子は、バックグラウンドノイズを最小限に抑えるために、検出器システムから約1m下流のビーム停止位置に収集されます。CG-1Dビームストップは、幅0.75m、高さ0.5m、厚さ35mmで、エポキシのB4Cで作られています。ビームストップは、中性子ビームが当たる耐火エポキシの10mmの95%濃縮炭酸リチウム(6 Li2CO3)で補強され、6Li、鉛(Pb)、および鋼で裏打ちされた空洞は、高率の二次ガンマ線を含むように設計されています。ビームストップは、ビームラインの鋼製シールド壁に直接取り付けられています。CG-1Dビームラインの写真を図2に示します。
3つの再構成ソフトウェアを使用して、3つの実験データをそれぞれ3Dで再構築しました。マウス肺サンプル再構成は、FBPを利用する市販の再建ソフトウェアであるOctopus32を用いて行った。OctopusソフトウェアはサーバーPC上に設置され、ビームラインで収集されたデータを再構築するために使用できます。iMARS3Dという名前の再構成ソフトウェアは、CG-1Dで入手できます。これは、オープンソースコードTomoPY33に基づいており、自動傾斜補正、後処理フィルタなどの機能が追加されています。iMARS3Dには、データの前処理(バックグラウンドとノイズの減算)、トリミング、メディアンフィルタリング(ガンマストライクとドット抜けの補正)、自動ビーム強度変動補正、サンプルチルト補正が含まれます。シノグラムが作成されたら、リングアーチファクトの除去や平滑化などのさらなるデータ処理がオプションです。再構築のさまざまなステップは解析サーバーに保存され(後で提案の共有フォルダーに移動されます)、最終的な2Dスライスはすぐに提案の共有フォルダーに保存されます。ラット大腿骨はiMARS3Dを用いて再建した。植物の根/土壌サンプルは、TomoPYを使用してデータを中央値フィルタリングし、その後PythonのSciPyライブラリを使用して傾斜軸補正することによって前処理されました。 再構成は、ベースラインFBPから高度なモデルベースの反復再構成技術35までの一連の断層撮影アルゴリズムを実装する、社内で開発されたPythonパッケージを使用して実行されました-pyMBIR(ASTRAツールボックス34のカーネルを使用して構築)非常にまばらでノイズの多い中性子データセットから高品質の再構成を取得できます。前述の再構築ツールに基づいてレンダリングされたすべてのボリュームは、減衰コントラストで表されます。すべての視覚化は、市販の視覚化、セグメンテーション、およびデータ分析ソフトウェアパッケージAMIRA36を使用して実行されました。
本稿は、HFIR CG-1Dビームラインにおける中性子イメージング(nRおよびnCT)の利用手順を実証することを目的としています。この研究はまた、生物学的サンプル、特にマウスの肺、ラットの骨、および植物の根/土壌システムに対する現在の最先端のnRおよびnCT機能を示しています。マウスの肺は、肺組織を測定するための中性子の相補性を説明するために選択されましたが、X線は主に骨に敏感です。ラット大腿骨である骨サンプルにはチタン(Ti)インプラントがあり、骨と金属のコントラストと、骨と金属の界面を見る機会がありました(金属はそれらを強く減衰させるため、X線で測定することは困難です4)。最後に、植物の根水システムは、その場で根/土壌システムを測定するためのnCTの3次元(3D)機能を示しています。さらに、生物学的サンプルにnRを使用することの利点/欠点を示します。明らかに、この方法は、植物の根系における水動態を測定するために安全に使用できるが、放射線被曝に関連するリスクのために生きた動物またはヒトの画像技術と見なすことができず、したがって、研究を(死んだ)マウスまたは病理学のような測定のいずれかに限定し、例えば、組織サンプルを患者(動物またはヒト)から切除し、中性子ビームで測定する前に固定することによって調製する。
Protocol
Representative Results
Discussion
生体試料の中性子ラジオグラフィーとCTは、X線イメージングや磁気共鳴イメージングを補完する有望なイメージング技術です。生物学的試料の中性子イメージング実験を行う際の重要なステップは、その準備とビームラインでの封じ込めに関連しています。実験の最適化は、答えられるべき科学的な質問によって推進されます。科学の問題が現象を観察するために高い空間分解能を必要とする場合、長い取得時間が必要であり、nCT(cmサイズの視野を持つ)の欠点は、スキャンの実行に数時間かかることです。これは主に、X線CTスキャンが数mm2 の視野で数秒から数分かかるシンクロトロン源と比較して、原子炉で利用可能な全体的な中性子束の違いによるものです。この方法は、動物から抽出された生体外組織サンプルに適用できますが、放射線被曝のリスク(中性子によって生成されるガンマ線やサンプル中の原子との中性子相互作用など)のために、生体内で生きた動物や人間に拡張することはできません。ただし、水分吸収動態などの植物の根/土壌相互作用(図7)のイメージングに適しています。
植物のダイナミクスに高速nCTを使用する利点は、X線CTとは異なり、水中のHに対する感度と植物への放射線損傷がないことです。さらに、ラット大腿骨などの骨/金属サンプルでは、金属が周囲の組織に比べて比較的透明であるため(図5)、X線CT39によって誘発される金属アーチファクトを回避できる可能性があります。マウス肺(図6)などの動物組織は、中性子がHに敏感であるため、軟組織構造の印象的な検出を示しますが、空間分解能はこれらの測定のやや制限要因です。対照は、生物学的試料19、39中に存在するH原子によって提供される。
中性子格子干渉法などの新しい技術の進歩と空間分解能の向上(数ミクロンが最近報告されています42,43)により、中性子イメージングは、空間分解能が向上した生物学的組織にさらに新しいコントラストメカニズムを提供する可能性があります。より高エネルギーの中性子の探査(厚いサンプルの測定を可能にするため)は、無傷のマウスなどの動物組織のより大きな切片を測定する能力も約束し、生物医学研究にさらに新しい可能性を提供します。
Disclosures
The authors have nothing to disclose.
Acknowledgements
この研究の一部は、ORNLが運営し、UT-Battelle, LLCとの契約DE-AC05-00OR22725に基づいて、米国エネルギー省科学局、ユーザー施設が後援する高フラックス同位体原子炉のリソースを利用しました。 この研究の一部は、ユージンウィグナー特別スタッフフェローシッププログラムを通じてORNLによってサポートされました。この研究は、DOE科学局、生物環境研究局によっても後援されました。ラット大腿骨サンプルは、NIH(R01AR066562)および整形外科研究教育財団-スミスアンドネフュー賞から得た資金で、ラッシュ大学医療センターのリックサムナー博士と共同で実施された実験から得られました。チームは、中性子散乱ビームラインの使用を可能にしたHFIRサポートチームに感謝したいと思います。
Materials
| Aluminum containers | custom | Made from aluminum plates or tubing (alternate is quartz), plant and mouse sample | |
| Aluminum foil | Fisher | 01-213-100 | Mouse lung sample containment |
| Deionized water or deuterium oxide | Water or D2O can be used to enhance contrast, plant sample | ||
| Ethanol | Fisher | 04-355-223 | Mouse lung sample |
| Gauze sponges | CardinalHealth | Fully submerged in phosphate-buffered saline (PBS) and used to wrap samples, rat femur sample | |
| Growth chamber | Conviron | A1000 | Any growth chamber or greenhouse with controlled conditions would work, plant sample |
| Laboratory balance | Weighing plant system can be used to measure actual water content in the soils, plant sample | ||
| Pure silica sand | US Silica Co. | Flint#13 | Pure SiO2 provides low neutron attenuation compared to soils, plant sample |
| Sprague-Dawley Rats | Harlan | Order Code: 002-US | Rat femur sample |
| Titanium Rod | Goodfellow | TI007905 | Rat femur sample |
References
- Cekanova, M., Donnell, R., Bilheux, H., Bilheux, J. -. C. Neutron imaging: Detection of cancer using animal model. Proceedings of the 2014 Biomedical Sciences and Engineering Conference – 5th Annual ORNL Biomedical Sciences and Engineering Conference: Collaborative Biomedical Innovations – The Multi-Scale Brain: Spanning Molecular, Cellular, Systems, Cognitive, Behaviour. , (2014).
- Bilheux, H. Z., et al. Neutron imaging a. The Oak Ridge National Laboratory: Application to biological research. Proceedings of the 2014 Biomedical Sciences and Engineering Conference – 5th Annual ORNL Biomedical Sciences and Engineering Conference: Collaborative Biomedical Innovations – The Multi-Scale Brain: Spanning Molecular, Cellular, Systems, Cognitive, Behaviour. , (2014).
- Bilheux, H. Z., et al. A novel approach to determine post mortem interval using neutron radiography. Forensic Science International. 251, (2015).
- Isaksson, H., et al. Neutron tomographic imaging of bone-implant interface: Comparison with X-ray tomography. Bone. 103, 295-301 (2017).
- Le Cann, S., et al. Characterization of the bone-metal implant interface by Digital Volume Correlation of in-situ loading using neutron tomography. Journal of the Mechanical Behavior of Biomedical Materials. 75, 271-278 (2017).
- Burca, G., et al. Exploring the potential of neutron imaging for life sciences on IMAT. Journal of Microscopy. 272 (3), 242-247 (2018).
- Metzke, R. W., et al. Neutron computed tomography of rat lungs. Physics in Medicine and Biology. 56 (1), 1-10 (2011).
- Altieri, S., et al. Neutron autoradiography imaging of selective boron uptake in human metastatic tumours. Applied Radiation and Isotopes. 66 (12), 1850-1855 (2008).
- Altieri, S., Bortolussi, S., Bruschi, P., Pedroni, P., Zonta, A. Neutron radiography of human liver metastases after BPA infusion. Proceedings of 11th World congress on Neutron Capture Therapy. , (2004).
- Holz, M., Zarebanadkouki, M., Kaestner, A., Kuzyakov, Y., Carminati, A. Rhizodeposition under drought is controlled by root growth rate and rhizosphere water content. Plant and Soil. 423 (1-2), 429-442 (2018).
- Esser, H. G., Carminati, A., Vontobel, P., Lehmann, E. H., Oswald, S. E. Neutron radiography and tomography of water distribution in the root zone. Journal of Plant Nutrition and Soil Science. 173 (5), 757-764 (2010).
- Warren, J. M., et al. Neutron imaging reveals internal plant water dynamics. Plant and Soil. 366 (1-2), (2013).
- Dhiman, I., et al. Quantifying root water extraction after drought recovery using sub-mm in situ empirical data. Plant and Soil. 424, 73-89 (2018).
- Kroener, E., Zarebanadkouki, M., Kaestner, A., Carminati, A. Non-equilibrium dynamics of rhizosphere. Water Resources Research. 50 (8), 6479-6495 (2014).
- Moradi, A. B., et al. Three-dimensional visualization and quantification of water content in the rhizosphere. New Phytologist. 192 (3), 653-663 (2011).
- Banhart, J., et al. X-ray and neutron imaging – Complementary techniques for materials science and engineering. International Journal of Materials Research. 101 (9), 1069-1079 (2010).
- LaManna, J. M., Hussey, D. S., Baltic, E. M., Jacobson, D. L. Improving material identification by combining x-ray and neutron tomography. Proceedings 10391, Developments in X-Ray Tomography XI. 1039104, (2017).
- Tengattini, A., et al. NeXT-Gremoble, the Neutron and X-ray tomograph in Grenoble. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 968, 163939 (2020).
- Bilheux, H. Z., McGreevy, R. L., Anderson, I. S. . Neutron Imaging and Applications. , (2009).
- Zhang, P., Wittmann, F. H., Zhao, T. J., Lehmann, E. H., Vontobel, P. Neutron radiography, a powerful method to determine time-dependent moisture distributions in concrete. Nuclear Engineering and Design. 241 (12), 4758-4766 (2011).
- Lobo, R. M., Andrade, A. H. P., Castagnet, M. Hydride embrittlement in zircaloy components. Inac 2011 Int Nucl.Atlantic Conference. , 1-6 (2011).
- Kardjilov, N., et al. New trends in neutron imaging. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 605 (1-2), 13-15 (2009).
- Schillinger, B., et al. Detection systems for short-time stroboscopic neutron imaging and measurements on a rotating engine. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 542 (1-3), 142-147 (2005).
- Thewlis, J. Neutron Radiography. British Journal of Applied Physics. 7, 345-350 (1956).
- Anderson, J. Neutron Radiography in Man. British Journal of Radiology. 37, 957-958 (1964).
- Brown, M., Parks, P. B. Neutron radiography in biologic media: techniques, observations, and implications. American Journal of Roentgenology. 106 (3), 472-485 (1969).
- Metzke, R. W., Runck, H., Stahl, C. A., Schillinger, B., Calzada, E. Neutron computed tomography of rat lungs. Physics in Medicine and Biology. 56 (1), 1-10 (2011).
- Crow, L., et al. The CG1 instrument development test station at the high flux isotope reactor. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 634 (1), 71-74 (2011).
- Santodonato, L., et al. The CG-1D neutron imaging beamline at the Oak Ridge National Laboratory High Flux Isotope Reactor. Physics Procedia. 69, 104-108 (2015).
- Tremsin, A. S., et al. Improved efficiency of high resolution thermal and cold neutron imaging. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 628 (1), 415-418 (2011).
- Tremsin, A. S., Vallerga, J. V., McPhate, J. B., Siegmund, O. H. W. Optimization of high count rate event counting detector with microchannel plates and quad Timepix readout. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 787, 20-25 (2015).
- Vlassenbroeck, J., et al. Software tools for quantification of X-ray microtomography at the UGCT. Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 580 (1), 442-445 (2007).
- Gürsoy, D., De Carlo, F., Xiao, X., Jacobsen, C. TomoPy: A framework for the analysis of synchrotron tomographic data. Journal of Synchrotron Radiation. 21 (5), 1188-1193 (2014).
- Pelt, D. M., et al. Integration of TomoPy and the ASTRA toolbox for advanced processing and reconstruction of tomographic synchrotron data. Journal of Synchrotron Radiation. 23 (3), 842-849 (2016).
- Venkatakrishnan, S. V., Cakmak, E., Billheux, H., Bingham, P., Archibald, R. K. Model-based iterative reconstruction for neutron laminography. Conference Record of 51st Asilomar Conference on Signals, Systems and Computers, ACSSC 2017. , (2018).
- Stalling, D., Westerhoff, M., Hege, H. C. Amira: A highly interactive system for visual data analysis. Visualization Handbook. 1, 749-767 (2005).
- Grünzweig, C., Frei, G., Lehmann, E., Kühne, G., David, C. Highly absorbing gadolinium test device to characterize the performance of neutron imaging detector systems. Review of Scientific Instruments. 78 (5), 1-4 (2007).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Meagher, M. J., Parwani, R. N., Virdi, A. S., Sumner, D. R. Optimizing a micro-computed tomography-based surrogate measurement of bone-implant contact. Journal of Orthopaedic Research. 36 (3), 979-986 (2018).
- Dalesio, L. R., et al. The experimental physics and industrial control system architecture: past, present, and future. Nuclear Inst. and Methods in Physics Research, A. 352 (1-2), 179-184 (1994).
- Bilheux, J., Lin, J. Y. Y., Bilheux, H. Z. Jupyter notebooks for neutron radiography data processing and analysis. Neutron Radiography-WCNR-11. 15, 198-204 (2020).
- Trtik, P., et al. Improving the spatial resolution of neutron imaging at Paul Scherrer Institut – The Neutron Microscope Project. Physics Procedia. 69, 169-176 (2015).
- Morgano, M., et al. Unlocking high spatial resolution in neutron imaging through an add-on fibre optics taper. Optics Express. 26 (2), 9-12 (2018).

