Undersøgelse af proteindynamik via neutronspinekkospektroskopi
Summary
Denne protokol beskriver metoder til undersøgelse af strukturen og dynamikken i to modelproteiner, der har en vigtig rolle i menneskers sundhed. Teknikken kombinerer bench-top biofysisk karakterisering med neutron spin ekko spektroskopi for at få adgang til dynamikken på tids- og længdeskalaer, der er relevante for protein interdomain bevægelser.
Abstract
De fleste menneskelige kropsproteiners aktivitet og funktionalitet er relateret til konfigurationsændringer af hele underdomæner inden for proteinkrystalstrukturen. Krystalstrukturerne bygger grundlaget for enhver beregning, der beskriver strukturen eller dynamikken i et protein, det meste af tiden med stærke geometriske begrænsninger. Disse begrænsninger fra krystalstrukturen er imidlertid ikke til stede i opløsningen. Strukturen af proteinerne i opløsningen kan afvige fra krystallen på grund af omlejringer af sløjfer eller underdomæner på pico til nanosekund tidsskala (dvs. det interne proteindynamiktidsregime). Det nuværende arbejde beskriver, hvordan langsomme bevægelser på tidsskalaer på flere titalls nanosekunder kan tilgås ved hjælp af neutronspredning. Især den dynamiske karakterisering af to store humane proteiner, et iboende uordnet protein, der mangler en veldefineret sekundær struktur og et klassisk antistofprotein, behandles ved neutron spin ekkospektroskopi (NSE) kombineret med en bred vifte af laboratoriekarakteriseringsmetoder. Yderligere indsigt i proteindomænedynamik blev opnået ved hjælp af matematisk modellering til at beskrive de eksperimentelle neutrondata og bestemme crossover mellem kombinerede diffusive og interne proteinbevægelser. Ekstraktionen af det interne dynamiske bidrag til den mellemliggende spredningsfunktion opnået fra NSE, herunder tidsskalaen for de forskellige bevægelser, tillader yderligere syn på de mekaniske egenskaber af enkeltproteiner og proteinernes blødhed i deres næsten naturlige miljø i den overfyldte proteinopløsning.
Introduction
Sonderende dynamik af blødt stof med neutroner
Undersøgelse af proteiners og peptiders dynamiske egenskaber er en stor del af biofysisk forskning, og der findes i dag mange veludviklede metoder til at få adgang til en bred vifte af energilandskaber1. At relatere proteinernes eksperimentelt afslørede dynamik til deres biologiske funktion er en langt vanskeligere opgave, der kræver komplekse matematiske modeller og computerstøttede dynamiksimuleringer. Betydningen af neutronspektroskopi for analyse af proteinbevægelser er blevet understreget i flere velmodtagne og bredt anerkendte undersøgelser 1,2,3,4,5. Før du udforsker det mangfoldige energilandskab af intern proteindynamik, kræves der et kort overblik over de dynamiske processer i blødt stof, og hvordan neutroner kan få adgang til dem.
Neutronernes følsomhed over for isotopkonfiguration og den type interaktioner, de viser med blødt stof, gør neutronspredning til en af de mest alsidige undersøgelsesteknikker6. Der er et bredt spektrum af korrelationslængdeskalaer og korrelationstider, som neutroner kan få adgang til, fra nukleare excitationer og atomvibrationer til kollektive bevægelser og langsomme afslapningsprocesser som isotropiske rotationer og diffusive bevægelser. Når man undersøger de spredte neutroner for deres energioverførsel, kan der skelnes mellem tre hovedinteraktioner: den elastiske spredning, hvor der ikke er nogen energiudveksling mellem indkommende neutron og partikel i prøven; den uelastiske spredning med en stor, kvantificerbar energiudveksling mellem neutron og partikel; og det ejendommelige tilfælde af kvasi-elastisk spredning, der betegner en meget lille energioverførsel sammenlignet med den indfaldende neutronenergi 1,7. Disse interaktioner giver præcise oplysninger om det undersøgte materiale og danner det teoretiske grundlag for en lang række neutronspredningsteknikker.
I elastisk spredning registrerer detektoren neutronernes retninger som et diffraktionsmønster, der viser prøveatomernes position i forhold til hinanden. Oplysninger om korrelationerne mellem atompositioner erhverves (dvs. integreret intensitet S (Q) vedrørende momentumoverførslen Q, som kun vedrører strukturel information). Dette princip danner grundlaget for neutrondiffraktion8.
Kompleksitet opstår, når energioverførslen ikke længere er nul på grund af excitationer og interne udsving i prøvematerialet. Dette danner grundlag for neutronspektroskopi, hvor de spredte neutroner undersøges som en funktion af både energioverførslen E og momentumoverførslen Q. Dynamisk og strukturel information opnås. Neutronspektroskopi måler den samme integrerede intensitet S(Q) for energioverførsel (dvs. hastighedsændring af neutronerne på grund af prøvespredning, S(Q,ω) = S(Q, E), som også kaldes den dynamiske strukturfaktor)9.
Til beregning af spredningen fra et materiale er det mere hensigtsmæssigt at anvende parkorrelationsfunktionen 7,10. I diffraktionssagen giver den statiske parkorrelationsfunktion G (r) sandsynligheden for at finde midten af en partikel i en given afstand r fra centrum af en anden partikel. Spektroskopien generaliserer den statiske parkorrelationsfunktion og inkluderer energi / frekvens / tid i spredningsligningen. Parkorrelationsfunktionen G(r) bliver en funktion af tiden G(r, t), som kan nedbrydes til en særskilt atomparkorrelationsfunktion GD(r, t) og en selvkorrelationsfunktion GS(r, t). Disse beskriver to typer korrelationer: parkorrelerede bevægelser af atomer, der styrer den sammenhængende spredning, og selvkorrelation, der styrer den usammenhængende spredning10.
Sammenhængende spredning er spredningen fra “gennemsnittet” og afhænger af den relative fase af de spredte bølger. I småvinkelspredningsregimet interfererer de spredte neutronbølger fra forskellige spredningscentre (forskellige atomer) konstruktivt (har lignende faser), og atomernes kollektive bevægelse observeres med stærk intensitetsforbedring. Sammenhængende spredning beskriver i det væsentlige spredningen af en enkelt neutron fra alle kernerne i prøve10.
Når der ikke opstår nogen konstruktiv interferens mellem de spredte neutronbølger fra forskellige centre, følges et enkelt atom i tide, og selvkorrelationen mellem atomets position på tidspunktet t = 0 og det samme atom på tidspunktet t observeres. Således går oplysningerne om atomernes relative positioner tabt, og fokus er kun på lokale udsving. Spredning fra lokale udsving styrer usammenhængende spredning. Usammenhængende spredning er isotrop, bidrager til baggrundssignalet og nedbryder signal-til-støj10,11.
Ved at kombinere alt det ovenstående skelner vi mellem fire store neutronspredningsprocesser10: (1) elastisk sammenhængende (måler korrelationerne mellem atompositioner), (2) uelastisk sammenhængende (måler kollektive bevægelser af atomer), (3) elastisk usammenhængende (bidrager til baggrunden, reducerer spredningsintensiteten ved Debye-Waller-faktor (DWF) og måler elastisk usammenhængende strukturfaktor (EISF), der beskriver geometrien af diffusive bevægelser i begrænset geometri, og (4) uelastisk usammenhængende (måler enkeltatomdynamik og selvkorrelation).
Dynamikprocesser, som neutroner kan få adgang til i biologi, spænder fra dæmpning af lavfrekvente atomare og molekylære vibrationer, interaktionen mellem opløsningsmiddelmolekyler og biooverflader og diffusionsprocesser i hydreringslaget af makromolekyler og begrænset geometri til kortdistance translationelle, roterende og tumlende diffusive bevægelser og proteindomæner og allosteriske bevægelser1 . Den brede mangfoldighed af neutronmetoder og instrumenter til måling af proteindynamik er baseret på, hvordan akromatiseringen af den indfaldende eller udgående neutronstråle opnås, og hvordan energianalysen af de spredte neutroner udføres. Fra triple-axis til time-of-flight, backscattering og spin-echo spektrometre kan man udforske dynamiske processer med karakteristiske tider mellem 1 x 10-14 s og 1 x 10-6 s (femtosekund til mikrosekunder)12.
Oak Ridge National Laboratory, med sine to berømte neutronkilder, Spallation Neutron Source – SNS13 og High Isotope Flux Reactor – HFIR14, har en af de bedste suiter af spektrometre til undersøgelse af dynamik i biomaterialer. Nogle af de mest veltalende eksempler inkluderer brugen af det kolde neutronhakkerspektrometer (CNCS) ved SNS15 til at undersøge den dynamiske forstyrrelse af hydreringsvand omkring grønt fluorescerende protein i opløsning16 eller de sub-picosekund kollektive vibrationer af flere proteiner17. Et tilbagevendende problem med uelastiske neutronspredningsundersøgelser er, at nogle biologiske processer er for langsomme til at blive observeret. Uden ekstreme opsætninger, der fører til et enormt tab af neutronintensitet, er time-of-flight-spektrometre begrænset til 10 μeV energiopløsning, svarende til en maksimal tidsskala på ~ 200 ps10,11. Dette er ikke tilstrækkeligt til at observere store bevægelser i proteiner. Derfor er der ofte brug for instrumenter med højere energiopløsning som backscattering-spektrometrene. Kombinationen af flyvetids- og backscatteringsteknikker har vist sig at være effektiv til at undersøge ændringen i den interne dynamik i Cytochrome P450cam (CYP101), et enzym, der katalyserer hydroxyleringskamferen18.
Mikroskopisk diffusivitet målt ved backscattering-spektrometeret ved SNS-BASIS19 var overraskende veldefineret og kunne adskilles i diffusiviteten af vand (hydrering, cytoplasmisk og bulklignende vand) og diffusiviteten af cellebestanddele i planariske fladorme, det første levende dyr, der blev undersøgt ved neutronspredning20 . Backscattering er en højopløsningsspektroskopisk teknik, men den er også begrænset til flere μeV = flere nanosekunder, mens den langsomme dynamik i biomaterialer også manifesterer sig som overlevelsestiden for korrelation mellem atomposition eller spinorientering (f.eks. Afslapningsprocesser, som regelmæssigt sker i tidsintervallet ti til hundreder af nanosekunder).
Neutron spin ekko spektroskopi (NSE) er den eneste neutronspredningsteknik, der når så høj opløsning. I modsætning til andre neutronteknikker kræver NSE ikke akromatisering af strålen, da den bruger neutronernes kvantemekaniske fase, som er deres magnetiske øjeblikke. Manipulationen af magnetiske øjeblikke tillader anvendelse af en bred neutronstrålebølgelængdefordeling, mens teknikken er følsom over for meget små neutronhastighedsændringer i størrelsesordenen 1 x 10-4. NSE er med succes blevet brugt til at undersøge den langsomme dynamik af proteiner i opløsning for mange proteiner. Blandt disse mange pionerstudier anerkender vi undersøgelsen af segmentfleksibiliteten af svineimmunoglobulin21; de koblede domænebevægelser i Taq-polymerase22; domænebevægelserne i tetrameren af gæralkoholdehydrogenase23; ændringen af konformation i phosphoglyceratkinase ved substratbinding3; aktivering af domænebevægelser og dynamisk udbredelse af allosteriske signaler i Na+/H+-udvekslingsregulerende cofaktor 1 (NHERF1)-proteinet 4,24,25 dynamikken i en kompakt tilstand af mercurisk ionreduktase26; og diffusion af hæmoglobin i røde blodlegemer27. To nyere undersøgelser i proteindynamik har afsløret fleksibiliteten af humant antistof Immunoglobulin G (IgG) som en entropisk forår28 og egenskaberne ved opløsningsmiddelbidrag til dynamikken i iboende forstyrret myelinbasisk protein (MBP)5.
Denne artikel forklarer de grundlæggende principper for NSE, de mange forberedende metoder, der anbefales til en grundig proteindynamikundersøgelse, samt metoden og den eksperimentelle protokol til NSE-dataindsamling ved NSE-spektrometeret ved SNS, SNS-NSE. Protokollen karakteriserer to proteiner: IgG, et almindeligt humant antistofprotein, og det iboende uordnede protein MBP. De biofysiske implikationer, eksemplernes forskningsrelevans og teknikkens begrænsninger diskuteres kort.
NSE-spektroskopi, metoden til målinger af langsom dynamik
NSE er en polariseret teknik, der bruger neutron-time-of-flight til at måle udvekslingen af energi (tab af polarisering) på grund af den kvasi-elastiske interaktion mellem neutroner og atomer i en prøve. Kernen i NSE-spektroskopi ligger to grundlæggende principper: (1) neutronspinets evne til at præcessere i magnetfeltet med en frekvens, der er proportional med magnetisk styrke 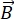 , nemlig Larmor-frekvensen29, og (b) spin-ekkoet eller Hann-ekkoet, der repræsenterer manipulation og refokusering af polarisationssignalet, når der anvendes en række radiofrekvensimpulser30.
, nemlig Larmor-frekvensen29, og (b) spin-ekkoet eller Hann-ekkoet, der repræsenterer manipulation og refokusering af polarisationssignalet, når der anvendes en række radiofrekvensimpulser30.
Det grundlæggende i NSE-processen kan opsummeres i et par enkle trin 6,11 ved hjælp af figur 1. (1) Neutronstrålen produceret af kilden (position 1) polariseres (position 2), styres og transporteres (position 3) og ankommer til indgangen til NSE-spektrometeret, hvor den roteres med 90 ° af den første pi-half flipper (position 4). (2) Den polariserede stråle (f.eks. neutronmagnetiske øjeblikke) bliver vinkelret på den første magnets magnetfeltlinjer (første præcessionszone, position 5) og begynder at gå forud. (3) I slutningen af magneten akkumulerer neutronspins en bestemt præcessionsvinkel, der er proportional med magnetfeltstyrken og flyvetiden brugt indeni (stort set omvendt proportional med neutronhastigheden). De enkelte neutronhastigheder er kodet inden for deres præcessionsvinkel i slutningen af den første præcessionszone. (4) Tæt på prøvepositionen vender pi-flipperen (position 6) orienteringen af spin med 180° og ændrer tegnet på præcessionsvinklen. (5) Neutronerne interagerer med prøvens molekyler (position 7) og bliver spredt. (6) De spredte neutroner kommer ind og går ind i den anden præcessionszone (position 8), men bliver omvendt orienterede. (7) En anden pi-half flipper (position 9) bruges til at rotere spinnets retning fra vinkelret på vandret retning. Dette vil stoppe præcessionen og oversætte præcessionsvinklen φ til polarisering proportional med cos (φ). (8) Analysatoren (position 10) vælger neutronerne baseret på en retning. Hvis interaktionen med prøven er elastisk, ændres neutronens hastighed ikke. Neutronerne vil bruge en identisk mængde tid på at flyve i den første og anden præcessionszone, og de akkumulerede præcessionsvinkler genvindes fuldt ud. Den fulde polarisering genoprettes på detektoren (position 11) som et ekko af den oprindelige polarisering (dvs. spin-ekko). (9) I NSE er spredningen imidlertid kvasi-elastisk, så en lille energiudveksling mellem neutroner og prøvemolekyler fører til forskellige neutronhastigheder efter spredning af prøven. På grund af de forskellige hastigheder vil neutronerne bruge en ekstra tid på at flyve gennem den anden præcessionszone og vil ikke have genoprettet deres præcessionsvinkel korrekt. En delvis polarisering hentes på detektoren, og tabet af polarisation på grund af spinafslapning er proportional med cos-Fourier-transformationen af spektralfunktionen S(Q, ω), den mellemliggende spredningsfunktion F(Q, t). (10) Tidsparameteren for funktionen F(Q, t) er proportional med præcessionens magnetfeltstyrke. Scanning af tabet af polarisation som en funktion af magnetfeltstyrken giver derfor en afslapningsfunktion, der afhænger af de dynamiske processer i prøven.

Figur 1: Fotografi af NSE-spektrometeret ved SNS (SNS-NSE) og neutronfluebane skematisk med de vigtigste funktionelle komponenter. Fra højre mod venstre: 1 = neutronkilde; 2 = choppers-bender-polarizer-sekundær lukkersystem; 3 = bjælketransport guider; 4 = pi/2 flipper til første 90° spin-turn; 5 = første præcessionszone; 6 = pi flipper til 180 ° spin-turn; 7 = prøveareal og prøvemiljø (her vises kryoovnen); 8 = anden præcessionszone; 9 = pi/2 flipper til anden 90° spin-turn; 10 = analysator; 11 = detektor. (Bemærk, at dele af 3, såvel som 2 og 1, er placeret bag den blå væg inde i afskærmning; chopperne erstattes af en hastighedsvælger til reaktorbaseret NSE). Klik her for at se en større version af denne figur.
Protocol
Representative Results
Discussion
NSE-spektroskopi giver et unikt og detaljeret billede af proteinernes dynamik, som andre spektroskopiske teknikker ikke kan producere. Målinger over en længere tidsskala giver observationer af både proteinernes translationelle og rotationsdiffusion, som præsenteret her. Segmentdynamikken og andre interne svingninger afslører sig som et stærkt henfald af den sammenhængende spredningsfunktion S(Q, t) på en kort tidsskala og er godt adskilt fra de overordnede diffusionsafspændingsprocesser. De vigtigste be…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Denne forskning brugte ressourcer på Spallation Neutron Source (BL-15, BL-6, Biologi og Kemi laboratorier), et DOE Office of the Science User Facility, der drives af Oak Ridge National Laboratory. Denne forskning brugte også ressourcer på MLZ-FRM2-reaktoren Garching (KWS-2, Phoenix-J-NSE) og JCNS1 på Forschungszentrum Jülich GmbH, Tyskland. Forfatteren anerkender Dr. Ralf Biehl og Dr. Andreas Stadler for deres hjælp med modellering og deres bidrag til både IgG- og MBP-proteinforskning, Dr. Piotr A. Żołnierczuk for NSE-datareduktionsstøtte, Dr. Changwoo Do for støtte med SANS-målinger og Rhonda Moody og Dr. Kevin Weiss for SNS biokemilaboratoriestøtte.
Materials
| Bovine MBP protein solution | Sigma-Aldrich | M1891 | lyophilized powder reconstituted in D2O |
| D2O – heavy water | Sigma-Aldrich | Product No. 151882 | liquid |
| Dionized water | in house | – | for washing / cleanning cells |
| DLS instrument | Zetasizer Nano ZS, FZ-Jülich | – | dynamic light scattering instrument |
| Elastic scattering standards | SNS-NSE, ORNL | – | Al2O3 and Graphite powders |
| Ethanol | Sigma-Aldrich | 65350-M | 70% ethanol for cleaning cells |
| IgG protein solution | Sigma-Aldrich | I4506 | lyophilized powder reconstituted in D2O |
| KWS-2 instrument | JCNS outstation at the MLZ, Garching, Germany | – | small angle neutron instrument |
| Liquinox dish detergent | Alconox | – | Phosphate-free liquid lab glassware cleaner |
| Na2HPO4·7H2O | Sigma-Aldrich | Product No.S9390 | disodium phosphate heptahydrate salt |
| NaCl | Sigma-Aldrich | Product No.S9888 | sodium chloride salt |
| NaH2PO4·H2O | Sigma-Aldrich | Product No. S9638 | monosodium phosphate monohydrate salt |
| Nanodrop spectrophotometer | Thermo Scientific | Catalog number: ND-2000 | NanoDrop 2000/2000c Spectrophotometer |
| Neutron alignment camera | NeutronOptics, Grenoble | NOG210222 | 100 x 100 mm camera with Sony IMX249 CMOS sensor |
| Parafilm M – wax parafilm | Bemis | Parafilm M – 5259-04LC PM996 | all-purpose laboratory film in cardboard dispenser |
| Phoenix-J-NSE Spectrometer | JCNS outstation at the MLZ, Garching, Germany | – | neutron spectrometer |
| SasView | https://www.sasview.org/ | ||
| SAXSpace, Anton Paar instrument | FZ-Jülich | – | small angle x-ray instrument |
| Slide-A-Lyzer dialysis membranes | Thermo Scientific | 88400-88405 | Slide-A-Lyzer mini dialysis devices tubes of 3.5 K MWCO |
| SNS Remote Analysis Cluster | Neutron Science Remote Analysis (sns.gov) | https://analysis.sns.gov | |
| SNS-NSE spectrometer | ORNL, Oak Ridge, TN, USA | – | neutron spectrometer |
| Sterile syringe filters | VWR | N.A. PN:28145-501 | 0.2 µm pore size filters |
| Temperature Forcing System (TFS) | SP Scientific | Part Number 100004055 | sample environment equipment |
| Urea -d4 | Sigma-Aldrich | Product No. 176087 | deuterated Urea salt |
| Viscometer | FZ-Jülich | – | falling ball viscometer |
References
- Fitter, J., Gutberlet, T., Katsaras, J. . Neutron Scattering in Biology: Techniques and Applications. , (2006).
- Stadler, A., Monkenbusch, M., Biehl, R., Richter, D., Ollivier, J. Neutron spin-echo and TOF reveals protein dynamics in solution. Journal of the Physical Society of Japan. 82, (2013).
- Inoue, R. Large domain fluctuations on 50-ns timescale enable catalytic activity in phosphoglycerate kinase. Biophysical Journal. 99 (7), 2309-2317 (2010).
- Callaway, D. J. E., et al. Controllable activation of nanoscale dynamics in a disordered protein alters binding kinetics. Journal of Molecular Biology. 429 (7), 987-998 (2017).
- Stingaciu, L. R., Biehl, R., Changwoo, D., Richter, D., Stadler, A. M. Reduced internal friction by osmolyte interaction in intrinsically disordered myelin basic protein. Journal of Physical Chemistry Letters. 11 (1), 292-296 (2020).
- Monkenbusch, M., Richter, D. High resolution neutron spectroscopy-a tool for the investigation of dynamics of polymers and soft matter. Comptes Rendus Physique. , (2007).
- Richter, D., Monkenbusch, M., Schwahn, D. Neutron Scattering. Polymer Science: A Comprehensive Reference, 10 Volume Set. , (2012).
- Wilson, C. C. . Single Crystal Neutron Diffraction From Molecular Materials. , (2000).
- Marshall, W. . Theory of thermal neutron scattering. , (1971).
- . Roger Pynn Introduction & Neutron Scattering "Theory" Available from: https://neutrons.ornl.gov/sites/default/files/intro_to_neutron_scattering.pdf (2004)
- Richter, D. Neutron scattering in polymer physics. Physica B: Condensed Matter. 276-278, 22-29 (2000).
- Harroun, T. A., Wignall, G. D., Katsaras, J. Neutron scattering for biology. Neutron Scattering in Biology. , (2006).
- . SNS Available from: https://neutrons.ornl.gov/sna (2020)
- . HFIR Available from: https://neutrons.ornl.gov/hfir (2020)
- . CNCS Available from: https://neutrons.ornl.gov/cncs (2020)
- Perticaroli, S., et al. Description of hydration water in protein (green fluorescent protein) solution. Journal of the American Chemical Society. 139 (3), 1098-1105 (2017).
- Perticaroli, S., Nickels, J. D., Ehlers, G., Sokolov, A. P. Rigidity, secondary structure, and the universality of the boson peak in proteins. Biophysical Journal. 106 (12), 2667-2674 (2014).
- Miao, Y., et al. Coupled flexibility change in cytochrome p450cam substrate binding determined by neutron scattering, NMR, and molecular dynamics simulation. Biophysical Journal. 103 (10), 2167-2176 (2012).
- Mamontov, E., Zamponi, M., Hammons, S., Keener, W. S., Hagen, M., Herwig, K. W. BASIS: A new backscattering spectrometer at the SNS. Neutron News. 19 (3), 22-24 (2008).
- Mamontov, E. Microscopic diffusion processes measured in living planarians. Scientific Reports. 9, 8708 (2018).
- Alpert, Y., Cser, L., Faragó, B., Franěk, F., Mezei, F., Ostanevich, Y. M. Segmental flexibility in pig immunoglobulin G studied by neutron spin-echo technique. Biopolymers. 24 (9), 1769-1784 (1985).
- Bu, Z., Biehl, R., Monkenbusch, M., Richter, D., Callaway, D. J. E. Coupled protein domain motion in Taq polymerase revealed by neutron spin-echo spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 102 (49), 17646-17651 (2005).
- Biehl, R., et al. Direct observation of correlated interdomain motion in alcohol dehydrogenase. Physical Review Letters. 101, 138102 (2008).
- Farago, B., Li, J., Cornilescu, G., Callaway, D. J. E., Bu, Z. Activation of nanoscale allosteric protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 99 (10), 3473-3482 (2010).
- Bu, Z., Callaway, D. J. E. Dynamic propagation of long-range allosteric signals by nanoscale protein domain motion revealed by neutron spin echo spectroscopy. Biophysical Journal. 100 (3), 223 (2011).
- Hong, L., et al. Structure and dynamics of a compact state of a multidomain protein, the mercuric ion reductase. Biophysical Journal. 107 (2), 393-400 (2014).
- Longeville, S., Stingaciu, L. -. R. Hemoglobin diffusion and the dynamics of oxygen capture by red blood cells. Scientific Reports. 7, 10448 (2017).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Hahn, E. L. Nuclear induction due to free larmor precession. Physical Review. 77, 297 (1950).
- Hahn, E. L. Spin echoes. Physical Review. 80, 580 (1950).
- Svergun, D. I., Koch, M. H. J. Small-angle scattering studies of biological macromolecules in solution. Reports on Progress in Physics. 66 (10), 1735-1782 (2003).
- . Sasview Available from: https://www.sasview.org (2020)
- Zhao, J. K., Gao, C. Y., Liu, D. The extended Q-range small-angle neutron scattering diffractometer at the SNS. Journal of Applied Crystallography. 43, 1068-1077 (2010).
- Ohl, M., et al. The high-resolution neutron spin-echo spectrometer for the SNS with τ ≥ 1 µs. Physica B: Condensed Matter. 350, 147-150 (2004).
- . SNS-NSE web page Available from: https://neutrons.ornl.gov/nse (2022)
- Ohl, M., et al. The spin-echo spectrometer at the Spallation Neutron Source (SNS). Nuclear Instruments and Methods in Physics Research, Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 696, 85-99 (2012).
- Zolnierczuk, P. A., Holderer, O., Pasini, S., Kozielewski, T., Stingaciu, L. R., Monkenbusch, M. Efficient data extraction from neutron time-of-flight spin-echo raw data. Journal of Applied Crystallography. 52 (5), 1022-1034 (2019).
- . Dr Spine hub Available from: https://jugit.fz-juelich.de/nse/drspine (2022)
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), (2014).
- . FZJ Available from: https://www.fz-juelich.de/portal/EN/AboutUs (2022)
- . KWS2 Available from: https://miz-garching.de/kws-2 (2022)
- Moorhouse, M., Barry, P. The protein databank. Bioinformatics Biocomputing and Perl. , (2005).
- Tria, G., Mertens, H. D. T., Kachala, M., Svergun, D. I. Advanced ensemble modelling of flexible macromolecules using X-ray solution scattering. IUCrJ. 2 (2), 207-217 (2015).
- Holderer, O., Monkenbusch, M., Schätzler, R., Kleines, H., Westerhausen, W., Richter, D. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 90 (4), 043107 (2008).
- Biehl, R., Monkenbusch, M., Richter, D. Exploring internal protein dynamics by neutron spin echo spectroscopy. Soft Matter. 7 (4), 1299-1307 (2011).
- Stingaciu, L. R., Ivanova, O., Ohl, M., Biehl, R., Richter, D. Fast antibody fragment motion: Flexible linkers act as entropic spring. Scientific Reports. 6, 22148 (2016).
- Stadler, A. M., et al. Internal nanosecond dynamics in the intrinsically disordered myelin basic protein. Journal of the American Chemical Society. 136 (19), 6987-6994 (2014).
- Biehl, R., Richter, D. Slow internal protein dynamics in solution. Journal of Physics: Condensed Matter. 26 (50), 503103 (2014).
- Hinsen, K. The molecular modeling toolkit: A new approach to molecular simulations. Journal of Computational Chemistry. 21 (2), 79-85 (2000).
- Uhlenbeck, G. E., Ornstein, L. S. On the theory of the Brownian motion. Physical Review. 36 (5), 823 (1930).
- Wang, M. C., Uhlenbeck, G. E. On the theory of the Brownian motion II. Reviews of Modern Physics. 17, 323 (1945).
- Callaway, D. J. E., Bu, Z. Nanoscale protein domain motion and long-range allostery in signaling proteins-a view from neutron spin echo spectroscopy. Biophysical Reviews. 7 (2), 165-174 (2015).
- Liu, Y. Intermediate scattering function for macromolecules in solution probed by neutron spin echo. Physical Review E. 95, 020501 (2017).
- Liu, Y. Short-time dynamics of proteins in solutions studied by neutron spin echo. Current Opinion in Colloid & Interface Science. 42, 147-156 (2019).

