En 3D-kartografisk beskrivelse af cellen ved Hjælp af Cryo Soft X-ray Tomography
Summary
Her præsenteres en protokol, der beskriver de prøveforberedelses- og dataindsamlingstrin, der kræves i kryo blød røntgentomografi (SXT) for at afbilde ultrastrukturen af hele kryokonserverede celler i en opløsning på 25 nm halv tonehøjde.
Abstract
Billeddannelsesteknikker er grundlæggende for at forstå celleorganisation og maskiner i biologisk forskning og de beslægtede områder. Blandt disse teknikker tillader cryo soft X-ray tomography (SXT) billeddannelse af hele kryo-bevarede celler i vandvinduet røntgenenergiområde (284-543 eV), hvor kulstofstrukturer har iboende højere absorption end vand, hvilket muliggør 3D-rekonstruktion af den lineære absorptionskoefficient for materialet indeholdt i hver voxel. Kvantitativ strukturel information på niveau med hele celler op til 10 μm tyk kan derefter opnås på denne måde med høj gennemstrømning og rumlig opløsning ned til 25-30 nm halv tonehøjde. Cryo-SXT har vist sig relevant for den nuværende biomedicinske forskning, idet den har givet 3D-information om cellulære infektionsprocesser (virus, bakterier eller parasitter), morfologiske ændringer på grund af sygdomme (såsom recessive genetiske sygdomme) og hjulpet os med at forstå lægemiddelhandling på celleniveau eller lokalisere specifikke strukturer i det 3D-cellulære miljø. Ved at drage fordel af den indstillelige bølgelængde ved synkrotronfaciliteter kan spektromikroskopi eller dens 3D-modstykke, spektrotomografi, også bruges til at afbilde og kvantificere specifikke elementer i cellen, såsom calcium i biomineraliseringsprocesser. Cryo-SXT giver supplerende information til andre biologiske billeddannelsesteknikker såsom elektronmikroskopi, røntgenfluorescens eller synlig lysfluorescens og bruges generelt som en partnermetode til 2D- eller 3D-korrelativ billeddannelse ved kryogene forhold for at forbinde funktion, placering og morfologi.
Introduction
Cryo-SXT kan spille en central rolle i biologisk billeddannelsesforskning, da det giver 3D-medium opløsning (25-30 nm halv tonehøjde) volumener af hydrerede hele celler 1,2,3,4,5,6. I vandvinduets energiområde, mellem kulstof- og iltabsorptionen K-kanterne (4,4-2,3 nm), absorberer kulstofrige cellulære strukturer 10 gange mere end det iltrige medium, der gennemsyrer og omgiver dem. I dette energiområde kan glaserede celler op til 10 μm tykkelse afbildes uden behov for sektionering eller farvning, hvilket fører til kvantitative højabsorptionskontrastfremskrivninger, som kombineret med prøverotationsfunktioner muliggør tomografisk rekonstruktion af den cellulære struktur. Cryo-SXT udfylder en niche med hensyn til prøvedimensioner og rumlig opløsning, der ikke er let tilgængelig med nogen anden billeddannelsesteknik.
Kort sagt er absorptionskontrasten af cryo-SXT kvantitativ, da dæmpning af fotonerne gennem prøven af tykkelse t adlyder Beer-Lambert-loven som følger: 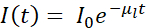 , hvor I0 repræsenterer hændelsesintensiteten og μl den lineære absorptionskoefficient, som afhænger af bølgelængden λ og den imaginære del β af prøvens brydningsindeks (
, hvor I0 repræsenterer hændelsesintensiteten og μl den lineære absorptionskoefficient, som afhænger af bølgelængden λ og den imaginære del β af prøvens brydningsindeks (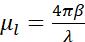 ). Dæmpningen er en funktion af den biokemiske sammensætning og tykkelsen af de strukturer, der afbildes, hvor hver biokemisk komponent har en specifik røntgen lineær absorptionskoefficient μl (LAC). Det betyder, at hver tomografi voxelværdi afhænger af de kemiske elementer og deres koncentration i den voxel7. Dette muliggør naturlig diskrimination af forskellige organeller såsom kerner, nukleoler, lipidlegemer eller mitokondrier eller forskellige komprimeringstilstande af kromatin udelukkende baseret på deres iboende LAC-værdier rekonstrueret 2,8,9.
). Dæmpningen er en funktion af den biokemiske sammensætning og tykkelsen af de strukturer, der afbildes, hvor hver biokemisk komponent har en specifik røntgen lineær absorptionskoefficient μl (LAC). Det betyder, at hver tomografi voxelværdi afhænger af de kemiske elementer og deres koncentration i den voxel7. Dette muliggør naturlig diskrimination af forskellige organeller såsom kerner, nukleoler, lipidlegemer eller mitokondrier eller forskellige komprimeringstilstande af kromatin udelukkende baseret på deres iboende LAC-værdier rekonstrueret 2,8,9.
Derudover er cryo-SXT en teknik med høj gennemstrømning, hvor tomogrammer indsamles på få minutter. Dette muliggør specifikt mesoskala billeddannelse af cellepopulationer, der kan fanges på vigtige tidspunkter såsom opdeling, differentiering og apoptose, men også ved forskellige responstilstande som dem, der induceres af kemisk eksponering for specifikke lægemiddelterapier eller patogene infektioner. Data indsamlet på disse nøglepunkter vil levere 3D-beskrivelse af systemet med en trofast registrering af den rumlige organisering af de forskellige cellulære organeller på disse specifikke øjeblikke.
Normalt anvendes cryo-SXT i kombination med andre teknikker efter korrelative tilgange, der gør det muligt at lokalisere specifikke funktioner, begivenheder eller makromolekyler inden for 3D-cellulære miljø 4,10,11,12,13,14,15,16 eller hårde røntgenfluorescensdata 17,18 . Korrelative tilgange ved kryogene forhold er af afgørende betydning for at opnå det mest komplette og værdifulde billede af det system, der er af interesse. Et kortfattet resumé af den typiske arbejdsgang på Mistral (Alba) og B24 (Diamond) cryo-SXT strålelinjer er skitseret i figur 1.
Ved at udnytte bølgelængdeindstillingsevnen ved synkrotronfaciliteter kan der desuden opnås spektroskopisk information ud over den strukturelle ved anvendelse af den specifikke differentielle absorption af bestemte elementer indeholdt i prøven. Et eksempel på dette ville være placeringen af calcium i undersøgelsen af biomineraliseringsprocesser i celler 19,20,21. Ved at tage 2D-billeder ved forskellige fotonenergier (spektre) eller tomogrammer nedenfor og ved den røntgenabsorptionskant, der er af interesse, kan pixels eller voxeller, der indeholder det valgte element, identificeres. Spektre tillader også differentiering af kemiske tilstande (dvs. udviklingen af amorft calcium til hydroxyapatit som i det tidligere biomineraliseringseksempel20). Kvantificering af forskellige elementer er mulig i 2D og 3D. Spektroskopisk billeddannelse af forglasede celler udføres typisk i vandvinduet, men er også mulig i andre energiområder, hvis vandindholdet er lavt nok, eller hvis der anvendes andre prøveforberedelsesprotokoller, herunder dehydrering,22. En detaljeret spektroskopi trin-for-trin protokol er uden for protokollens fokus heri.
I det følgende fokuserer protokollen på kort at opsummere de vigtigste prøveforberedelsestrin, selvom hvert system muligvis har brug for individuel forfining, efterfulgt af en detaljeret trin-for-trin dataindsamlingsprocedure for kryo blød røntgentomografi.
Protocol
Representative Results
Discussion
Prøveforberedelse er et kritisk skridt for at opnå bløde røntgentomogrammer af høj kvalitet, da deres kvalitet direkte afhænger af kvaliteten af prøvens forglasning og islagstykkelsen, hvori cellen er indlejret. Fremskrivninger med højt signal-støj-forhold vil blive indsamlet i områder med tyndt islag, hvilket gør det muligt at minimere den strålingsdosis, der kræves for at opnå den højest mulige opløsning. Derudover vil cellesammenløbet også påvirke den endelige tomogramkvalitet, da man bør undgå at have naboceller, der kommer ind i FoV ved rotation. Endelig vil den rigtige spredning af Au fiducial markører bestemme nøjagtigheden af projektionsjusteringen og derefter i sidste ende bestemme kvaliteten af det endelige 3D-rekonstruerede volumen. Bemærk, at en korrekt spredning af Au fiducials på nettet muliggør automatisering af projektionsjusteringstrinnet, uden hvilket der er behov for en høj ekspertise til et så kritisk trin.
Protokollen heri viser kun en mulig prøveforberedelsesstrategi, som har ligheder med dem, der anvendes i kryoelektrontomografi (cryo-ET). I begge tilfælde vil protokoller, der forbedrer den krævende prøveforberedelse for bedre reproducerbarhed, være grundlæggende for succesen med disse teknikker, og der gøres en indsats for at nå dette mål29. Det er værd at nævne, at ud over billeddannelse af isolerede celler kan dele af væv også visualiseres, forudsat at transmissionssignalet gennem sektionen vil være nok ved høje hældningsvinkler. Typisk vil dette indebære sektioner på få mikron (under 10 μm).
For at afbilde en bestemt struktur eller begivenhed inde i en celle skal man sørge for, at denne særlige funktion er inde i FoV i vippeserien. Da FoV i cryo-SXT er begrænset til 10 x 10 μm2 til 15 x 15 μm2 afhængigt af objektivet og tegner sig for en pixeloversampling af opløsningen til mindst en faktor 2, er den ofte mindre end fuldcelleudvidelsen (se de røde firkanter angivet i figur 5). Derfor skal ROI findes og mærkes korrekt. Dette gøres normalt ved hjælp af fluorescerende tags og synlige lyskorrelative tilgange. 2D-strategier, der kombinerer epifluorescens, er ligetil, da det bløde røntgentransmissionsmikroskop har et integreret online synligt lysfluorescensmikroskop, men andre tilgange til 2D- eller 3D-fluorescenssignal i høj opløsning er også tilgængelige 4,12,13,15,16 . I disse tilfælde skal gitteret først afbildes i specifikke instrumenter såsom superopløsningsmikroskoper. Bemærk, at de mest effektive korrelative tilgange er dem, der involverer dataindsamling under kryogene forhold. Dette skyldes, at tidsforløbet mellem stuetemperatur (RT), synlig lysfluorescensbilleddannelse og prøveglasning for eksempel vil forhindre at fange den rigtige cellulære begivenhed til tiden; Desuden kan forglasningsproceduren fjerne den celle af interesse, der er afbildet på RT, fra gitteret. Selv om de fleste korrelative billeddannelsesmetoder kan indebære, at prøvegitterene skal manipuleres og transporteres fra det ene instrument til det andet, og på trods af den øgede risiko for netforurening eller skade, dette udgør, er belønningen klar: at være i stand til at lokalisere specifikke begivenheder eller molekyler i det cellulære landskab.
Når helcellebilleddannelse er påkrævet, er det muligt at sy forskellige tomogrammer, forudsat at den samlede påførte dosis ikke overstiger strålingsskadegrænsen. Normalt er den deponerede dosis til opsamling af få tomogrammer på den samme celle langt under grænsen ved den opnåelige opløsning (109 Gy), og der kræves derfor ingen specifik strategi for at sænke dosis, selv om denne er prøve- og eksperimentafhængig. I tilfælde af intensiv dataindsamling såsom spektrotomografi ville det faktisk være nødvendigt at minimere dosis, og der ville være behov for praktisk dataindsamling og specifikke behandlingsstrategier.
Cryo-SXT har flere begrænsninger, som bør nævnes her. Den første er den velkendte manglende kile, som er iboende ved at bruge flade prøvestøtter. Kapillærprøvestøtter, der tillader 180 graders rotation, er tidligere blevet brugt og bruges stadig på nogle anlæg, men de præsenterer også ulemper såsom en fattig kontrast på grund af glasabsorptionen og begrænsningen af at bruge celler i suspension. En måde at mindske effekten af den manglende kile på er ved at udføre dobbelt vippetomografi. Dette er faktisk muligt på Mistral beamline i dag. Den anden begrænsning er indstillet af Fresnel zone plade linsen, der anvendes i sådanne mikroskoper. Dette objektiv indstiller den ultimative opløsning, der kan opnås, og dybdeskadslen (DoF), som begge er tæt beslægtede. Dette indebærer, at forøgelse af opløsningen vil mindske DoF, mens cellens tykkelse ofte vil være større. For eksempel vil en 40 nm linse i teorien have en DoF på 3 μm og en opløsning på 24,4 nm halv tonehøjde. Kompromiset mellem opløsning og DoF er derfor strategisk, og valget af linsen afhænger af typen af cellen30,31. Endelig er operationelle TXM’er over hele verden langt fra at være ideelle mikroskoper, og der gøres en indsats for at forbedre de optiske systemer for at nå de teoretiske forventninger. Endelig kan visualisering og segmentering af de rekonstruerede volumener udføres med specifikke softwareværktøjer 25,32,33,34.
Sammenfattende tillader cryo-SXT billeddannelse af celler kvantitativt ved medium opløsning (25-30 nm halv tonehøjde) og i statistiske tal (få titalls tomogrammer pr. Dag). Dette gør det muligt at opnå organisering, distribution og dimension af organeller under specifikke forhold, for eksempel under patogeninfektion eller sygdomme, på præcise tidspunkter eller efter bestemte behandlinger. Det er derfor en nyttig komplementær biologisk billeddannelsesteknik til de mere almindelige elektron- og synlige lysmikroskopier, der hver især tackler et specifikt udvalg af prøvedimensioner og opløsning. Cryo-SXT anvendes ofte i korrelative tilgange, der involverer fluorescens i synligt lys, men andre cryo korrelative strategier er også mulige.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dette projekt har modtaget støtte fra Europa-Kommissionens Horizon 2020 iNEXT-Discovery-projekt og EU’s Horizon 2020-forsknings- og innovationsprogram under Marie Skłodowska-Curie-tilskudsaftale nr. 75439.
Materials
| Amira | Thermo Fisher | Software for segmentation | |
| Au Holey Carbon Films finder grids | (Quantifoil Micro Tools Gmb | R 2/2 Au G200F1 | Au Holey Carbon Films finder grids |
| Au nanoparticles | BBI Group, Cardiff, UK | Au nanoparticles 100nm | 100 nm Au nanoparticles (NPs) at Mistral (Alba) |
| Au nanoparticles | BBI Group, Cardiff, UK | Au nanoparticles 250nm | 250 nm Au nanoparticles (NPs) at B24 (Diamond) |
| Axio Scope A1 | Zeiss | 430035 9060 | Fluorescence microscope |
| Blotting No.1 filter paper | Whatman | WHA10010155 | Blotting filter |
| Bsoft | Software for projection alignment, reconstruction and visualization (Heymann et al., 2008) | ||
| Chimera | Software for segmentation (Pettersen et al. 2004) | ||
| Cryo-EM Glow Discharge Set | PELCO easiGlow | 91000S | Glow Discharge Cleaning System |
| Cu Holey Carbon Films finder grids | (Quantifoil Micro Tools Gmb | R 2/2Cu G200F1 | Cu Holey Carbon Films finder grids |
| Fetal calf serum | Sigma | F9665 | Heat Inactivated, sterile-filtered, suitable for cell culture |
| ImageJ | Software for image processing and analysis in Java (NIH & LOCI University of Wisconsin) | ||
| IMOD | Software for projection alignment, reconstruction and visualization (Kremer et al., 1996) | ||
| Leica EM GP Grid Plunger | Leica | 16706401 | Automatic Plunge Freezer EM |
| LINKAM cryo-stage | Linkam Scientific Instruments | CMS 196 | Cryo-Correlative Microscopy Stage |
| MIB | Software for segmentation (Belevich et al. 2016) | ||
| P100 Petri dish | Sigma | P6106 | Treated for cell culture and sterile |
| P60 Petri dish | Sigma | D8054 | Treated for cell culture and sterile |
| Polylysin | Sigma | P4707 | Poly-L-lysine solution 0.01%, sterile-filtered |
| Soft X-Ray microscope 0.25-1.2keV | Xradia | NCT-SB | Transmission soft X-Ray microscope |
| SURVOS | Software for segmentation (Luengo et al. 2017) | ||
| Tomo3d | Software for reconstruction (SIRT, WBP) (Agulleiro et al. 2011) | ||
| TomoJ | Software for reconstruction (ART) (Messaoudi et al., 2007) | ||
| XM Data Explorer | Zeiss | TXM software |
References
- Carrascosa, J. L., et al. Cryo X-ray tomography of vaccinia virus membranes and inner compartments. Journal of Structural Biology. 168, 234-239 (2009).
- Uchida, M., et al. Soft X-ray tomography of phenotypic switching and the cellular response to antifungal peptoids in Candida albicans. PNAS. 106, 19375-19380 (2009).
- Schneider, G., et al. Three-dimensional cellular ultrastructure resolved by X-ray microscopy. Nature Methods. 10, 1-3 (2010).
- Chichón, F. J., et al. Cryo nano-tomography of vaccinia virus infected cells. Journal of Structural Biology. 177, 202-211 (2012).
- Groen, J., Conesa, J. J., Valcárcel, R., Pereiro, E. The cellular landscape by soft X-ray tomography. Biophysical Reviews. 11, 611-619 (2019).
- Kepsutlu, B., et al. Cells undergo major changes in the quantity of cytoplasmic organelles after uptake of gold nanoparticles with biologically relevant surface coatings. ACS Nano. 14, 2248-2264 (2020).
- Natterer, F. . The mathematics of computerized tomography. , (1986).
- Weiss, D., et al. Computed tomography of cryogenic biological specimens based on X-ray microscopic images. Ultramicroscopy. 84, 185-197 (2000).
- Clowney, E. J., et al. Nuclear aggregation of olfactory receptor genes governs their monogenic expression. Cell. 151, 724-737 (2012).
- Duke, E. M. H., et al. Imaging endosomes and autophagosomes in whole mammalian cells using correlative cryo-fluorescence and cryo-soft X-ray microscopy (cryo-CLXM). Ultramicroscopy. 143, 77-87 (2014).
- Cinquin, B., et al. Putting molecules in their place. Journal of Cellular Biochemistry. 115, 209-216 (2014).
- Hagen, C., et al. Multimodal nanoparticles as alignment and correlation markers in fluorescence/soft X-ray cryo-microscopy/tomography of nucleoplasmic reticulum and apoptosis in mammalian cells. Ultramicroscopy. 146, 46-54 (2014).
- Pérez-Berná, A. J., et al. Structural changes in cells images by soft X-ray cryo-tomography during Hepatitis C virus infection. ACS Nano. 10, 6597-6611 (2016).
- Chiappi, M., et al. Cryo-soft X-ray tomography as a quantitative three-dimensional tool to model nanoparticle:cell interaction. Journal of Nanobiotechnology. 14, 15 (2016).
- Varsano, N., et al. Development of correlative cryo-soft X-ray tomography and stochastic reconstruction microscopy. A study of cholesterol crystal early formation in cells. Journal of the American Chemical Society. 138, 14931-14940 (2016).
- Kounatidis, I., et al. Correlative cryo-structured illumination fluorescence microscopy and soft X-ray tomography elucidates reovirus intracellular release pathway. Cell. 182, 1-16 (2020).
- Kapishnikov, S., et al. Mode of action of quinoline antimalarial drugs in red blood cells infected by Plasmodium falciparum revealed in vivo. PNAS. 116 (46), 22946-22952 (2019).
- Conesa, J. J., et al. Unambiguous intracellular localization and quantification of a potent iridium anticancer compound by correlative 3D cryo X-ray imaging. Angewandte Chemie International Edition. 59, 1270-1278 (2020).
- Gal, A., et al. Native-state imaging of calcifying and noncalcifying microalgae reveals similarities in their calcium storage organelles. PNAS. 115 (43), 11000-11005 (2018).
- Procopio, A., et al. Chemical fingerprint of Zn-hydroxyapatite in the early stages of osteogenic differentiation. ACS Central Science. 5, 1449-1460 (2019).
- Kahil, K., et al. Cellular pathways of calcium transport and concentration toward mineral formation in sea urchin larvae. PNAS. 117 (49), 30957-30965 (2020).
- Conesa, J. J., et al. Intracellular nanoparticles mass quantification by near-edge absorption soft X-ray nanotomography. Scientific Reports. 6, 22354 (2016).
- Otón, J., Sorzano, C. O. S., Maribini, R., Pereiro, E., Carazo, J. M. Measurement of the modulation transfer function of an X-ray microscope based on multiple Fourier orders analysis of a Siemens star. Optics Express. 23 (8), 9567-9572 (2015).
- Otón, J., et al. Characterization of transfer function, resolution and depth of field of a soft X-ray microscope applied to tomography enhancement by Wiener deconvolution. Biomedical Optics Express. 7, 5092-5103 (2016).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116, 71-76 (1996).
- Heymann, J. B., Cardone, G., Winkler, D. C., Steven, A. C. Computational resources for cryo-electron tomography in Bsoft. Journal of Structural Biology. 161, 232-242 (2008).
- Messaoudi, C., et al. Three-dimensional chemical mapping by EFTEM-TomoJ including improvement of SNR by PCA and ART reconstruction of volume by noise supression. Microscopy Microanalysis. 19, 1669-1677 (2013).
- Agulleiro, J. I., Fernández, J. J. Fast tomographic reconstruction on multicore computers. Bioinformatics. 27, 582-583 (2011).
- Toro-Nahuelpan, M., et al. Tailoring cryo-electron microscopy grids by photo-micropatterning for in-cell structural studies. Nature Methods. 17, 50-54 (2020).
- Attwood, D. . Soft X-rays and Extreme Ultraviolet Radiation. Principles and Applications. , (2000).
- Howells, M., Jacobsen, C., Warwick, T. . Principles and Applications of Zone Plate X-ray. Microscopes. Science of Microscopy. , (2007).
- Pettersen, E. F., et al. UCSF Chimera–a visualization system for exploratory research and analysis. Journal of Computational Chemistry. 25, 1605-1612 (2004).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy image browser: a platform for segmentation and analysis of multidimensional datasets. PLOS Biology. 14 (1), 1002340 (2016).
- Luengo, I., et al. SuRVoS: Super-region volume segmentation workbench. Journal of Structural Biology. 198, 43-53 (2017).

