Fluorimetria a scansione nano-differenziale per lo screening nella scoperta di piombo basata su frammenti
Summary
Il monitoraggio delle variazioni della temperatura di fusione di una proteinabersaglio (ad es.saggio di spostamento termico, TSA) è un metodo efficiente per lo screening di librerie di frammenti di poche centinaia di composti. Presentiamo un protocollo TSA che implementa la fluorimetria a scansione nano-differenziale assistita da robotica (nano-DSF) per il monitoraggio della fluorescenza intrinseca del triptofano e la retrodepersione della luce per lo screening dei frammenti.
Abstract
I saggi di spostamento termico (TSA) esaminano come la temperatura di fusione (Tm) di una proteina bersaglio cambia in risposta ai cambiamenti nel suo ambiente(ad esempio,composizione tampone). L’utilità della TSA, e in particolare della fluorimetria a scansione nano-differenziale (nano-DSF), è stata stabilita nel corso degli anni, sia per trovare condizioni che aiutano a stabilizzare una proteina specifica sia per esaminare il legame del ligando monitorando i cambiamenti nel Tmapparente . Questo documento presenta uno screening efficiente della libreria di frammenti Diamond-SGC-iNEXT Poised (DSi-Poised) (768 composti) mediante l’uso di nano-DSF, monitorando Tm per identificare potenziali legami di frammenti. I prerequisiti relativi alla qualità e alla concentrazione delle proteine per l’esecuzione di esperimenti nano-DSF sono brevemente delineati seguiti da un protocollo passo-passo che utilizza un distributore robotico da nano litri comunemente usato nei laboratori di biologia strutturale per preparare i campioni richiesti in piastre a 96 pozzetti. Il protocollo descrive come le miscele di reagenti vengono trasferite ai capillari necessari per le misurazioni nano-DSF. Inoltre, questo documento fornisce protocolli per misurare la denaturazione termica (monitoraggio della fluorescenza intrinseca del triptofano) e l’aggregazione (monitoraggio della diffusione della luce) e le fasi successive per il trasferimento e l’analisi dei dati. Infine, vengono discussi esperimenti di screening con tre diversi target proteici per illustrare l’uso di questa procedura nel contesto delle campagne di scoperta dei lead. Il principio generale del metodo descritto può essere facilmente trasferito ad altre librerie di frammenti o adattato ad altri strumenti.
Introduction
I programmi di scoperta di farmaci spesso iniziano con lo screening di composti chimici per la loro capacità di interagire e / o modificare la funzione dei bersagli farmacologici, il più delle volte proteine. I cosiddetti “successi” che si trovano in tali schermi, gettarono le basi per la scoperta di nuovi lead e candidati allo sviluppo, e per la maggior parte dei nuovi farmaci che vengono concessi in licenza in questi giorni. La disponibilità di metodi ad alto rendimento è quindi indispensabile per vagli un numero enorme di bersagli disponibili con un numero enorme di composti diversi per identificare rapidamente il loro legame stretto o la loro capacità di modulare una funzione specifica del bersaglio. Dopo che gli hit sono stati identificati, le combinazioni hit-target più promettenti vengono spinte in una vasta pipeline di sviluppo di farmaci utilizzando altre tecnologie, spesso costose e dispendiose in termini di tempo, per comprendere le “relazioni struttura-attività” (SAR).
Gli approcci di biologia strutturale, come quelli offerti dai programmi di accesso finanziati dall’UE “Infrastructure for NMR, EM, and X-rays for Translational Research” (iNEXT) e dal suo attuale successore iNEXT-Discovery, sono spesso utilizzati per studiare le interazioni di numerosi composti in modo estremamente dettagliato, migliorando al contempo l’affinità e le proprietà farmacologiche dei colpi iniziali tipicamente diversi cicli di chimica sintetica1. I composti di piombo che emergono da queste campagne “da hit to lead” diventano candidati allo sviluppo e entrano in studi preclinici. La metodologia di screening molecolare ben sviluppata può essere approssimativamente classificata in due approcci, vale a dire la scoperta del piombo basata sul ligando (LBLD) e la scoperta del piombo basata su frammenti (FBLD). Nelle campagne LBLD, i recettori proteici vengono sottoposti a screening con poche migliaia di ligandi scelti a mano (in base alla struttura dei ligandi naturali o alla struttura del bersaglio), o con molte decine di migliaia di composti in librerie di ligandi simili a farmaci che coprono una grande porzione di spazio chimico.
Di solito, i composti vengono testati per la loro attività inibitoria in un test di attività, in genere monitorando una funzione enzimatica. Nelle campagne FBLD2,3,4,5,tuttavia, alcune centinaia di composti che sono tipicamente più piccoli dei farmaci (100-200 Dalton) vengono testati per la loro capacità di legare direttamente il bersaglio, senza l’uso di un test di attività. Questo legame potrebbe interferire con l’attività del bersaglio e può essere misurato con molti metodi biofisici che riportano direttamente sulla capacità dei frammenti di legarsi al bersaglio, o con metodi strutturali come la cristallografia a raggi X6 e la spettroscopia di risonanza magnetica nucleare7e, più recentemente, anche la crio-microscopia elettronica. Quando i frammenti si legano in luoghi diversi che sono vicini l’uno all’altro sulla proteina, i diversi frammenti di legame, di solito a bassa affinità, possono essere combinati razionalmente chimicamente per creare un piccolo insieme di lead che possono essere studiati in modo più dettagliato. Ciò si traduce spesso in composti più potenti e di maggiore affinità e questa metodologia ha iniziato a produrre molecole importanti con potenziale clinico. La scelta di una libreria di frammenti “ideale” che sfrutta efficacemente i gruppi chimici è stata un’area di ricerca attiva per molti anni8,9,10.
Mentre l’enfasi iniziale era sulla copertura dell’intero spazio chimico, l’attenzione successiva si è concentrata sul consentire alla combinazione chimica a valle di colpi di frammento di produrre composti di piombo. Tale ricerca ha portato alle cosiddette biblioteche “in bilico”. Questi contengono frammenti con almeno un gruppo funzionale che consentono una chimica sintetica di follow-up rapida ed economica per progressi efficienti nello studio del SAR. Una delle attività catalizzato da iNEXT è stata quella di aggiornare la libreria in bilico sviluppata dai ricercatori del Diamond Light Source and Structural Genomics Consortium. Questo sforzo congiunto ha portato alla libreria DSi-Poised11, che è stata convalidata anche all’interno di iNEXT12. Successivamente, questa libreria è stata allineata con la disponibilità di composti nel REAL Database di Enamine Ltd., un’organizzazione di ricerca chimica e produttrice di grandi collezioni di blocchi di costruzione e librerie composte per lo screening. DSi-Poised è ora disponibile per chiunque per l’acquisto, ma anche disponibile in molti laboratori partner di iNEXT-Discovery per progetti di screening dei frammenti supportati.
Le tecnologie di cristallografia a raggi X di fascia alta e di biologia strutturale NMR hanno entrambi i loro vantaggi e svantaggi per FBLD. Entrambi richiedono campioni target isolati e producono i dettagli atomici ad alta risoluzione necessari per FBLD. Tuttavia, i cristalli sono necessari per la cristallografia a raggi X e i frammenti si legano a cavità nelle regioni proteiche ben ordinate che non sono coinvolte nella costruzione del reticolo cristallino tridimensionale. La NMR della soluzione spesso produce risultati diversi dalla cristallografia a raggi X, in quanto non è influenzata dall’ambiente cristallino ed è buona nel rilevare il legame anche in regioni proteiche parzialmente ordinate. Tuttavia, mentre gli esperimenti NMR basati su ligando sono relativamente veloci, richiedono ancora una notevole quantità di tempo e materiale e possono essere eseguiti di routine solo per bersagli o domini proteici relativamente piccoli. Allo scopo di dare priorità ai composti per esperimenti cristallografici o NMR, sono stati utilizzati approcci biofisici13,14,15.
Poiché la strumentazione recente e i protocolli computazionali consentono uno screening cristallografico efficiente per FBLD determinando le strutture e analizzando ~ 1.000 frammenti in modo molto efficiente, questa prioritizzazione è diventata meno essenziale nella ricerca basata sui raggi X. Per NMR, tuttavia, rimane auspicabile utilizzare esperimenti più economici e veloci per dare priorità allo screening della biblioteca e risparmiare tempo sugli strumenti su apparecchiature di fascia più alta. Allo stesso tempo, l’utilizzo di una combinazione di tecnologie essenzialmente diverse può fornire una conferma indipendente di eventi di legame, o anche ulteriori colpi che non vengono raccolti utilizzando solo la cristallografia o il metodo NMR. Le tecniche cristallografiche e NMR richiedono entrambe attrezzature molto costose e spesso possono essere eseguite solo in strutture esterne dedicate con l’aiuto di esperti locali altamente qualificati. Inoltre, la corretta analisi dei risultati richiede anche un’elevata competenza. Mentre programmi come iNEXT e iNEXT-Discovery stanno democratizzando l’accesso a tali strutture16, è stato riconosciuto che lo screening FBLD economico, veloce e ad alto rendimento con altri metodi può incoraggiare programmi di screening dei farmaci in una gamma molto più ampia di laboratori. Tali risultati possono quindi essere utilizzati come indicazione per costruire collaborazioni con chimici medicinali e per dare priorità agli esperimenti di screening più costosi ai composti più promettenti se le strutture NMR e cristallografiche impongono restrizioni sul numero di composti che possono essere sottoposti a screening.
La TSA forma un metodo biofisico rapido, efficiente e relativamente economico e accessibile17 che può essere utilizzato per lo screening FBLD. È stato utilizzato in più contesti, dall’aiutare a trovare condizioni proteiche stabili per gli studidi cristallizzazione 18, alla ricerca di composti che si legano a bersagli specifici nelle cellule19. I TSA sono stati utilizzati anche per misurare le costanti di dissociazione per i ligandi che legano le proteine bersaglio, poiché il legame del ligando spesso porta ad alterazioni della stabilità termica. In tutti i TSA, il cambiamento della temperatura di denaturazione di una proteina (la sua stabilità) viene misurato in funzione di un lento aumento della temperatura. Un modo efficace per seguire la denaturazione delle proteine al momento del riscaldamento è il DSF o Thermofluor, che quantifica la tempra fluorescente di un colorante idrofobo (tipicamente Sypro Orange) all’interazione con regioni idrofobiche esposte di proteine che si dispiegano a causa dell’aumento della temperatura.
Nano-DSF si riferisce tipicamente alla misurazione della stabilità termica delle proteine in assenza di coloranti esterni. Uno dei primi strumenti che ha offerto questa possibilità è stato l’OPTIM1000 che misura un ampio spettro di intensità luminosa e la diffusione della luce di un campione. Questa macchina ha permesso la misurazione simultanea dello sviluppo proteico (tipicamente dopo la fluorescenza del triptofano) e dell’aggregazione proteica (poiché le nanoparticelle formate provocano un aumento della diffusione della luce a ~ 400 nm). Successivamente, il Prometeo introdusse l’uso della retroriflessione per misurare l’aggregazione e il rilevamento sensibile del segnale di fluorescenza, consentendo lo screening di basse concentrazioni proteiche con una buona sensibilità20. La sezione seguente descrive come Prometheus è stato utilizzato per dimostrare un protocollo di screening dei frammenti per rilevare gli hit per diversi bersagli proteici. Una breve introduzione sulla qualità e la quantità di proteine previste è seguita da un protocollo passo-passo per preparare, eseguire e analizzare gli esperimenti di screening dei frammenti. I risultati dello screening per tre proteine sono stati mostrati come dati di esempio ottenuti nell’ambito delle collaborazioni iNEXT-Discovery.
Protocol
Representative Results
Discussion
Questo protocollo descrive un metodo di throughput medio-alto per lo screening di librerie di frammenti utilizzando alcuni strumenti di robotica e misurazione comuni. Schermate come quelle descritte in questo protocollo possono essere eseguite di routine dalla NKI Protein Facility di Amsterdam, ad esempio come servizio iNEXT-Discovery, spesso anche gratuitamente per gli utenti dopo l’applicazione della proposta e la revisione tra pari. In questi casi, la libreria DSi-Poised può essere fornita dalla struttura, ma l’uso di altre librerie può anche essere discusso nel contesto di ogni diversa applicazione utente e contratto di servizio. La scelta degli strumenti in questo protocollo rappresenta soluzioni pratiche per molti laboratori, ma non deve essere considerata come un gold standard. Per misurare la stabilità termica della proteina bersaglio per lo screening dei frammenti sono raccomandati metodi privi di etichette per misurare la stabilità termica della proteina bersaglio, piuttosto che metodi che utilizzano etichette sensibili all’ambiente per rilevare lo svolgimento in un termociclatore a reazione a catena della polimerasi a trascrizione inversa.
I metodi senza etichetta, come quello qui presentato con lo strumento Prometheus, hanno alcuni vantaggi: usano basse quantità di proteine, spesso un paio di ordini di grandezza in meno; possono essere utilizzati per misurare simultaneamente lo scattering del campione e quindi l’aggregazione; e le etichette utilizzate per rilevare lo svolgimento in altri approcci possono interagire in modo diverso con ciascun frammento, con conseguenti artefatti di misurazione. Questo protocollo è stato descritto nel contesto del robot Mosquito, che consente il pipettaggio di un volume molto piccolo di campione (0,3 μL) che non può essere fatto manualmente. Il Mosquito è un robot popolare, presente in molti laboratori che lavorano su progetti di biologia strutturale e scoperta di farmaci; tuttavia, il protocollo può chiaramente utilizzare approcci alternativi per il pipettaggio a basso volume.
Le librerie di frammenti contengono composti disciolti in DMSO. Una delle sfide iniziali è trovare la concentrazione ottimale di DMSO alla quale la proteina rimane stabile e i composti rimangono solubili. Ciò comporta l’esecuzione delle misurazioni a varie concentrazioni di DMSO per determinare le condizioni ottimali per lo screening. La diluizione da proteina a frammento qui utilizzata si traduce in concentrazioni di DMSO dello 0,2%; la maggior parte delle proteine sono abbastanza stabili in queste condizioni. La quantità di proteine necessaria per effettuare lo screening per la libreria di 768 composti è di ~ 2-3 mg in totale, poiché le misurazioni vengono in genere eseguite a basse concentrazioni proteiche (0,2 mg mL-1). Lavorare con concentrazioni proteiche relativamente basse non solo riduce i costi di produzione delle proteine, ma riduce anche le possibilità di precipitazione delle proteine. La bassa concentrazione proteica non influisce sul rilevamento del legame dei frammenti, poiché la concentrazione dei frammenti nell’esperimento è di ~ 2 mM, consentendo di identificare anche i leganti deboli.
Poiché il rilevamento della transizione di fusione in questi esperimenti si basa sull’intensità della fluorescenza, un aspetto critico è determinare il potere di eccitazione del laser a cui effettuare le misurazioni. L’interazione dei composti con la proteina può (i) non avere alcun effetto sulla sua fluorescenza intrinseca, (ii) provocare la tempra o (iii) aumentare la sua fluorescenza intrinseca. Inoltre, lavorare con basse concentrazioni proteiche significa che il conteggio di fluorescenza per la proteina nativa sarebbe basso. Il potere di eccitazione deve quindi essere regolato in modo tale da poter misurare la maggior parte dei campioni. Il profilo di scattering di ogni esecuzione fornisce informazioni importanti sugli effetti di aggregazione che potrebbero essere attivati dall’aggiunta di qualsiasi frammento. Inoltre, l’effetto della temperatura sulla solubilità del composto può essere visto anche sul profilo di scattering.
Inaspettatamente, per molti composti è stato osservato che lo scattering in realtà diminuiva con l’aumentare della temperatura (Figura 6). È quindi importante esaminare sia la curva di transizione di fusione che il profilo di scattering di accompagnamento per decidere l’affidabilità di ciascun esperimento, specialmente per quei frammenti che sono considerati candidati per misurazioni più impegnative mediante cristallografia a raggi X o spettroscopia NMR, o addirittura considerati come hit per la chimica di follow-up. Una limitazione specifica del metodo ai fini dello screening dei frammenti è che molti frammenti nella libreria DSi-Poised hanno una fluorescenza intrinseca significativa, a volte anche oltre il limite di saturazione del rivelatore, e quindi questi non possono essere adeguatamente sottoposti a screening per il legame target anche a basso potere di eccitazione. Un altro punto da notare per questo metodo è che può essere utilizzato solo con proteine contenenti residui di triptofano.
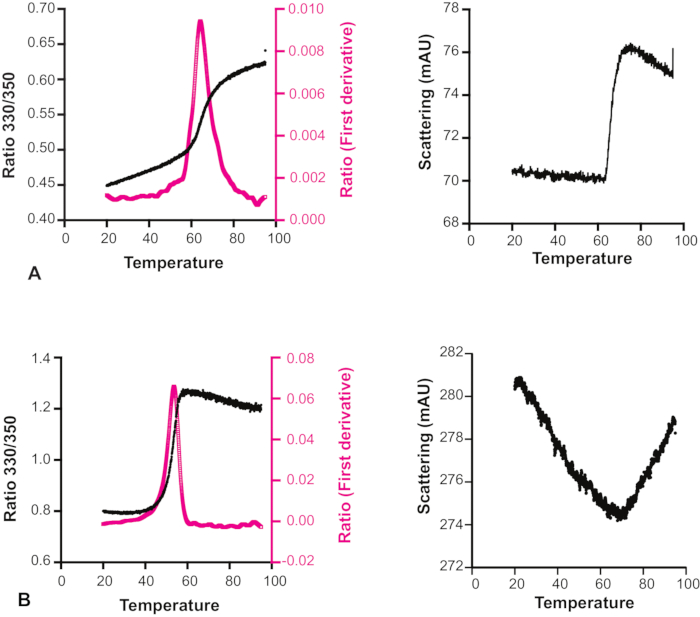
Figura 6: Effetto della temperatura sulla solubilità del composto. Curva di transizione di fusione e profilo di scattering di Hec1 con due composti diversi. (A) Il profilo di dispersione mostra che per questo campione la solubilità non è influenzata dalla temperatura. (B) Il profilo di dispersione mostra che la solubilità di questo campione aumenta con l’aumentare della temperatura. La curva di transizione di fusione in questo caso non è quindi affidabile. Fare clic qui per visualizzare una versione più grande di questa figura.
Una domanda aperta è cosa dovrebbe essere considerato come un cambiamento significativo in Tm, non da una prospettiva matematica, ma dal punto di vista pratico: quale cambiamento in Tm è importante considerare come indicativo del legame di un ligando a una proteina? In questi esempi, si vedono spostamenti inferiori a 1 °C per il 74% dei frammenti per legare Hec1, il 66% per Mps1 e il 53% per Nsp5. Considerare 1 °C come “cambiamento significativo” difficilmente fornirebbe risultati degni di essere perseguiti dalla chimica di follow-up. Nei grafici di panoramica (Figura 5), sono stati considerati contenitori di 1, 2, 5 o più di 5 gradi di spostamento Tm, sia positivi che negativi. Ciò richiede modifiche in base a ciascun caso specifico per fornire una buona panoramica e consentire un processo decisionale informato, determinando il passo successivo. In particolare, per alcune proteine, sono state osservate sia la stabilizzazione che la destabilizzazione del bersaglio a seconda dei frammenti considerati. Entrambi gli eventi sono interessanti, in quanto entrambi possono essere il risultato del legame dei frammenti, ed entrambi possono portare a una buona molecola di follow-up per manipolare il comportamento delle proteine.
Rimane un’ultima domanda, vale a dire, “cosa definisce un colpo utile?”. In realtà, la risposta dipende dalla situazione specifica. Ad esempio, per Hec1, tutti i frammenti che stabilizzano la proteina di oltre 2 gradi o la destabilizzano di più di 5 sono stati comunicati ai nostri collaboratori di chimica, che hanno progettato nuove molecole basate su questi colpi. Per Nsp5, tuttavia, i colpi più destabilizzanti sono stati comunicati ai nostri collaboratori NMR per confermare i colpi derivati dal nanoDSF con esperimenti NMR. In altre parole, i risultati dello screening ottenuti da questo protocollo dovrebbero essere analizzati con cautela e in modo dipendente dal contesto, prendendo decisioni informate basate sulla domanda specifica e sulla metodologia circostante. In ogni caso, il metodo qui descritto è un approccio complementare alle metodologie esistenti come lo screening a raggi X e nmR, che può mirare a confermare, dare priorità o dare nuove idee per le campagne di chimica.
Tabella supplementare S1: Elenco dei tamponi utilizzati per lo screening delle proteine. Abbreviazioni: HEPES = acido 4-(2-idrossietil)-1-piperazineetansolfonico; DTT = ditiotritolo; MOBS = acido 4-(N-morfolino)butanisolfonico. Fare clic qui per scaricare questa tabella.
Tabella supplementare S2: Proprietà per la piastra MRC a 2 pozzi per l’uso con robot nanodispenser. Fare clic qui per scaricare questa tabella.
Tabella supplementare S3: Proprietà per piastra con fondo a V a 96 pozzi per l’uso con robot nanodispenser. Fare clic qui per scaricare questa tabella.
Tabella supplementare S4: Il tampone, la concentrazione proteica ela Tm delle proteine discusse nei risultati rappresentativi. Abbreviazioni: DTT = ditiotritolo; Hec1 = Altamente espresso nella proteina Cancer 1; Mps1 = chinasi monopolare del fuso 1; Nsp5 = proteasi simile a SARS-CoV-2 3C. Fare clic qui per scaricare questa tabella.
File supplementare 1: Parametri di panoramica–datidi esempio. Fare clic qui per scaricare questo file.
File supplementare 2: valori Tm e ΔTm per 406 frammenti-dati di esempio. Fare clic qui per scaricare questo file.
Disclosures
The authors have nothing to disclose.
Acknowledgements
“Questo lavoro ha beneficiato dell’accesso alla NKI Protein Facility, un centro Instruct-ERIC. Il sostegno finanziario è stato fornito da iNEXT, numero 653706, e iNEXT-Discovery, progetto numero 871037, finanziato dal programma Horizon 2020 della Commissione Europea”.
Materials
| ClearVue Sheets | Molecular Dimensions | adhesive sealing film for protein plate | |
| CORNING 6570 Aluminium Sealing Tape | CORNING | adhesive sealing film for fragment plate | |
| DSi poised library | Enamine | Fragment library containing 768 compounds used in this study | |
| Elisa Reagent Reservior | ThermoFisher Scientific | 15075 | Reagent reservior used for pipetting the protein |
| Greiner round (U) bottom plates | Cat. No. 650201 | Fragments supplied in these plates | |
| Mosquito type X1 | sptlabtech | Part nr- 3019-0003 | Nanolitre dispenser |
| MRC 2-well crystallization plate | MRC96T-PS | ||
| Pierce ELISA Reagent Reservoirs | Pierce | ||
| Prometheus High Sensitivity capillaries | Catalog PR-C006 | ||
| Prometheus NT.48 nanoDSF | Nanotemper | Catalog nr PR001 (+ Aggregation Detection Optics, catalog nr PR-AGO) | nanoDSF and light back scattering |
| Prometheus Standard capillary type | Catalog PR-C002 | ||
| TX-1000 | Thermoscientific | Centrifuge for plates | |
References
- Hoffer, L., Muller, C., Roche, P., Morelli, X. Chemistry-driven hit-to-lead optimization guided by structure-based approaches. Molecular Informatics. 37 (9-10), 1800059 (2018).
- Lamoree, B., Hubbard, R. E. Current perspectives in fragment-based lead discovery (FBLD). Essays in Biochemistry. 61 (5), 453-464 (2017).
- Ress, D. C., Congreve, M., Murray, C. W., Carr, R. Fragment-based lead discovery. Nature Reviews Drug Discovery. 3 (8), 660-672 (2004).
- Carr, R. A. E., Congreve, M., Murray, C. W., Rees, D. C. Fragment-based lead discovery: Leads by design. Drug Discovery Today. 10 (14), 987-992 (2005).
- Bradley, A. R., et al. The SGC beyond structural genomics: Redefining the role of 3D structures by coupling genomic stratification with fragment-based discovery. Essays in Biochemistry. 61 (5), 495-503 (2017).
- Davies, T. G., Tickle, I. J. Fragment screening using X-ray crystallography. Topics in Current Chemistry. 317, 33-59 (2012).
- Ma, R., Wang, P., Wu, J., Ruan, K. Process of fragment-based lead discovery – A perspective from NMR. Molecules. 21 (7), 854 (2016).
- Troelsen, N. S., Clausen, M. H. Library design strategies to accelerate fragment-based drug discovery. Chemistry. 26 (50), 11391-11403 (2020).
- Shi, Y., von Itzstein, M. How size matters: Diversity for fragment library design. Molecules. 24 (15), 2838 (2019).
- Taylor, A., Doak, B. C., Scanlon, M. J. Design of a fragment-screening library. Methods in Enzymology. 610, 97-115 (2018).
- Cox, O. B., et al. A poised fragment library enables rapid synthetic expansion yielding the first reported inhibitors of PHIP(2), an atypical bromodomain. Chemical Science. 7, 2322-2330 (2016).
- Sreeramulu, S., et al. NMR quality control of fragment libraries for screening. Journal of Biomolecular NMR. 74 (10-11), 555-563 (2020).
- Pfaff, S. J., Chimenti, M. S., Kelly, M. J. S., Arkin, M. R. Biophysical methods for identifying fragment-based inhibitors of protein-protein interactions. Methods in Molecular Biology. 1278, 587-613 (2015).
- Fattori, D., Squarcia, A., Bartoli, S. Fragment-based approach to drug lead discovery: Overview and advances in various techniques. Drugs in R & D. 9 (4), 217-227 (2008).
- Winter, A., et al. Biophysical and computational fragment-based approaches to targeting protein-protein interactions: Applications in structure-guided drug discovery. Quarterly Reviews of Biophysics. 45 (4), 383-426 (2012).
- Boelens, R., et al. iNEXT: a European facility network to stimulate translational structural biology. FEBS Letters. 592 (12), 1909-1917 (2018).
- Zhang, R., Monsma, F. Fluorescence-based thermal shift assays. Current Opinion in Drug Discovery and Development. 13 (4), 389-402 (2010).
- Boivin, S., Kozak, S., Meijers, R. Optimization of protein purification and characterization using Thermofluor screens. Protein Expression and Purification. 91 (2), 192-206 (2013).
- Martinez Molina, D., Nordlund, P. The cellular thermal shift assay: a novel biophysical assay for in situ drug target engagement and mechanistic biomarker studies. Annual Review of Pharmacology and Toxicology. 56, 141-161 (2016).
- Bruce, D., Cardew, E., Freitag-Pohl, S., Pohl, E. How to stabilize protein: stability screens for thermal shift assays and nano differential scanning fluorimetry in the Virus-X Project. Journal of Visualized Experiments JoVE. (144), e58666 (2019).

