DNAメチル化のバイオインフォマティクス分析へのサンプル調製:肥満および関連形質研究の関連戦略
Summary
本研究では、マイクロアレイ技術によって得られたDNAメチル化データを管理するワークフローについて説明する。このプロトコルは、サンプル調製からデータ分析までの手順を示しています。すべての手順が詳細に説明されており、ビデオは重要な手順を示しています。
Abstract
肥満は生活習慣に直接関連しており、脂肪生成および脂質貯蔵プロセスの変化を引き起こす可能性のあるDNAメチル化変化と関連しており、疾患の発症に寄与する。我々は、肥満の有無にかかわらず、患者の選択からエピジェネティックなデータ分析までの完全なプロトコルを実証する。プロトコルのすべてのステップは、パイロットスタディでテストおよび検証されました。32人の女性がこの研究に参加し、15人がボディマス指数(BMI)(45.1 ± 5.4kg/m2)に従って肥満に分類された。17人がBMIに従って肥満なしに分類された(22.6±1.8kg/m2)。肥満の群では、脂肪量に関連する564のCpGサイトが線形回帰分析によって同定された。CpGサイトはプロモーター領域にあった。鑑別解析では、肥満患者において470のCpGs低メチル化部位と94の高メチル化部位が見つかった。最も低メチル化された濃縮経路は、 RUNX、 WNT シグナル伝達、および低酸素症に対する応答であった。高メチル化経路は、インスリン分泌、グルカゴンシグナル伝達、およびCa2+に関連していた。我々は、このプロトコルがDNAメチル化パターンおよび形質関連DNAメチル化を効果的に同定したと結論付ける。これらのパターンは、脂肪生成および脂質貯蔵に影響を及ぼす遺伝子発現の変化と関連している可能性がある。その結果、肥満のライフスタイルがヒトDNAのエピジェネティックな変化を促進する可能性があることが確認されました。
Introduction
大規模なオミックス技術は、慢性疾患の研究においてますます使用されている。これらの方法の興味深い特徴は、生成された大量のデータが科学コミュニティに利用可能であることです。したがって、研究間の技術的比較を可能にするためにプロトコルを標準化する要求が生じている。本研究は、DNAメチル化データを取得および分析するためのプロトコルの標準化を、適用された例としてパイロット研究を用いて示唆している。
負のエネルギー消費は現代人のライフスタイルにおいて優勢であり、脂肪組織の過剰な蓄積をもたらし、その結果、肥満の発症につながる¹。座りがちな食事、高カロリーの食事、ストレスの多いルーチンなど、多くの要因が肥満率を高めています。世界保健機関(WHO)は、2016年に19億人の成人が肥満であったと推定しており、これは世界人口の20%以上が30kg/m2以上のBMI2を有することを意味します。2018年の最新の更新では、米国(USA)における肥満の有病率が42%よりも高いことが明らかになりました3。
エピジェネティクスとは、染色体領域の構造的適応であり、変化した活性状態を登録、シグナル伝達、または永続化させるものです4。DNAメチル化は、シトシン-グアノシンジヌクレオチド部位(CpGサイト)における可逆的な化学変化であり、5-メチルシトシン-pG(5mCpG)を形成する。 DNA5,6,7,8への転写機構へのアクセスを調節することによって遺伝子発現を調節することができる。この文脈では、どのCpGサイトが肥満関連形質と関連しているかを理解することが不可欠です9。多くの因子が部位特異的DNAメチル化を支持または防止することができる。このプロセスに必要な酵素、例えばDNAメチルトランスフェラーゼ10(DNTM)やテン-イレブン転座(TET)は、環境暴露下でDNAメチル化または脱メチル化を促進することができます11。
ここ数年のDNAメチル化研究への関心の高まりを考えると、各質問に正確に答えるために最も適切な分析戦略を選択することは、研究者の本質的な関心事でした12,13,14。450K DNAメチル化アレイは最も一般的な方法であり、DNAメチル化プロファイルを決定するために360以上の出版物14で使用されています。既知の遺伝子の99%に位置する最大485,000 CpGのメチル化を決定することができます15。ただし、このアレイは廃止され、850,000のCpGサイトをカバーするEPICに置き換えられました。本プロトコルは、450KおよびEPIC16,17,18の両方に適用することができる。
このプロトコルは、 図1 に段階的に示されており、集団選択、サンプリング、実験準備、DNAメチル化パイプライン、およびバイオインフォマティクス分析のステップで構成されています。私たちの研究室で行われたパイロット研究は、提案されたプロトコルのステップを説明するためにここで実証されています。
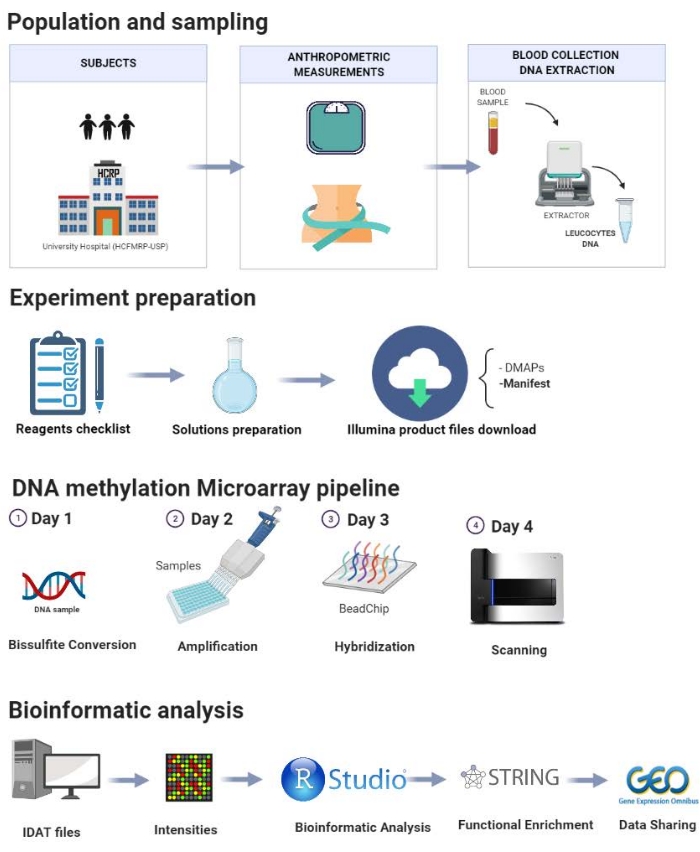
図 1: 提示されたプロトコルの概略。 この図の拡大版を表示するには、ここをクリックしてください。
Protocol
Representative Results
Discussion
DNAメチル化アレイは、その費用対効果比のためにDNAメチル化にアクセスするために最も使用される方法です14。本研究は、ブラジルのコホートで実施されたパイロット研究においてDNAメチル化を評価するために市販のマイクロアレイプラットフォームを用いた詳細なプロトコールを記載した。パイロット研究から得られた結果は、プロトコルの有効性を確認した。 図3 は、サンプル比較可能性および完全な亜硫酸水素塩変換32を示す。
品質管理ステップとして、ChAMP アルゴリズムはフィルタリング プロセス中に CpGs サイトを除外することを推奨しました。プローブを除外する目的は、データ分析を改善し、バイアスを排除することです。低品質のCpGs(p値は0.05未満)は、データセット内の実験ノイズを除去するために削除されました。ターゲットは密度プロット分析で渡されたままでした。Zhou33は 、ミスマッチ、多型シトシンのメチル化の誤解、およびI型プローブ設計のスイッチカラーを引き起こすことを避けるために、SNPsの近くのCpGsをフィルタリングすることの重要性を説明しました34。また、XY染色体はインプリンティングによって差次的に影響を受けるため、HeissとJust35 は、女性ではハイブリダイゼーションの問題が交絡因子である可能性があるため、これらのプローブをフィルタリングすることの重要性を強化しました35。
DMAPsの有効期限、ホルムアミドの開封日、無水エタノールの分析品質、および総白血球数は、プロトコルにおける重要なステップとみなされます。
さらに、我々の観察によると、バイオインフォマティクス解析を行う上では、細胞型推定が不可欠です。ハウスマン法は、Tianの研究30に記載されているように細胞型推定を行う。この方法は、顆粒球、単球、B細胞、T細胞などの最も重要な細胞型の割合を予測できる473の特定のCpGサイトに基づいています36。ChAMP パッケージから推奨されている関数 “myRefbase” を使用しました。推定後、ChAMP アルゴリズムはベータ値を調整し、データセットからこのバイアスを除去します。このステップは、この集団が慢性炎症状態のために白血球にかなりの違いを有するため、肥満に焦点を当てた研究において重要である。
メソッドの変更とトラブルシューティングに関して、一般的なPCRシールの元のキャップマップのみを変更しました。各遠心分離プロセスの後、シールを新しいものに交換した。標準のヒートシールは使えず、板の周りのアルミホイルで対応しました。
市販のアッセイはエピジェネティック研究のゴールドスタンダードと考えられてきましたが、プロトコルの限界の1つは、独自のブランドの試薬および機器の特異性である可能性があります37,38,39,40。別の制限は、実験41の正しい進行の同定を可能にする指標の欠如である。
本プロトコールの標準化は、エピジェネティック研究の優れた指針であり、プロセス中のヒューマンエラーを減らし、異なる研究間のデータ分析と比較可能性を成功に導きます。
我々の結果によると、DNAメチル化実験は肥満の有無を比較する研究に適しています43。また、提案されたバイオインフォマティクス解析は、質の高いデータを提供し、大規模な研究で検討することができます。
SVD分析を用いて、肥満関連形質(BMI、WC、FM)がDNAメチル化データの変動性に影響することを同定した。有意な結果として、細胞型推定は、ナチュラルキラー細胞(NK)およびB細胞の両方が肥満の女性において肥満のない女性よりも高かったことを示している(図5)。これらの細胞の数が多いことは、これらの個体の低悪性度の炎症状態によって説明できる44。我々は、肥満患者が脂肪量に関連する遺伝子のプロモーター領域に低メチル化および高メチル化CpGを有することを観察した。ほとんどの部位は低メチル化されており、これはこれらの個体における活性酸素種(ROS)レベルの自然な増加に関連している可能性がある。この酸化ストレス状態は、ジヌクレオチド部位におけるグアニン摂動を促進し、8−ヒドロキシ−2’−デオキシグアノシン(8−OHdG)を形成し、5mCp−8−OHdGジヌクレオチド部位を生じさせ、TET酵素の動員を引き起こす可能性がある。これらすべての事象は、異なる作用機序によるDNAの低メチル化および高メチル化の促進に関与している可能性がある45。
さらに、脂肪形成の割合は肥満の個体で増加するようであり、新しい細胞の約10%が古い細胞に46,47。肥満環境を強調するエピジェネティックな貢献は、細胞の増殖および分化速度を変化させ、脂肪塊の発達を促進する可能性がある48。エピジェネティックな変化はまた、脂肪生成プログラムに影響を与え、その発達を促進または制限し得る。一次転写因子(PPARγまたはC/EBPα)または多タンパク質複合体の集合体は、エピジェネティック修飾酵素を含むかまたは除外することによって操作される下流プロモーター領域に位置し、超メチル化または低メチル化を介して遺伝子発現を調節する45。PPARγ経路は、この研究で富化された遺伝子を有するWNT経路を変化させることが以前に記載されていた。脂肪形成中にWNTシグナル伝達がどのように起こるかはまだ不明であるが、最近の研究では、特に肥満条件下で脂肪細胞代謝に不可欠な役割を果たす可能性があることが報告されている49。
Disclosures
The authors have nothing to disclose.
Acknowledgements
Yuan Tian博士(tian.yuan@ucl.ac.uk)がChAMPパッケージに関するすべての疑問に答えてくれたことに感謝します。我々はまた、この論文の技術的および科学的問題の両方に対するギリェルメ・テレス氏への貢献に感謝する。彼はエピジェネティクスとビデオのキャプチャとフォーマット技術(guilherme.telles@usp.br)に関して重要な検討をしました。消耗品の資金調達:サンパウロ研究財団(FAPESP)(#2018/24069-3)および国家科学技術開発評議会(CNPq:#408292/2018-0)。個人的な資金:(FAPESP:#2014/16740-6)および高等教育スタッフ開発のための調整からのアカデミックエクセレンスプログラム(CAPES:88882.180020/2018-01)。データは、制限なく公開され、自由に利用できるようになります。NYN(電子メール:nataliayumi@usp.br)またはCBN(電子メール:carla@fmrp.usp.br)へのアドレス対応。
Materials
| Absolute ethanol | J.T. Baker | B5924-03 | |
| Agarose gel | Kasvi | K9-9100 | |
| Electric bioimpedance | Quantum BIA 450 Q – RJL System | ||
| Ethylenediaminetetraacetic acid (EDTA) | Corning | 46-000-CI | |
| EZ DNA Methylation-Gold kit | ZymoResearch, Irvine, CA, USA | D5001 | |
| Formamide | Sigma | F9037 | |
| FMS—Fragmentation solution | Illumina | 11203428 | Supplied Reagents |
| HumanMethylation450 BeadChip | Illumina | ||
| Maxwell Instrument | Promega, Brazil | AS4500 | |
| MA1—Multi-Sample Amplification 1 Mix | Illumina | 11202880 | Supplied Reagents |
| MicroAmp Optical Adhesive Film | Thermo Fisher Scientific | 201703982 | |
| MSM—Multi-Sample Amplification Master Mix | Illumina | 11203410 | Supplied Reagents |
| NaOH | F. MAIA | 114700 | |
| PB1—Reagent used to prepare BeadChips for hybridization | Illumina | 11291245 | Supplied Reagents |
| PB2—Humidifying buffer used during hybridization | Illumina | 11191130 | Supplied Reagents |
| 2-propanol | Emsure | 10,96,34,01,000 | |
| RA1—Resuspension, hybridization, and wash solution | Illumina | 11292441 | Supplied Reagents |
| RPM—Random Primer Mix | Illumina | 15010230 | Supplied Reagents |
| STM—Superior Two-Color Master Mix | Illumina | 11288046 | Supplied Reagents |
| TEM—Two-Color Extension Master Mix | Illumina | 11208309 | Supplied Reagents |
| Ultrapure EDTA | Invitrogen | 155576-028 | |
| 96-Well Reaction Plate with Barcode (0.1mL) | ByoSystems | 4346906 | |
| 96-Well Reaction Plate with Barcode (0.8mL) | Thermo Fisher Scientific | AB-0859 | |
| XC1—XStain BeadChip solution 1 | Illumina | 11208288 | Supplied Reagents |
| XC2—XStain BeadChip solution 2 | Illumina | 11208296 | Supplied Reagents |
| XC3—XStain BeadChip solution 3 | Illumina | 11208392 | Supplied Reagents |
| XC4—XStain BeadChip solution 4 | Illumina | 11208430 | Supplied Reagents |
References
- Manna, P., Jain, S. Obesity, oxidative stress, adipose tissue dysfunction, and the associated health risks: causes and therapeutic strategies. Metabolic Syndrome and Related Disorders. 13 (10), 423-444 (2015).
- . Obesity Available from: https://www.who.int/health-topics/obesity#tab=tab_1 (2017)
- Adult Obesity Facts. Center for Disease Control and Prevention Available from: https://www.cdc.gov/obesity/data/adult.html (2021)
- Bird, A. Perceptions of epigenetics. Nature. 396, (2007).
- Kouzarides, T. Chromatin modifications, and their function. Cell. 128 (4), 693-705 (2007).
- Berger, F. The strictest usage of the term epigenetic. Seminars in Cell & Developmental Biology. 19 (6), 525-526 (2008).
- Henikoff, S., Greally, J. M. Epigenetics, cellular memory, and gene regulation. Current Biology. 26 (14), 644-648 (2016).
- Laker, R. C., et al. Transcriptomic and epigenetic responses to short-term nutrient-exercise stress in humans. Scientific Reports. 7 (1), 1-12 (2017).
- Wahl, S., et al. Epigenome-wide association study of body mass index, and the adverse outcomes of adiposity. Nature. 541, 81-86 (2017).
- Jin, B., Robertson, K. D. DNA methyltransferases, DNA damage repair, and cancer. Epigenetic Alterations in Oncogenesis. 754, 3-29 (2013).
- Jang, H. S., Shin, W. J., Lee, J. E., Do, J. T. CpG and non-CpG methylation in epigenetic gene regulation and brain function. Genes. 8 (6), 148 (2017).
- Shen, L., Waterland, R. A. Methods of DNA methylation analysis. Current Opinion in Clinical Nutrition & Metabolic Care. 10 (5), 576-581 (2007).
- Olkhov-Mitsel, E., Bapat, B. Strategies for discovery and validation of methylated and hydroxymethylated DNA biomarkers. Cancer Medicine. 1, 237-260 (2012).
- Kurdyukov, S., Bullock, M. DNA methylation analysis: choosing the right method. Biology. 5 (1), 3 (2016).
- Yong, W. -. S., Hsu, F. -. M., Chen, P. -. Y. Profiling genome-wide DNA methylation. Epigenetics & Chromatin. 9 (1), 1-16 (2016).
- Dugué, P. A., et al. Alcohol consumption is associated with widespread changes in blood DNA methylation: Analysis of cross-sectional and longitudinal data. Addiction Biology. 1, 12855 (2021).
- Colicino, E., et al. Blood DNA methylation sites predict death risk in a longitudinal study of 12, 300 individuals. Aging. 14, 14092-14124 (2020).
- Karlsson, L., Barbaro, M., Ewing, E., Gomez-Cabrero, D., Lajic, S. Genome-wide investigation of DNA methylation in congenital adrenal hyperplasia. The Journal of Steroid Biochemistry and Molecular Biology. 201, 105699 (2020).
- Ribeiro, R. R., Guerra-Junior, G., de Azevedo Barros-Filho, A. Bone mass in schoolchildren in Brazil: the effect of racial miscegenation, pubertal stage, and socioeconomic differences. Journal of Bone and Mineral Metabolism. 27 (4), 494-501 (2009).
- Filozof, C., et al. Obesity prevalence and trends in Latin-American countries. Obesity Reviews. 2 (2), 99-106 (2001).
- Chadid, S., Kreger, B. E., Singer, M. R., Bradlee, M. L., Moore, L. L. Anthropometric measures of body fat and obesity-related cancer risk: sex-specific differences in Framingham Offspring Study adults. International Journal of Obesity. 44 (3), 601-608 (2020).
- Nicoletti, C. F., et al. DNA methylation pattern changes following a short-term hypocaloric diet in women with obesity. European Journal of Clinical Nutrition. 74 (9), 1345-1353 (2020).
- Assessing Your Weight. Centers for Disease Control and Prevention Available from: https://www.cdc.gov/healthyweight/assessing/index.html (2020)
- Giavarina, D., Lippi, G. Blood venous sample collection: Recommendations overview and a checklist to improve quality. Clinical Biochemistry. 50 (10-11), 568-573 (2017).
- Duijs, F. E., Sijen, T. A rapid and efficient method for DNA extraction from bone powder. Forensic Science International: Reports. 2, 100099 (2020).
- . Infinium HD Methylation Assay, Manual Protocol Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/chemistry_documentation/infinium_assays/infinium_hd_methylation/infinium-hd-methylation-guide-15019519-01.pdf (2015)
- Serrano, J., Snuderl, M. Whole genome DNA methylation analysis of human glioblastoma using Illumina BeadArrays. Glioblastoma. , 31-51 (2018).
- Leti, F., Llaci, L., Malenica, I., DiStefano, J. K. Methods for CpG methylation array profiling via bisulfite conversion. Disease Gene Identification. 1706, 233-254 (2018).
- Noble, A. J., et al. A validation of Illumina EPIC array system with bisulfite-based amplicon sequencing. PeerJ. 9, 10762 (2021).
- Tian, Y., et al. ChAMP: updated methylation analysis pipeline for Illumina BeadChips. Bioinformatics. 33, 3982-3984 (2017).
- Turinsky, A. L., et al. EpigenCentral: Portal for DNA methylation data analysis and classification in rare diseases. Human Mutation. 41 (10), 1722-1733 (2020).
- . The Chip Analysis Methylation Pipeline Available from: https://www.bioconductor.org/packages/release/bioc/vignettes/ChAMP/inst/doc/ChAMP.html (2020)
- Zhou, W., Laird, P. W., Shen, H. Comprehensive characterization, annotation and innovative use of Infinium DNA methylation BeadChip probes. Nucleic Acids Research. 45 (4), 22 (2017).
- Wanding, Z., Triche, T. J., Laird, P. W., Hui, S. SeSAMe: reducing artifactual detection of DNA methylation by Infinium BeadChips in genomic deletions. Nucleic Acids Research. 46 (20), 123 (2018).
- Heiss, J. A., Just, A. C. Improved filtering of DNA methylation microarray data by detection p values and its impact on downstream analyses. Clinical Epigenetics. 11, 15 (2019).
- Houseman, E. A., et al. DNA methylation arrays as surrogate measures of cell mixture distribution. BMC Bioinformatics. 13, 86 (2012).
- Valavanis, I., Sifakis, E. G., Georgiadis, P., Kyrtopoulos, S., Chatziioannou, A. A. A composite framework for the statistical analysis of epidemiological DNA methylation data with the Infinium human Methylation 450K BeadChip. IEEE Journal of Biomedical and Health Informatics. 18 (3), 817-823 (2014).
- Sun, N., Zhang, J., Zhang, C., Shi, Y., Zhao, B., Jiao, A., Chen, B. Using Illumina Infinium HumanMethylation 450K BeadChip to explore genomewide DNA methylation profiles in a human hepatocellular carcinoma cell line. Molecular Medicine Reports. 18 (5), 4446-4456 (2018).
- Moran, S., Arribas, C., Esteller, M. Validation of a DNA methylation microarray for 850,000 CpG sites of the human genome enriched in enhancer sequences. Epigenomics. 8 (3), 389-399 (2016).
- Bibikova, M., et al. High density DNA methylation array with single CpG site resolution. Genomics. 98 (4), 288-295 (2011).
- Lehne, B., et al. A coherent approach for analysis of the Illumina HumanMethylation450 BeadChip improves data quality and performance in epigenome-wide association studies. Genome Biology. 16 (1), 1-12 (2015).
- Wang, J., Zhang, H., Rezwan, F. I., Relton, C., Arshad, S. H., Holloway, J. W. Pre-adolescence DNA methylation is associated with BMI status change from pre- to post-adolescence. Clinical Epigenetics. 25, 64 (2021).
- Maugeri, A. The effects of dietary interventions on DNA methylation: implications for obesity management. International Journal of Molecular Sciences. 21, 8670 (2020).
- DeFuria, J., et al. B cells promote inflammation in obesity and type 2 diabetes through regulation of T-cell function and an inflammatory cytokine profile. Proceedings of the National Academy of Sciences. 110 (13), 5133-5138 (2013).
- Kietzmann, T., Petry, A., Shvetsova, A., Gerhold, J. M., Görlach, A. The epigenetic landscape related to reactive oxygen species formation in the cardiovascular system. British Journal of Pharmacology. 174, 1533-1554 (2017).
- Chase, K., Sharma, R. P. Epigenetic developmental programs and adipogenesis: implications for psychotropic induced obesity. Epigenetics. 8 (11), 1133-1140 (2013).
- Spalding, K. L., et al. Dynamics of fat cell turnover in humans. Nature. 453, 783-787 (2008).
- Ross, S. E., et al. Inhibition of adipogenesis by Wnt signaling. Science. 289, 950-953 (2000).
- Bagchi, D. P., et al. Wnt/β-catenin signaling regulates adipose tissue lipogenesis and adipocyte-specific loss is rigorously defended by neighboring stromal-vascular cells. Molecular Metabolism. 42, 101078 (2020).

