Ein Workflow zur Optimierung der Formulierung von Lipid-Nanopartikeln (LNP) unter Verwendung von Experimenten mit designten Mischungsprozessen und selbstvalidierten Ensemblemodellen (SVEM)
Summary
Dieses Protokoll bietet einen Ansatz zur Formulierungsoptimierung über Mischungs-, kontinuierliche und kategoriale Studienfaktoren, der subjektive Entscheidungen bei der Konstruktion des Versuchsdesigns minimiert. Für die Analysephase wird ein effektives und einfach zu handhabendes Modellierungsanpassungsverfahren verwendet.
Abstract
Wir stellen einen Quality by Design (QbD)-ähnlichen Ansatz zur Optimierung von Lipid-Nanopartikel-Formulierungen (LNP) vor, der darauf abzielt, Wissenschaftlern einen zugänglichen Arbeitsablauf zu bieten. Die inhärente Beschränkung in diesen Studien, bei der die molaren Verhältnisse von ionisierbaren, Helfer- und PEG-Lipiden bis zu 100 % betragen müssen, erfordert spezielle Design- und Analysemethoden, um diese Mischungsbeschränkung zu berücksichtigen. Wir konzentrieren uns auf Lipid- und Prozessfaktoren, die üblicherweise bei der LNP-Designoptimierung verwendet werden, und bieten Schritte, die viele der Schwierigkeiten vermeiden, die traditionell bei der Planung und Analyse von Mischungsprozessexperimenten auftreten, indem wir raumfüllende Designs verwenden und den kürzlich entwickelten statistischen Rahmen der selbstvalidierten Ensemblemodelle (SVEM) verwenden. Neben der Erstellung optimaler Formulierungen erstellt der Workflow auch grafische Zusammenfassungen der angepassten statistischen Modelle, die die Interpretation der Ergebnisse vereinfachen. Die neu identifizierten Kandidatenformulierungen werden mit Bestätigungsläufen bewertet und können optional im Rahmen einer umfassenderen Zweitphasenstudie durchgeführt werden.
Introduction
Lipid-Nanopartikel-Formulierungen (LNP) für In-vivo-Gentransfersysteme enthalten im Allgemeinen vier Bestandteile von Lipiden aus den Kategorien ionisierbare, Helfer- und PEG-Lipide 1,2,3. Unabhängig davon, ob diese Lipide allein oder gleichzeitig mit anderen Nicht-Mischungsfaktoren untersucht werden, erfordern Experimente für diese Formulierungen “Mischungs”-Designs, da – bei einer Kandidatenformulierung – das Erhöhen oder Verringern des Verhältnisses eines der Lipide notwendigerweise zu einer entsprechenden Abnahme oder Zunahme der Summe der Verhältnisse der anderen drei Lipide führt.
Zur Veranschaulichung wird angenommen, dass wir eine LNP-Formulierung optimieren, die derzeit eine festgelegte Rezeptur verwendet, die als Benchmark behandelt wird. Ziel ist es, die Wirksamkeit des LNP zu maximieren, während sekundär die durchschnittliche Partikelgröße minimiert wird. Die Untersuchungsfaktoren, die im Experiment variiert werden, sind die molaren Verhältnisse der vier konstituierenden Lipide (ionisierbar, Cholesterin, DOPE, PEG), das N:P-Verhältnis, die Flussrate und der ionisierbare Lipidtyp. Die ionisierbaren Lipide und Hilfslipide (einschließlich Cholesterin) dürfen über einen größeren Bereich des molaren Verhältnisses, 10-60 %, variieren als PEG, das in dieser Abbildung von 1-5 % variiert wird. Die Benchmark-Formulierungsrezeptur und die Bereiche der anderen Faktoren sowie deren Rundungsgranularität sind in der Ergänzungsdatei 1 angegeben. Für dieses Beispiel sind die Wissenschaftler in der Lage, 23 Durchläufe (einzigartige Chargen von Partikeln) an einem einzigen Tag durchzuführen und möchten diese als Stichprobengröße verwenden, wenn sie die Mindestanforderungen erfüllt. Simulierte Ergebnisse für dieses Experiment finden Sie in den Ergänzungsdateien 2 und 3 .
Rampado und Peer4 haben ein aktuelles Übersichtspapier zum Thema geplante Experimente zur Optimierung von Nanopartikel-basierten Drug-Delivery-Systemen veröffentlicht. Kauffman et al.5 betrachteten LNP-Optimierungsstudien unter Verwendung von fraktionsfaktoriellen und definitiven Screening-Designs6; Diese Arten von Versuchsplänen können jedoch keine Mischungsbeschränkung berücksichtigen, ohne auf die Verwendung ineffizienter “Schlupfvariablen”7 zurückzugreifen, und werden in der Regel nicht verwendet, wenn Mischungsfaktoren vorhanden sind 7,8. Stattdessen werden traditionell “optimale Designs”, die in der Lage sind, eine Mischungsbeschränkung zu berücksichtigen, für Mischungsprozessexperimenteverwendet 9. Diese Designs zielen auf eine benutzerdefinierte Funktion der Studienfaktoren ab und sind nur dann optimal (in einem von mehreren möglichen Sinne), wenn diese Funktion die wahre Beziehung zwischen den Studienfaktoren und den Antworten erfasst. Beachten Sie, dass im Text zwischen “optimalen Designs” und “optimalen Formulierungskandidaten” unterschieden wird, wobei sich letztere auf die besten Formulierungen beziehen, die durch ein statistisches Modell identifiziert wurden. Optimale Designs bringen drei Hauptnachteile für Experimente mit Mischprozessen mit sich. Erstens, wenn der Wissenschaftler bei der Spezifizierung des Zielmodells keine Wechselwirkung der Studienfaktoren antizipiert, wird das resultierende Modell verzerrt und kann minderwertige Kandidatenformulierungen hervorbringen. Zweitens platzieren optimale Versuchspläne die meisten Durchläufe an der äußeren Begrenzung des Faktorraums. In LNP-Studien kann dies zu einer großen Anzahl von verlorenen Durchläufen führen, wenn sich die Partikel an keinen Extremen der Lipid- oder Prozesseinstellungen korrekt bilden. Drittens bevorzugen Wissenschaftler oft experimentelle Läufe im Inneren des Faktorraums, um ein modellunabhängiges Gefühl für die Wirkungsfläche zu gewinnen und den Prozess direkt in bisher unerforschten Regionen des Faktorraums zu beobachten.
Ein alternatives Designprinzip besteht darin, mit einem raumfüllenden Design 10 eine annähernd gleichmäßige Abdeckung des (mischungsbeschränkten) Faktorraumsanzustreben. Diese Versuchspläne opfern eine gewisse experimentelle Effizienz im Vergleich zu optimalen Versuchsplänen9 (unter der Annahme, dass der gesamte Faktorraum zu gültigen Formulierungen führt), bieten jedoch mehrere Vorteile in einem Kompromiss, die in dieser Anwendung nützlich sind. Das raumfüllende Design macht keine a priori Annahmen über die Struktur der Antwortfläche; Dies gibt ihm die Flexibilität, unvorhergesehene Beziehungen zwischen den Studienfaktoren zu erfassen. Dadurch wird auch die Entwurfserstellung optimiert, da keine Entscheidungen darüber getroffen werden müssen, welche Regressionsterme hinzugefügt oder entfernt werden sollen, wenn die gewünschte Laufgröße angepasst wird. Wenn einige Designpunkte (Rezepte) zu fehlgeschlagenen Formulierungen führen, ermöglichen raumfüllende Designs die Modellierung der Fehlergrenze über die Studienfaktoren und unterstützen gleichzeitig statistische Modelle für die Studienantworten über die erfolgreichen Faktorkombinationen. Schließlich ermöglicht die innere Abdeckung des Faktorraums eine modellunabhängige grafische Erkundung der Wirkungsfläche.
Zur Visualisierung des Mischungsfaktor-Unterraums eines Mischungsprozess-Experiments werden spezielle dreieckige “ternäre Plots” verwendet. Abbildung 1 motiviert diese Verwendung: Im Punktwürfel, in dem jeweils drei Zutaten im Bereich von 0 bis 1 liegen dürfen, werden die Punkte, die die Bedingung erfüllen, dass die Summe der Zutaten gleich 1 ist, rot hervorgehoben. Die Mischungsbeschränkung für die drei Zutaten reduziert den möglichen Faktorraum auf ein Dreieck. In LNP-Anwendungen mit vier Mischungsbestandteilen erzeugen wir sechs verschiedene ternäre Diagramme, um den Faktorraum darzustellen, indem wir zwei Lipide gleichzeitig gegen eine “Andere”-Achse auftragen, die die Summe der anderen Lipide darstellt.
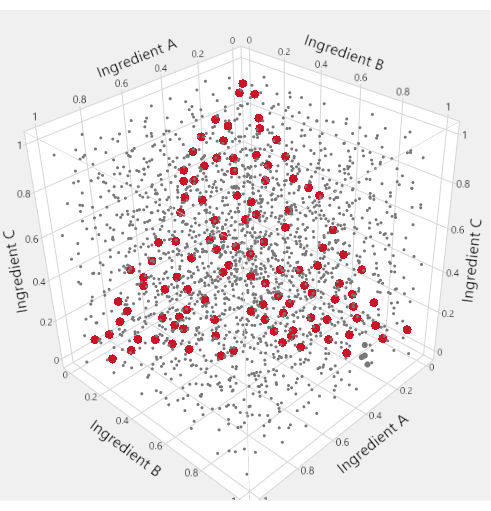
Abbildung 1: Bereiche des Dreiecksfaktors. Im raumfüllenden Diagramm innerhalb des Würfels stellen die kleinen grauen Punkte Formulierungen dar, die nicht mit der Mischungsbeschränkung übereinstimmen. Die größeren roten Punkte liegen auf einem Dreieck, das in den Würfel eingeschrieben ist, und stellen Formulierungen dar, für die die Mischungsbeschränkung erfüllt ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Zusätzlich zu den Lipidmischungsfaktoren gibt es oft einen oder mehrere kontinuierliche Prozessfaktoren wie das N:P-Verhältnis, die Pufferkonzentration oder die Durchflussrate. Es können kategoriale Faktoren vorhanden sein, wie z. B. der ionisierbare Lipidtyp, der Helferlipidtyp oder der Puffertyp. Ziel ist es, in In-vivo-Studien eine Formulierung (eine Mischung aus Lipiden und Einstellungen für Prozessfaktoren) zu finden, die ein gewisses Maß an Wirksamkeit maximiert und/oder physikalisch-chemische Eigenschaften wie die Minimierung der Partikelgröße und des PDI (Polydispersitätsindex), die Maximierung der prozentualen Verkapselung und die Minimierung von Nebenwirkungen – wie z. B. Körpergewichtsverlust – verbessert. Selbst wenn man von einem vernünftigen Benchmark-Rezept ausgeht, kann es bei einer Änderung der genetischen Nutzlast oder bei der Berücksichtigung von Änderungen der Prozessfaktoren oder Lipidtypen an einer erneuten Optimierung interessiert sein.
Cornell7 bietet einen definitiven Text über die statistischen Aspekte von Mischungs- und Mischprozessexperimenten, wobei Myers et al.9 eine hervorragende Zusammenfassung der relevantesten Mischungsdesign- und Analysethemen für die Optimierung liefern. Diese Arbeiten können Wissenschaftler jedoch mit statistischen Details und Fachterminologie überfrachten. Moderne Software für die Planung und Analyse von Experimenten bietet eine robuste Lösung, die die meisten LNP-Optimierungsprobleme ausreichend unterstützt, ohne sich auf die entsprechende Theorie berufen zu müssen. Während kompliziertere Studien oder Studien mit hoher Priorität immer noch von der Zusammenarbeit mit einem Statistiker profitieren und eher optimale als platzfüllende Designs verwenden können, ist es unser Ziel, den Komfort der Wissenschaftler zu verbessern und die Optimierung von LNP-Formulierungen zu fördern, ohne an ineffiziente OFAT-Tests (One-Factor-at-a-Time)11 zu appellieren oder sich einfach mit der ersten Formulierung zufrieden zu geben, die den Spezifikationen entspricht.
In diesem Artikel wird ein Workflow vorgestellt, der statistische Software verwendet, um ein generisches LNP-Formulierungsproblem zu optimieren und Design- und Analyseprobleme in der Reihenfolge anzugehen, in der sie auftreten werden. Tatsächlich wird die Methode für allgemeine Optimierungsprobleme funktionieren und ist nicht auf LNPs beschränkt. Auf dem Weg dorthin werden mehrere häufig auftretende Fragen angesprochen und Empfehlungen gegeben, die auf Erfahrungen und Simulationsergebnissen basieren12. Das kürzlich entwickelte Framework der selbstvalidierten Ensemble-Modelle (SVEM)13 hat den ansonsten fragilen Ansatz zur Analyse von Ergebnissen aus Mix-Prozess-Experimenten erheblich verbessert, und wir verwenden diesen Ansatz, um eine vereinfachte Strategie für die Formulierungsoptimierung bereitzustellen. Während der Arbeitsablauf in einer allgemeinen Art und Weise aufgebaut ist, die mit anderen Softwarepaketen befolgt werden könnte, ist JMP 17 Pro einzigartig, da es SVEM zusammen mit den grafischen Zusammenfassungswerkzeugen bietet, die wir als notwendig erachtet haben, um die ansonsten obskure Analyse von Mischprozessexperimenten zu vereinfachen. Daher werden auch JMP-spezifische Anweisungen im Protokoll bereitgestellt.
SVEM verwendet die gleiche lineare Regressionsmodellgrundlage wie der traditionelle Ansatz, ermöglicht es uns jedoch, langwierige Modifikationen zu vermeiden, die erforderlich sind, um ein “vollständiges Modell” von Kandidateneffekten anzupassen, indem wir entweder einen Vorwärtsselektions- oder einen Penalized Selection-Basisansatz (Lasso) verwenden. Darüber hinaus bietet SVEM eine verbesserte “reduzierte Modellanpassung”, die das Potenzial für die Einbeziehung von Rauschen (Prozess plus analytische Varianz), das in den Daten auftritt, minimiert. Es funktioniert, indem die prognostizierten Modelle gemittelt werden, die sich aus der wiederholten Neugewichtung der relativen Wichtigkeit der einzelnen Durchläufe im Modell 13,14,15,16,17,18 ergeben. SVEM bietet einen Rahmen für die Modellierung von Mix-Prozess-Experimenten, der sowohl einfacher zu implementieren ist als die herkömmliche Single-Shot-Regression als auch qualitativ bessere optimale Formulierungskandidaten liefert12,13. Die mathematischen Details von SVEM würden den Rahmen dieses Artikels sprengen, und selbst eine oberflächliche Zusammenfassung über die relevante Literaturrecherche hinaus würde von seinem Hauptvorteil in dieser Anwendung ablenken: Es ermöglicht ein einfaches, robustes und genaues Click-to-Run-Verfahren für Praktiker.
Der vorgestellte Workflow steht im Einklang mit dem Quality by Design (QbD)19-Ansatz für die pharmazeutische Entwicklung20. Das Ergebnis der Studie wird ein Verständnis der funktionalen Beziehung sein, die die Materialattribute und Prozessparameter mit kritischen Qualitätsmerkmalen (CQAs) verbindet21. Daniel et al.22 diskutieren die Verwendung eines QbD-Frameworks speziell für die RNA-Plattformproduktion: Unser Workflow könnte als Werkzeug innerhalb dieses Frameworks verwendet werden.
Protocol
Representative Results
Discussion
Moderne Software für die Planung und Analyse von Experimenten mit Mischungsprozessen ermöglicht es Wissenschaftlern, ihre Lipid-Nanopartikel-Formulierungen in einem strukturierten Arbeitsablauf zu verbessern, der ineffiziente OFAT-Experimente vermeidet. Der kürzlich entwickelte SVEM-Modellierungsansatz eliminiert viele der obskuren Regressionsmodifikationen und Modellreduktionsstrategien, die Wissenschaftler zuvor möglicherweise mit überflüssigen statistischen Überlegungen abgelenkt haben. Sobald die Ergebnisse ge…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Wir danken dem Herausgeber und den anonymen Gutachtern für Vorschläge, die den Artikel verbessert haben.
References
- Dolgin, E. Better lipids to power next generation of mRNA vaccines. Science. 376 (6594), 680-681 (2022).
- Hou, X., Zaks, T., Langer, R., Dong, Y. Lipid nanoparticles for mRNA delivery. Nature Reviews Materials. 6 (12), 1078-1094 (2021).
- Huang, X., et al. The landscape of mRNA nanomedicine. Nature Medicine. 28, 2273-2287 (2022).
- Rampado, R., Peer, D. Design of experiments in the optimization of nanoparticle-based drug delivery systems. Journal of Controlled Release. 358, 398-419 (2023).
- Kauffman, K. J., et al. Optimization of lipid nanoparticle formulations for mRNA delivery in vivo with fractional factorial and definitive screening designs. Nano Letters. 15, 7300-7306 (2015).
- Jones, B., Nachtsheim, C. J. A class of three-level designs for definitive screening in the presence of second-order effects. Journal of Quality Technology. 43, 1-15 (2011).
- Cornell, J. . Experiments with Mixtures: Designs, Models, and the Analysis of Mixture Data. Wiley Series in Probability and Statistics. , (2002).
- Jones, B. Proper and improper use of definitive screening designs (DSDs). JMP user Community. , (2016).
- Myers, R., Montgomery, D., Anderson-Cook, C. . Response Surface Methodology. , (2016).
- Lekivetz, R., Jones, B. Fast flexible space-filling designs for nonrectangular regions. Quality and Reliability Engineering International. 31, 829-837 (2015).
- Czitrom, V. One-factor-at-a-time versus designed experiments. The American Statistician. 53, 126-131 (1999).
- Karl, A., Wisnowski, J., Rushing, H. JMP Pro 17 remedies for practical struggles with mixture experiments. JMP Discovery Conference. , (2022).
- Lemkus, T., Gotwalt, C., Ramsey, P., Weese, M. L. Self-validated ensemble models for design of experiments. Chemometrics and Intelligent Laboratory Systems. 219, 104439 (2021).
- Gotwalt, C., Ramsey, P. Model validation strategies for designed experiments using bootstrapping techniques with applications to biopharmaceuticals. JMP Discovery Conference. , (2018).
- Xu, L., Gotwalt, C., Hong, Y., King, C. B., Meeker, W. Q. Applications of the fractional-random-weight bootstrap. The American Statistician. 74 (4), 345-358 (2020).
- Ramsey, P., Levin, W., Lemkus, T., Gotwalt, C. SVEM: A paradigm shift in design and analysis of experiments. JMP Discovery Conference Europe. , (2021).
- Ramsey, P., Gaudard, M., Levin, W. Accelerating innovation with space filling mixture designs, neural networks and SVEM. JMP Discovery Conference. , (2021).
- Lemkus, T. Self-Validated Ensemble modelling. Doctoral Dissertations. 2707. , (2022).
- Juran, J. M. . Juran on Quality by Design: The New Steps for Planning Quality into Goods and Services. , (1992).
- Yu, L. X., et al. Understanding pharmaceutical quality by design. The AAPS Journal. 16, 771 (2014).
- Simpson, J. R., Listak, C. M., Hutto, G. T. Guidelines for planning and evidence for assessing a well-designed experiment. Quality Engineering. 25, 333-355 (2013).
- Daniel, S., Kis, Z., Kontoravdi, C., Shah, N. Quality by design for enabling RNA platform production processes. Trends in Biotechnology. 40 (10), 1213-1228 (2022).
- Scheffé, H. Experiments with mixtures. Journal of the Royal Statistical Society Series B. 20, 344-360 (1958).
- Brown, L., Donev, A. N., Bissett, A. C. General blending models for data from mixture experiments. Technometrics. 57, 449-456 (2015).
- Herrera, M., Kim, J., Eygeris, Y., Jozic, A., Sahay, G. Illuminating endosomal escape of polymorphic lipid nanoparticles that boost mRNA delivery. Biomaterials Science. 9 (12), 4289-4300 (2021).
- Lemkus, T., Ramsey, P., Gotwalt, C., Weese, M. Self-validated ensemble models for design of experiments. ArXiv. , 2103.09303 (2021).
- Goos, P., Jones, B. . Optimal Design of Experiments: A Case Study Approach. , (2011).
- Rushing, H. DOE Gumbo: How hybrid and augmenting designs can lead to more effective design choices. JMP Discovery Conference. , (2020).

