Fænotypisk profilering af humane stamcelleafledte dopaminerge neuroner i midthjernen
Summary
Denne protokol beskriver celledyrkning af humane dopaminerge neuroner i midthjernen, efterfulgt af immunologisk farvning og generering af neuronale fænotypiske profiler fra erhvervede mikroskopiske billeder med højt indhold, der muliggør identifikation af fænotypiske variationer på grund af genetiske eller kemiske modulationer.
Abstract
Parkinsons sygdom (PD) er forbundet med en række cellebiologiske processer, der forårsager dopaminerge (mDA) neurontab i midthjernen. Mange nuværende in vitro PD-cellulære modeller mangler kompleksitet og tager ikke højde for flere fænotyper. Fænotypisk profilering i humane inducerede pluripotente stamceller (iPSC)-afledte mDA-neuroner kan løse disse mangler ved samtidig at måle en række neuronale fænotyper i en PD-relevant celletype parallelt. Her beskriver vi en protokol til opnåelse og analyse af fænotypiske profiler fra kommercielt tilgængelige humane mDA-neuroner. Et neuronspecifikt fluorescerende farvningspanel bruges til at visualisere de nukleare, α-synuclein, tyrosinhydroxylase (TH) og mikrotubulusassocierede protein 2 (MAP2) relaterede fænotyper. Den beskrevne fænotypiske profileringsprotokol er skalerbar, da den bruger 384-brøndplader, automatisk væskehåndtering og højkapacitetsmikroskopi. Protokollens anvendelighed er eksemplificeret ved hjælp af sunde donor mDA-neuroner og mDA-neuroner, der bærer den PD-bundne G2019S-mutation i det leucinrige gentagelseskinase 2 (LRRK2) gen. Begge cellelinjer blev behandlet med LRRK2-kinasehæmmeren PFE-360, og fænotypiske ændringer blev målt. Derudover demonstrerer vi, hvordan multidimensionelle fænotypiske profiler kan analyseres ved hjælp af klynger eller maskinlæringsdrevne overvågede klassificeringsmetoder. Den beskrevne protokol vil især interessere forskere, der arbejder med neuronal sygdomsmodellering eller studerer kemiske sammensatte virkninger i humane neuroner.
Introduction
En række cellebiologiske processer forstyrres ved Parkinsons sygdom (PD). For eksempel er mitokondriel dysfunktion, oxidativ stress, proteinnedbrydningsdefekter, forstyrrelse af vesikulær handel og endolysosomal funktion blevet forbundet med dopaminerge (mDA) neurontab i midthjernen, almindeligt observeret i PD1. Derfor synes PD at involvere flere sygdomsmekanismer, der kan interagere med og forværre hinanden. En nyttig måde at undersøge dette mekanistiske samspil på er skabelsen af et omfattende fænotypisk fingeraftryk eller profil af dopaminerge (mDA) neuroner i midthjernen.
Fænotypisk profilering er en tilgang, der involverer oprettelse af en profil af en prøve baseret på en samling af målbare egenskaber, og for det andet involverer det at forudsige en prøve baseret på denne profil 2,3. Målet med profilering er at fange en bred vifte af funktioner, hvoraf nogle måske ikke tidligere har været forbundet med en sygdom eller behandling3. Som følge heraf kan profilering afsløre uventede biologiske processer. Fænotypisk profilering er typisk afhængig af fluorescerende farvede celler, og standardiserede assays, såsom cellemaleri, er blevet udviklet til at skabe fænotypiske profiler4. For nylig er fænotypisk profilering for eksempel blevet anvendt til karakterisering af små molekyler eller nøjagtig forudsigelse af PD-undertyper udelukkende baseret på patientafledte fibroblaster 5,6. På trods af disse fremskridt er fænotypisk profilering sjældent blevet anvendt på postmitotiske differentierede celler, såsom humane inducerede pluripotente stamceller (iPSC)-afledte mDA-neuroner, der udtrykker PD-bundne mutationer såsom LRRK2 G2019S. Væsentlige udfordringer ved iPSC-afledte modeller omfatter tilstedeværelsen af subtile eller variable patologiske træk på tværs af differentieringsbatcher eller genotyper og det faktum, at isolerede PD-fænotyper ikke fanger sygdommens fulde kompleksitet. Selvom iPSC neuronale modeller er fysiologisk relevante, anvendes de sjældent i PD-lægemiddelopdagelsesprocesser på grund af bekymringer om teknisk kompleksitet 7,8.
Vi har tidligere udviklet en robust metode til at måle flere PD-relaterede patofysiologiske fænotyper i humane mDA-neuroner, der både er følsomme over for genetiske og kemiske sammensætningsinducerede fænotypiske ændringer9. Denne artikel beskriver detaljeret en yderligere optimeret version af denne metode til at oprette fænotypiske profiler fra mDA-neuroner (figur 1). Denne protokol har flere fordele i forhold til de tidligere beskrevne fænotypiske profileringsmetoder, såsom brugen af mDA-neuroner af høj kvalitet og teknisk reproducerbarhed. For første gang muliggør denne protokol fænotypisk profilering i fysiologisk relevante postmitotiske mDA-neuroner efter kemiske forstyrrelser på en meget skalerbar måde. Fuldt differentierede og kryopræserverede mDA-neuroner er kommercielt tilgængelige, hvilket signifikant reducerer batch-til-batch-differentieringsvariabilitet. For det andet kan teknisk variabilitet reduceres yderligere ved at anvende et veldefineret eksperimentelt design (dvs. dyrkningsvarighed eller undgå kantbrønde), automatiseret væskehåndtering og automatiseret mikroskopi. Derudover er de indledende trin i fænotypisk profilanalyse ved hjælp af uovervåget klyngedannelse eller overvåget klassificeringsmetoder skitseret her, hvilket angiver, hvordan fænotypiske profileringsdata kan analyseres. Denne protokol vil være nyttig for forskere, der er interesseret i fænotypiske ændringer af mDA-neuroner induceret af genetiske eller kemiske forstyrrelser, specifikt når der kræves en meget skalerbar undersøgelsesopsætning, for eksempel under screeningskampagner, eller når virkningerne af et mindre antal forbindelser skal undersøges, for eksempel for at bestemme toksiske virkninger. Sammenfattende forventes det, at anvendelsen af fænotypisk profilering af humane neuroner er en værdifuld teknik til at studere komplekse sygdomsrelaterede fænotyper og karakterisere de cellulære virkninger af lægemiddelkandidater.
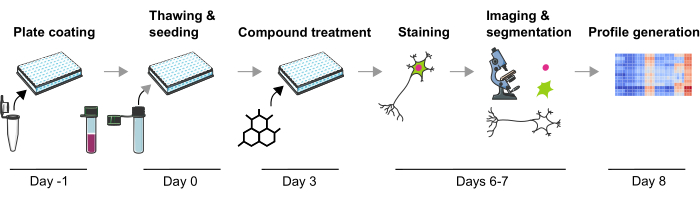
Figur 1: Skematisk skildring af den eksperimentelle protokol til generering af billedbaserede fænotypiske profiler fra humane iPSC-afledte mDA-neuroner. Klik her for at se en større version af denne figur.
Protocol
Representative Results
Discussion
Fænotypisk profilering er en teknik til at måle et stort antal fænotyper i celler ved at anvende fluorescerende farvninger, mikroskopi og billedanalyse3. Fænotypiske profiler kan opnås og sammenlignes på tværs af cellelinjer eller andre eksperimentelle betingelser for at forstå komplekse ændringer i cellulær biologi, der kan gå ubemærket hen, når man bruger en enkelt udlæsning. Her beskriver vi anvendelsen af fænotypisk profilering til humane iPSC-afledte mDA-neuroner, en celletype,…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Forfatterne vil gerne takke alle kolleger hos Ksilink for deres værdifulde hjælp og diskussioner, der fører til udformningen af den præsenterede protokol.
Materials
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse – Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit – Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
References
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson’s disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson’s disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson’s Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson’s Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson’s disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson’s disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson’s disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson’s disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).

