인간 줄기세포 유래 중뇌 도파민 뉴런의 표현형 프로파일링
Summary
이 프로토콜은 인간 중뇌 도파민 뉴런의 세포 배양에 이어 면역학적 염색 및 획득된 현미경 high-content 이미지에서 신경 세포 표현형 프로파일 생성을 설명하여 유전적 또는 화학적 조절로 인한 표현형 변이를 식별할 수 있습니다.
Abstract
파킨슨병(PD)은 중뇌 도파민(mDA) 뉴런 손실을 유발하는 다양한 세포 생물학적 과정과 관련이 있습니다. 현재의 많은 in vitro PD 세포 모델은 복잡성이 부족하고 여러 표현형을 고려하지 않습니다. 인간 유도 만능 줄기 세포(iPSC) 유래 mDA 뉴런의 표현형 프로파일링은 PD 관련 세포 유형에서 다양한 뉴런 표현형을 동시에 측정하여 이러한 단점을 해결할 수 있습니다. 여기서는 상업적으로 이용 가능한 인간 mDA 뉴런에서 표현형 프로파일을 얻고 분석하기 위한 프로토콜을 설명합니다. 뉴런 특이적 형광 염색 패널은 핵, α-시누클레인, 티로신 하이드록실라제(TH) 및 미세소관 관련 단백질 2(MAP2) 관련 표현형을 시각화하는 데 사용됩니다. 설명된 표현형 프로파일링 프로토콜은 384웰 플레이트, 자동 액체 처리 및 고처리량 현미경을 사용하기 때문에 확장 가능합니다. 프로토콜의 유용성은 건강한 기증자 mDA 뉴런과 류신이 풍부한 반복 키나아제 2(LRRK2) 유전자에서 PD 결합 G2019S 돌연변이를 운반하는 mDA 뉴런을 사용하여 예시됩니다. 두 세포주 모두 LRRK2 키나아제 억제제 PFE-360으로 처리하고 표현형 변화를 측정했습니다. 또한 클러스터링 또는 기계 학습 기반 지도 분류 방법을 사용하여 다차원 표현형 프로파일을 분석하는 방법을 보여줍니다. 설명된 프로토콜은 신경 질환 모델링을 연구하거나 인간 신경 세포의 화합물 효과를 연구하는 연구자들에게 특히 흥미로울 것입니다.
Introduction
파킨슨병(PD)에서는 다양한 세포 생물학적 과정이 방해를 받습니다. 예를 들어, 미토콘드리아 기능 장애, 산화 스트레스, 단백질 분해 결함, 소포 이동 및 엔도리소좀 기능 장애는 중뇌 도파민(mDA) 뉴런 손실과 관련이 있으며, PD1에서 일반적으로 관찰됩니다. 따라서 파킨슨병은 서로 상호작용하고 악화시킬 수 있는 여러 질병 기전을 포함하는 것으로 보입니다. 이 기계론적 상호 작용을 조사하는 한 가지 유용한 방법은 중뇌 도파민(mDA) 뉴런의 포괄적인 표현형 지문 또는 프로필을 생성하는 것입니다.
표현형 프로파일링은 측정 가능한 특성의 모음을 기반으로 샘플의 프로필을 생성하는 것과 관련된 접근 방식이며, 둘째, 이 프로필 2,3을 기반으로 샘플에 대한 예측을 수행하는 것을 포함합니다. 프로파일링의 목표는 다양한 특징을 포착하는 것이며, 그 중 일부는 이전에 질병이나 치료와 관련이 없었을 수 있다3. 결과적으로 프로파일링을 통해 예상치 못한 생물학적 과정을 밝힐 수 있습니다. 표현형 프로파일링은 일반적으로 형광 염색된 세포에 의존하며, 표현형 프로파일을 생성하기 위해 셀 페인팅(Cell Painting)과 같은 표준화된 분석법이 개발되었습니다4. 최근에, 표현형 프로파일링은 예를 들어, 소분자의 특성 분석 또는 환자 유래 섬유아세포만을 기반으로 하는 PD 아형의 정확한 예측에 적용되고 있다 5,6. 이러한 발전에도 불구하고 표현형 프로파일링은 LRRK2 G2019S와 같은 PD 연결 돌연변이를 발현하는 인간 유도만능줄기세포(iPSC) 유래 mDA 뉴런과 같은 유사분열 후 분화 세포에 거의 적용되지 않았습니다. iPSC 유래 모델의 중요한 과제로는 분화 배치 또는 유전자형에 걸쳐 미묘하거나 가변적인 병리학적 특징이 존재한다는 점과 분리된 PD 표현형이 질병의 전체 복잡성을 포착하지 못한다는 사실이 있습니다. 또한, iPSC 신경 세포 모델은 생리학적으로 관련이 있지만, 기술적 복잡성에 대한 우려로 인해 PD 약물 발견 프로세스에서는 거의 사용되지 않습니다 7,8.
우리는 이전에 유전적 및 화학적 화합물 유도 표현형 변화에 민감한 인간 mDA 뉴런에서 여러 PD 관련 병태생리학적 표현형을 측정하기 위한 강력한 방법론을 개발했습니다9. 이 기사에서는 mDA 뉴런에서 표현형 프로파일을 생성하기 위해 이 방법론을 더욱 최적화된 버전으로 만드는 방법에 대해 자세히 설명합니다(그림 1). 이 프로토콜은 고품질 mDA 뉴런의 사용 및 기술적 재현성과 같은 앞서 설명한 표현형 프로파일링 접근 방식에 비해 몇 가지 장점이 있습니다. 처음으로, 이 프로토콜은 확장성이 뛰어난 방식으로 화학적 섭동 후 생리학적으로 관련된 유사분열 후 mDA 뉴런에서 표현형 프로파일링을 가능하게 합니다. 완전히 분화되고 동결 보존된 mDA 뉴런은 상용화되어 배치 간 분화 변동성을 크게 줄여줍니다. 둘째, 잘 정의된 실험 설계(예: 배양 기간 또는 에지 웰 방지), 자동화된 액체 처리 및 자동화된 현미경을 사용하여 기술적 변동성을 더욱 줄일 수 있습니다. 또한 비지도 클러스터링 또는 지도 분류 접근 방식을 사용한 표현형 프로파일 분석의 초기 단계가 여기에 요약되어 표현형 프로파일링 데이터를 분석할 수 있는 방법을 나타냅니다. 이 프로토콜은 유전적 또는 화학적 섭동에 의해 유도된 mDA 뉴런의 표현형 변화에 관심이 있는 연구자, 특히 스크리닝 캠페인 중 또는 독성 효과를 결정하기 위해 더 적은 수의 화합물의 효과를 연구해야 하는 경우와 같이 확장성이 뛰어난 연구 설정이 필요한 경우에 사용됩니다. 요약하면, 인간 뉴런의 표현형 프로파일링의 적용은 복잡한 질병 관련 표현형을 연구하고 약물 후보의 세포 효과를 특성화하는 데 유용한 기술일 것으로 예상됩니다.
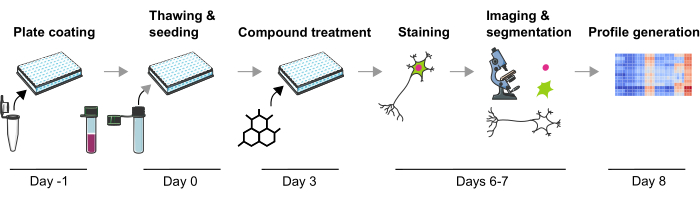
그림 1: 인간 iPSC 유래 mDA 뉴런에서 이미지 기반 표현형 프로파일을 생성하기 위한 실험 프로토콜의 개략도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
표현형 프로파일링(Phenotypic profiling)은 형광 염색, 현미경 검사, 이미지 분석 등을 적용하여 세포에서 많은 수의 표현형을 측정하는 기술이다3. 표현형 프로파일을 얻고 세포주 또는 기타 실험 조건에서 비교하여 단일 판독값을 사용할 때 눈에 띄지 않을 수 있는 세포 생물학의 복잡한 변화를 이해할 수 있습니다. 여기서는 PD 세포 생물학17,18,19<sup class="xr…
Disclosures
The authors have nothing to disclose.
Acknowledgements
저자는 제시된 프로토콜의 설계로 이어지는 귀중한 도움과 토론에 대해 Ksilink의 모든 동료에게 감사의 뜻을 전합니다.
Materials
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse – Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit – Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
References
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson’s disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson’s disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson’s Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson’s Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson’s disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson’s disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson’s disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson’s disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).

