Espressione di transgeni in cellule in coltura utilizzando vettori virali adeno-associati ricombinanti non purificati
Summary
Il virus adeno-associato ricombinante (rAAV) è ampiamente utilizzato per la somministrazione genica clinica e preclinica. Un uso sottovalutato per i rAAV è la robusta trasduzione delle cellule in coltura senza la necessità di purificazione. Per i ricercatori che non conoscono l’rAAV, forniamo un protocollo per il clonaggio di cassette transgeniche, la produzione di vettori grezzi e la trasduzione di colture cellulari.
Abstract
I vettori virali adeno-associati ricombinanti (rAAV) possono raggiungere un’espressione transgenica potente e duratura senza integrazione in un’ampia gamma di tipi di tessuto, rendendoli una scelta popolare per la somministrazione genica in modelli animali e in contesti clinici. Oltre alle applicazioni terapeutiche, gli rAAV sono un utile strumento di laboratorio per la somministrazione di transgeni su misura per le esigenze sperimentali e gli obiettivi scientifici del ricercatore nelle cellule in coltura. Alcuni esempi includono geni reporter esogeni, cassette di sovraespressione, interferenza dell’RNA e strumenti basati su CRISPR, compresi quelli per gli screening dell’intero genoma. Le trasduzioni di rAAV sono meno dannose per le cellule rispetto all’elettroporazione o alla trasfezione chimica e non richiedono attrezzature speciali o reagenti costosi per essere prodotte. I lisati grezzi o i terreni condizionati contenenti rAAV possono essere aggiunti direttamente alle cellule in coltura senza ulteriore purificazione per trasdurre molti tipi di cellule, una caratteristica sottovalutata degli rAAV. Qui, forniamo protocolli per il clonaggio di cassette transgeniche di base e dimostriamo come produrre e applicare preparazioni di rAAV grezze alle cellule in coltura. Come prova di principio, dimostriamo la trasduzione di tre tipi di cellule che non sono ancora state riportate nelle applicazioni rAAV: cellule placentari, mioblasti e organoidi dell’intestino tenue. Discutiamo gli usi appropriati per le preparazioni di rAAV grezze, i limiti dei rAAV per la consegna genica e le considerazioni per la scelta del capside. Questo protocollo delinea un metodo semplice, economico ed efficace per i ricercatori per ottenere la consegna produttiva del DNA in coltura cellulare utilizzando rAAV senza la necessità di laboriose fasi di titolazione e purificazione.
Introduction
Chiarire le basi molecolari delle funzioni cellulari spesso richiede l’espressione di DNA transgenico in coltura cellulare. Per essere espressi, i transgeni devono penetrare attraverso la membrana selettiva di una cellula e raggiungere il nucleo 1,2. Pertanto, la capacità di aggirare efficacemente le barriere fisiche della cellula e manipolare i suoi processi centrali è una necessità per applicare la transgenesi alla scoperta di nuovi fenomeni biologici. Un approccio sfrutta la capacità intrinseca dei virus di veicolare ed esprimere DNA estraneo 3,4.
Il virus adeno-associato (AAV) è uno dei più piccoli virus dei mammiferi: il suo genoma a DNA a singolo filamento di 4,7 kilobasi (kb) contiene due geni, rep (per la replicasi) e cap (per il capside), impacchettati all’interno di un capside icosaedrico di 60 mer che misura 25 nm. I geni rep/cap hanno più promotori, frame di lettura e prodotti di splicing che codificano per almeno nove proteine uniche necessarie per la replicazione, la produzione e il confezionamento virale 5,6. Inoltre, entrambe le estremità del genoma contengono strutture secondarie chiamate ripetizioni terminali invertite (ITR) che sono necessarie per la replicazione del DNA, l’impacchettamento del genoma e l’elaborazione a valle durante la trasduzione 7,8,9,10. Gli ITR sono gli unici elementi del DNA necessari per l’impacchettamento del genoma nel capside, e quindi l’AAV può essere clonato per scopi di trasporto del transgene sostituendo i geni rep/cap virali con elementi regolatori e/o geni di interesse6 scelti dal ricercatore. L’AAV ricombinante risultante (rAAV), con un genoma vettoriale ingegnerizzato (VG), è ampiamente utilizzato in clinica per la terapia genica umana e ha accumulato successi11. Un uso sottovalutato del vettore è in laboratorio; Gli rAAV possono raggiungere in modo efficiente l’espressione del transgene nelle cellule in coltura per soddisfare le esigenze sperimentali di un ricercatore12.
Il metodo più comune per la produzione di rAAV è la trasfezione a triplo plasmide in cellule HEK293 o 293T (Figura 1). Il primo plasmide, comunemente chiamato plasmide cis, contiene il transgene desiderato affiancato da ITR (pAAV). A seconda dell’applicazione, sono disponibili per l’acquisto plasmidi cis con elementi comuni, come promotori forti o strumenti basati su CRISPR. Il secondo è il plasmide pRep/Cap che contiene i geni wild-type AAV rep e cap forniti in-trans, cioè su un plasmide separato, non contenente ITR che esprime elementi regolatori e strutturali che poi interagiscono con il plasmide cis, ed è quindi chiamato plasmide trans. Oltre a racchiudere fisicamente il VG, il capside influenza il tropismo cellulare12,13. Fornendo il gene cap specifico per il sierotipo in-trans, i ricercatori sono facilmente in grado di massimizzare l’efficienza di trasduzione scegliendo un sierotipo del capside ottimizzato per la loro cellula bersaglio. Infine, come Dependoparvovirus, l’AAV richiede che un virus helper attivi l’espressione rep/cap dai suoi promotori virali, ottenuta dai geni helper adenovirali, forniti su un terzo plasmide come pAdΔF614,15. Dopo 72 ore di trasfezione a triplo plasmide, il vettore può essere rilasciato dalle cellule produttrici nel terreno di coltura mediante ripetuti cicli di congelamento/disgelo. L’intero contenuto della piastra viene quindi raccolto e i detriti cellulari di grandi dimensioni vengono rimossi mediante centrifugazione; il surnatante del terreno risultante è una preparazione grezza di rAAV pronta per le trasduzioni a valle.
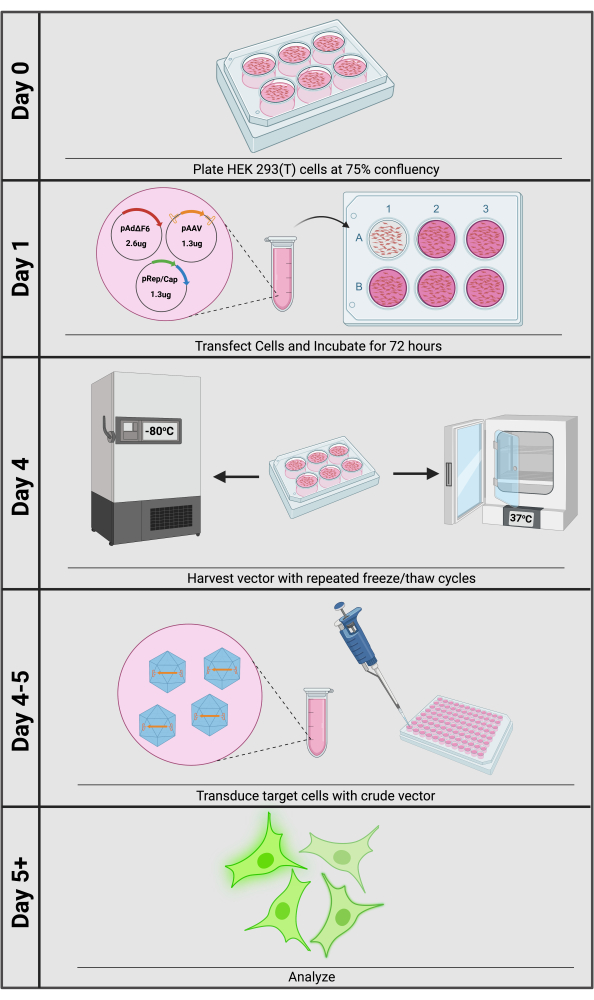
Figura 1: Panoramica della produzione di vettori rAAV grezzi. La produzione e la trasduzione di rAAV grezzi possono essere effettuate entro 5 giorni. Fare clic qui per visualizzare una versione più grande di questa figura.
rAAV può essere più favorevole per la somministrazione di transgeni rispetto ad altri metodi di trasfezione, che sono comunemente associati a tossicità cellulare, bassa efficienza e reagenti e attrezzature costosi, come per l’elettroporazione o la trasfezione a base chimica/lipidica16,17. rAAV aggira questi ostacoli e spesso fornisce una potente espressione transgenica con una tossicità minima e un tempo di intervento minimo. È importante sottolineare che la produzione di rAAV e la sua applicazione in coltura cellulare sono semplici e raramente richiedono la purificazione del vettore dal terreno di coltura (Figura 1). Inoltre, rAAV non integra il suo VG nel genoma dell’ospite, a differenza del rilascio del transgene lentivirale, e quindi riduce il rischio di mutagenesi inserzionale18. Nonostante i potenziali benefici dell’utilizzo di rAAV per la somministrazione di transgeni, è necessario considerare i limiti. È importante sottolineare che la dimensione del transgene, compresi gli ITR, non dovrebbe superare i 4,9 kb a causa dei vincoli fisici del capside, limitando così la capacità di un ricercatore di fornire efficacemente elementi regolatori e transgeni di grandi dimensioni. Inoltre, poiché rAAV è un virus non integrante, la trasduzione provoca un’espressione transgenica transitoria nelle cellule in divisione e potrebbe non essere pratica per un’espressione stabile. Tuttavia, i metodi che utilizzano il doppio Cas9 somministrato da rAAV e i modelli di riparazione diretta all’omologia (HDR) possono essere utilizzati per inserire stabilmente sequenze in loci genomici specifici, se un ricercatore lo desidera19.
Protocol
Representative Results
Discussion
Clonazione
Il protocollo di clonazione non è limitato al plasmide pAAV.CMV.Luc.IRES.EGFP.SV40 utilizzato in precedenza e può essere facilmente modificato in base alle esigenze sperimentali del ricercatore. Molti plasmidi contenenti ITR sono prontamente disponibili online per l’acquisto. Ad esempio, sono disponibili plasmidi contenenti sia Cas9 che un sito di clonaggio di sgRNA, ma richiedono alcuni passaggi aggiuntivi come la ricottura degli oligonucleotidi e il trattamento con PNK30. Inoltre, si possono trovare plasmidi contenenti un sito di clonazione multipla (MCS) con solo ITR e senza elementi regolatori interni31. Se devono essere utilizzati plasmidi diversi, gli enzimi di restrizione (RE) utilizzati per la digestione sono in genere gli unici elementi che potrebbero dover essere modificati in questo protocollo. Tuttavia, un limite dell’rAAV è la sua limitata capacità di carico. A causa delle limitazioni fisiche del capside, il genoma del vettore non dovrebbe superare i 4,9 kb, compresi gli ITR.
Quando si isola il plasmide dai batteri, è fondamentale utilizzare un kit midiprep o maxiprep a basso contenuto o privo di endotossine per mitigare i danni alle cellule durante la trasfezione o la trasduzione del triplo plasmide. I plasmidi dei kit miniprep contengono spesso impurità più elevate, concentrazioni ridotte e meno DNA superavvolto, che possono influenzare la produzione a valle di rAAV e quindi non sono raccomandati.
È fondamentale comprendere la struttura e le proprietà degli ITR durante la clonazione. In primo luogo, è estremamente difficile utilizzare la PCR attraverso l’ITR. I progetti di clonazione che richiedono l’amplificazione PCR tramite ITR dovrebbero essere evitati e dovrebbero inoltre limitare l’uso della tecnica di clonazione dell’assemblaggio Gibson. Pertanto, il clonaggio enzimatico di restrizione è il metodo preferito per la clonazione in plasmidi contenenti ITR. Inoltre, alcuni primer per il sequenziamento Sanger potrebbero non essere compatibili se la regione sequenziata contiene l’ITR. Invece, si consiglia di utilizzare primer che si allontanano dagli ITR e si inseriscono nel corpo del genoma del vettore per ottenere risultati di sequenziamento più precisi. In secondo luogo, gli ITR sono soggetti a delezioni, riarrangiamenti e mutazioni quando vengono trasformati in batteri per l’amplificazione plasmidica32,33. Per mitigare questi eventi, si consiglia di utilizzare ceppi batterici competenti carenti di ricombinazione, come Stbl3, e di incubarli a 30 °C per rallentare le divisioni cellulari. Infine, è stato osservato che le colonie più piccole possono corrispondere a cloni senza riarrangiamenti o delezioni, poiché quelle senza ITR possono conferire un vantaggio di crescita ed essere più grandi. Pertanto, si consiglia di scegliere colonie di piccole dimensioni.
Produzione vettoriale
Il successo della produzione del vettore rAAV può essere influenzato da più elementi. Un fattore critico è la salute delle cellule HEK293 o 293T utilizzate per la trasfezione. Generalmente, un basso numero di passaggi è l’ideale, poiché le cellule altamente passate possono presentare variazioni genotipiche e fenotipiche che possono ridurre i titoli di rAAV. Inoltre, la densità delle cellule seminate dovrebbe essere del 75%-90% di confluenza per una produzione efficace. Le cellule sparse generano basse rese vettoriali perché ci sono meno cellule disponibili per produrre vettori, mentre le cellule troppo cresciute non saranno trasfettate in modo efficiente.
Le variazioni tra i lotti di reagenti, le scorte di cellule e la variabilità generale da laboratorio a laboratorio contribuiscono alle differenze nell’efficienza di trasfezione e nel titolo di produzione. Un fattore ottimizzabile che può portare a miglioramenti del titolo è il rapporto plasmide:PEI nelle reazioni di trasfezione. È fondamentale utilizzare PEI MAX fresco (<1 mese). Si raccomanda di utilizzare un rapporto plasmide:PEI di 1:1 come punto di partenza e, se l'efficienza di trasfezione o trasduzione appare scarsa, testare diversi rapporti. L'ottimizzazione del titolo è più semplice se si utilizza un transgene con una lettura visiva, come il transgene reporter CMV.Luc.IRES.EGFP utilizzato in questo documento come materiale di partenza per la clonazione. Per eseguire l'ottimizzazione, seguire la fase 3 del protocollo utilizzando una piastra a 12 pozzetti e riducendo di due le masse del plasmide e i volumi dei reagenti (la massa finale del plasmide è di 2,6 μg). Regolare di conseguenza il volume PEI in modo che corrisponda a rapporti compresi tra 1:0,75 e 1:3, con incrementi crescenti di 0,25 (Figura 6). Diluire ogni reazione con 950 μL di terreno SF dopo 15 minuti. Per comodità, è possibile creare una miscela master contenente i plasmidi tripli e pipettare singolarmente in provette da 1,5 mL prima di aggiungere PEI (vedere il file supplementare 2). Raccogli il vettore, trasduci le cellule di interesse e l’immagine. Il pozzetto con la più alta efficienza di trasduzione (proporzione di cellule GFP+) corrisponde al titolo più alto e al rapporto ottimale di PEI:DNA.
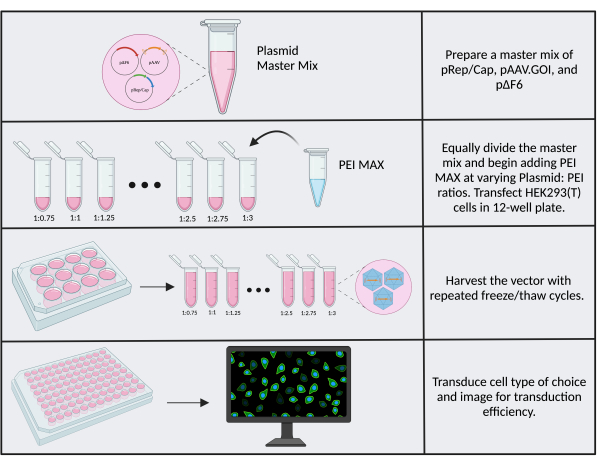
Figura 6: Flusso di lavoro di ottimizzazione PEI. Schema dei passaggi necessari per l’ottimizzazione PEI. Rapporti multipli di plasmidi: PEI vengono testati per determinare il rapporto ottimale. Fare clic qui per visualizzare una versione più grande di questa figura.
Considerazioni sul raccolto e sul titolo
La tecnica di congelamento/scongelamento utilizzata per raccogliere il vettore rAAV lisa efficacemente le cellule HEK293 in modo compatibile con l’uso diretto del lisato chiarificato per trasdurre le cellule in coltura. Alcuni sierotipi di rAAV, come AAV1, AAV8 e AAV9, vengono rilasciati dalle cellule durante la produzione del vettore e possono essere raccolti dal terreno cellulare coltivato senza cicli di congelamento/scongelamento34. Il metodo qui descritto produce in genere titoli dell’ordine di 1 x 1010 VG/mL quando si utilizzano capsidi AAV2 e 1 x10 11 VG/mL per AAV8. Sebbene titoli più elevati possano essere ottenuti mediante lisi detergente o altre sostanze chimiche, queste sono dannose per le cellule nell’uso a valle e richiedono che i rAAV siano ulteriormente purificati dal lisato. Il titolo più basso è un compromesso che un ricercatore dovrebbe considerare quando determina se i preparati grezzi sono appropriati per le proprie esigenze di ricerca, tuttavia, i titoli marginalmente più bassi prodotti dai metodi qui descritti possono trasdurre molto bene molti tipi di cellule (vedi risultati rappresentativi). Oltre all’efficienza di trasfezione e alla salute cellulare, i titoli dei vettori variano a seconda del capside utilizzato durante la produzione di rAAV e delle dimensioni e della sequenza del transgene all’interno del VG35.
Durante la raccolta di preparati vettoriali grezzi, può essere presente DNA plasmidico utilizzato durante la trasfezione a triplo plasmide e, sebbene raramente, provocare la trasfezione a valle durante la trasduzione. Inoltre, i VG non confezionati possono legarsi all’esterno dei capsidi e invocare una risposta immunitaria innata al DNA a singolo filamento nudo ed estraneo36,37. Pertanto, i tipi di cellule sensibili possono richiedere che le preparazioni vettoriali siano digerite e purificate con DNasi per rimuovere i VG e i plasmidi non confezionati.
Se si desidera calcolare il titolo di un preparato grezzo, è possibile eseguire la qPCR per quantificare il numero di VG confezionati all’interno di particelle resistenti alla DNasi (DRP). In breve, una piccola quantità di preparato grezzo viene digerita con DNasi per rimuovere il DNA plasmidico, contaminando gli acidi nucleici o il VG parzialmente confezionato. Il campione viene quindi sottoposto a qPCR e viene quantificato il VG protetto all’interno dei DRP, ottenendo un titolo con unità di genoma vettoriale per mL di preparato grezzo38. Non è consigliabile eseguire la titolazione vettoriale utilizzando saggi basati su ELISA che quantificano i titoli del capside. Rispetto al virus AAV wild-type, il rAAV soffre di una percentuale di capsidi vuoti e parzialmente impacchettati39. L’ELISA quantificherà tutti i capsidi indipendentemente dal loro contenuto genomico e sovrastimerà le unità trasducibili presenti in una preparazione, che richiede un VG impacchettato.
Considerazioni sulla trasduzione
Molti fattori influenzano le trasduzioni di rAAV e le opportune considerazioni dovrebbero essere fatte per ogni nuovo esperimento. A seconda del promotore che guida l’espressione del transgene, l’insorgenza dell’espressione può avvenire già 4 ore dopo la trasduzione (hpt) e il picco di espressione è tipicamente raggiunto entro 48 hpt. È importante tenere presente il periodo di tempo che intercorre tra la semina iniziale delle cellule e l’endpoint sperimentale. Questo per stimare la confluenza iniziale delle cellule e garantire che non crescano eccessivamente entro la fine dell’esperimento. Se le cellule diventano troppo confluenti, il comportamento cellulare può essere alterato a causa di una risposta allo stress e può confondere i risultati sperimentali. Alcuni tipi di cellule, come U2-OS, possono tollerare abbastanza bene la crescita eccessiva/inibizione da contatto. Inoltre, possono resistere a lunghi periodi (48 h+) in terreno condizionato privo di siero, il prodotto di questo protocollo di produzione. Tuttavia, i tipi di cellule sensibili possono richiedere l’aggiunta di siero o la diluizione del preparato grezzo con uno speciale terreno di crescita per mantenere la salute durante la trasduzione. Un’efficienza di trasduzione leggermente ridotta dall’uso di terreni contenenti siero è un potenziale compromesso per la salute delle cellule e dovrebbe essere presa in considerazione dal ricercatore.
Tipicamente, per celle che si dividono rapidamente, una confluenza iniziale di circa il 50% è ottimale per le applicazioni che saranno terminate a 48 hpt. Tuttavia, la confluenza può essere regolata di conseguenza in base alle esigenze dell’esperimento. Non è consigliabile trasdurre linee cellulari immortalizzate di tipo monostrato con una confluenza superiore al 75% a causa della diminuzione dell’efficienza di trasduzione. La maggior parte dei tipi di cellule in coltura viene trasdotta con successo e sana dopo l’incubazione notturna con preparati rAAV grezzi, seguita da un passaggio a terreni freschi contenenti siero al mattino.
Il sierotipo del capside è un fattore importante da considerare quando si produce rAAV per trasdurre una cellula bersaglio, poiché il capside è il determinante primario del tropismo cellulare e della successiva espressione del transgene13. AAV2 è un sierotipo ampiamente utilizzato grazie alla sua capacità di trasdurre efficacemente molti tipi di cellule in coltura12. Questa proprietà di AAV2 può essere attribuita ai proteoglicani dell’eparina solfato (HSPGs) che fungono da fattore di attacco primario per AAV2 e agli alti livelli di HSPGs sulle cellule in coltura dall’adattamento alla crescita in un piatto40. Altri capsidi, come AAV9, sono meno efficaci nel trasdurre i tipi di cellule larghe e possono essere spiegati dai loro fattori di attaccamento di dipendenza che non sono espressi in questo setting41. Pertanto, raccomandiamo AAV2 come capside di prima scelta nelle cellule in coltura se una cellula bersaglio desiderata non è stata precedentemente testata con rAAV in letteratura.
Si noti che una delle principali limitazioni dei preparati vettoriali grezzi è che sono inappropriati per la trasduzione di modelli animali. Gli studi in vivo richiedono che i preparati siano purificati e sottoposti a valutazione della qualità.
Considerazioni sull’espressione del transgene e sulla potenziale integrazione
Gli rAAV non determinano in modo affidabile l’espressione permanente del transgene. Nel corso del tempo, i VG possono essere silenziati e l’espressione transgenica può essere interrotta dopo diversi passaggi42. Inoltre, la maggior parte dei VG rimane episomiale e i rAAV non contengono le proteine Rep virali che medierebbero la frequente integrazione nel genoma dell’ospite come in un’infezione lisogenica virale wild-type o promuoverebbero la replicazione dei VG43. Di conseguenza, gli episomi nelle cellule trasdotte alla fine saranno diluiti tra le cellule figlie attraverso le divisioni.
L’integrazione a livello basale è una possibilità per tutto il materiale di DNA transgenico consegnato. Tuttavia, i VG contenenti ITR sono inclini all’integrazione a una frequenza più elevata44. Pertanto, l’espressione permanente di un transgene può essere osservata in un piccolo sottogruppo di cellule. Gli utenti dovrebbero considerare questa possibilità soprattutto quando utilizzano rAAV per fornire enzimi che tagliano il DNA, come Cas9, poiché le rotture a doppio filamento possono comportare una frequenza ancora maggiore di integrazione ed espressione permanente45. Sebbene ciò renda rAAV un buon candidato per la fornitura di modelli di riparazione diretti all’omologia per il tagging endogeno o l’aggiunta genica, la possibilità di inserimento di Cas9 dovrebbe essere considerata19,46.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ringraziamo Robert Tjian e Xavier Darzacq per il loro supporto e l’uso delle attrezzature di laboratorio. Ringraziamo Mark Kay per il suo dono dei plasmidi rep/cap KP1 e LK03, e Luk Vandenberghe per il plasmide AAV4 rep/cap. Il finanziamento è stato fornito dall’Howard Hughes Medical Institute (34430, R. T.) e dal California Institute for Regenerative Medicine Training Program EDUC4-12790. N.W. riconosce il finanziamento del Berkeley Stem Cell Center tramite una borsa di studio post-dottorato Siebel e della Fondazione tedesca per la ricerca (DFG) tramite una borsa di studio Walter Benjamin.
Materials
| Snapgene DNA viewing sofrware | Snapgene | ||
| pAAV.CMV.Luc.IRES.EGFP.SV40 | AddGene | 105533 | |
| pAAV2/2 (Rep/Cap) | AddGene | 104963 | |
| pAdΔF6 | AddGene | 112867 | |
| LB Agar Carbenicillin | Sigma-Aldrich | L0418 | |
| Boekel Scientific Economy Digital Incubator | Boekel Scientific | 133000 | |
| LB medium, powder | MP Biomedicals | 113002042 | |
| Carbencillin (Disodium) | GoldBio | C-103-5 | |
| New Brunswick I26 Shaker | Eppendorf | M1324-0000 | |
| 50 mL Centrifuge Tubes | Corning | 430828 | |
| Centrifuge 5810 R | Eppendorf | 22627040 | |
| QIAGEN Plasmid Plus Midi Kit | Qiagen | 12945 | |
| PCR PULL-APART 8-TUBE STRIPS | USA Scientific | 1402-3900 | |
| Mastercycler nexus | Eppendorf | 6333000022 | |
| dNTP | Thermo Fisher Scientific | 18427013 | |
| 5x Phusion Buffer | NEB | B0518S | Provided with purchase of Phusion Polymerase |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530S | |
| Gel Loading Dye, Orange (6X) | NEB | B7022S | |
| DNA Clean & Concentrator-100 | Zymo | D4029 | |
| Zymoclean Gel DNA Recovery Kit | Zymo | D4001 | |
| NotI-HF | NEB | R3189S | |
| EcoRI-HF | NEB | R3101S | |
| CutSmart Buffer (10x) | NEB | B6004S | Provided with purchase of restriction enzyme |
| UltraPure Agarose | Thermo Fisher Scientific | 16500100 | |
| Ethidium Bromide | Sigma-Aldrich | E1510 | |
| T4 DNA Ligase | NEB | M0202S | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | Provided with purchase of T4 Ligase |
| Eppendorf Safe-Lock Tubes (1.5mL) | Eppendorf | 22363204 | |
| Precision Microprocessor Water Bath | Thermo Scientific | 51221046 | |
| Sterile Plastic Culture Tubes | Fisher Scientific | 149566B | |
| 2.0 mL Microcentrifuge Tube | Thomas Scientific | 1149Y01 | |
| ZR Plasmid Miniprep – Classic | Zymo | D4015 | |
| Xma1 | NEB | R0180S | |
| Sma1 | NEB | R0141S | |
| HEK 293T cells | ATCC | CRL-3216 | |
| Falcon 6-well | Corning | 353046 | |
| Gibco DMEM, high glucose, pyruvate | Thermo-Fisher | 11995065 | |
| Sanyo MCO-18AIC(UV) CO2 Incubator | Marshall Scientific | MCO-18AIC | |
| PEI MAX (Polyethylenimine Hydrochloride) | Polysciences | 24765-100 | |
| Mixer Vortex Genie 2 | Electron Microscopy Sciences | 102091-234 | |
| Sanyo Ultra Low Freezer | Sanyo | 14656-15267-16219 | |
| INCU-Line IL 10 with transparent window | VWR | 390-0384 | |
| Eppendorf Microcentrifuges | Eppendorf | 05-400-005 | |
| Falcon 96-well | Corning | 353072 | |
| C57BL/6J mice | JAX | strain #000664 | |
| organoid growth medium | STEMCELL Technologies | 6005 | |
| L Wnt-3A cells | ATCC | CRL-2647 | |
| nicotinamide | Sigma | N0636-100G | |
| ROCK inhibitor | STEMCELL Technologies | 72302 | |
| CHIR99021 | STEMCELL Technologies | 72052 | |
| Corning Matrigel Growth Factor Reduced (GFR) Basement Membrane | Fisher | 356231 | |
| 24-well plate | Fisher | 08-772-1 | |
| D-PBS | Thermo Fisher Scientific | 14-190-250 | |
| TrypLE Express | Fisher | 12604013 | |
| DMEM/F-12 with 15 mM HEPES | STEMCELL Technologies | 36254 | |
| polybrene | Millipore Sigma | TR-1003-G | |
| 48-well plates | Fisher | 08-772-3D | |
| Thermo Scientific Nunc Lab-Tek Chambered Coverglass | Fisher | 12-565-470 | |
| BeWo cells | ATCC | CCL-98 | |
| F-12K Medium | ATCC | 30-2004 | |
| Hepa1-6 | ATCC | CRL-1830 | |
| Huh7 | UC Berkeley BSD Cell Culture Facility | HUH-7 | |
| C2C12 | ATCC | CRL-1772 | |
| HSkMC | ATCC | PCS-950-010 | |
| Skeletal Muscle Cell Growth Medium | Sigma | C-23060 | |
| Skeletal Muscle Differentiation Medium | Sigma | C-23061 | |
| Invitrogen EVOS Digital Color Fluorescence Microscope | Fisher Scientific | 12-563-340 | |
| Perkin Elmer Opera Phenix | Perkin Elmer | HH14001000 | |
| PhenoPlate 96-well | Perkin Elmer | 6055302 | |
| DMEM, high glucose, no glutamine, no phenol red | Thermo-Fisher | 31053028 | |
| GlutaMAX Supplement | Thermo-Fisher | 35050079 | |
| Sodium Pyruvate | Thermo-Fisher | 11360070 | |
| pAAV2/5 (Rep/Cap) | Addgene | 104964 | |
| pAAV2/8 (Rep/Cap) | Addgene | 112864 | |
| pAAV-DJ-N589X (Rep/Cap) | Addgene | 130878 | |
| pAAV2/9n (Rep/Cap) | Addgene | 112865 | |
| pAnc80L65AAP | Addgene | 92307 | |
| KP1 (rep/cap) | gifted by Professor Mark Kay (Stanford University) | ||
| LK03 (rep/cap) | gifted by Professor Mark Kay (Stanford University) | ||
| pAAV4 (rep/cap) | gifted by Professor Luk Vandenberghe (Harvard Medical School) | ||
| pAAV.Cas9.sgRNA | Addgene | 61591 | |
| pAAV.MCS | Addgene | 46954 | |
| gBlock (synthetic DNA fragement) | IDT |
References
- Pillay, S., et al. Corrigendum: An essential receptor for adeno-associated virus infection. Nature. 539 (7629), 456 (2016).
- Nicolson, S. C., Samulski, R. J. Recombinant adeno-associated virus utilizes host cell nuclear import machinery to enter the nucleus. Journal of Virology. 88 (8), 4132-4144 (2014).
- Lundstrom, K. Viral Vectors in Gene Therapy. Diseases. 6 (2), 42 (2018).
- Daya, S., Berns, K. I. Gene therapy using adeno-associated virus vectors. Clinical Microbiology Reviews. 21 (4), 583-593 (2008).
- Ling, C., et al. The Adeno-Associated Virus Genome Packaging Puzzle. Journal of Molecular and Genetic Medicine. 9 (3), 175 (2015).
- Maurer, A. C., Weitzman, M. D. Adeno-Associated Virus Genome Interactions Important for Vector Production and Transduction. Human Gene Therapy. 31 (9-10), 499-511 (2020).
- Srivastava, A. Replication of the adeno-associated virus DNA termini in vitro. Intervirology. 27 (3), 138-147 (1987).
- Wang, X. S., Ponnazhagan, S., Srivastava, A. Rescue and replication of adeno-associated virus type 2 as well as vector DNA sequences from recombinant plasmids containing deletions in the viral inverted terminal repeats: selective encapsidation of viral genomes in progeny virions. Journal of Virology. 70 (3), 1668-1677 (1996).
- Earley, L. F., et al. Adeno-Associated Virus Serotype-Specific Inverted Terminal Repeat Sequence Role in Vector Transgene Expression. Human Gene Therapy. 31 (3-4), 151-162 (2020).
- Yang, J., et al. Concatamerization of adeno-associated virus circular genomes occurs through intermolecular recombination. Journal of Virology. 73 (11), 9468-9477 (1999).
- Au, H. K. E., Isalan, M., Mielcarek, M. Gene Therapy Advances: A Meta-Analysis of AAV Usage in Clinical Settings. Frontiers in Medicine. 8, 809118 (2021).
- Ellis, B. L., et al. A survey of ex vivo/in vitro transduction efficiency of mammalian primary cells and cell lines with Nine natural adeno-associated virus (AAV1-9) and one engineered adeno-associated virus serotype. Virology Journal. 10, 74 (2013).
- Zincarelli, C., Soltys, S., Rengo, G., Rabinowitz, J. E. Analysis of AAV serotypes 1-9 mediated gene expression and tropism in mice after systemic injection. Molecular Therapy. 16 (6), 1073-1080 (2008).
- Atchison, R. W., Casto, B. C., Hammon, W. M. Adenovirus-Associated Defective Virus Particles. Science. 149 (3685), 754-756 (1965).
- Meier, A. F., Fraefel, C., Seyffert, M. The Interplay between Adeno-Associated Virus and its Helper Viruses. Viruses. 12 (6), 662 (2020).
- Djurovic, S., Iversen, N., Jeansson, S., Hoover, F., Christensen, G. Comparison of nonviral transfection and adeno-associated viral transduction on cardiomyocytes. Molecular Biotechnology. 28 (1), 21-32 (2004).
- Batista Napotnik, T., Polajzer, T., Miklavcic, D. Cell death due to electroporation – A review. Bioelectrochemistry. 141, 107871 (2021).
- McCarty, D. M., Young, S. M., Samulski, R. J. Integration of adeno-associated virus (AAV) and recombinant AAV vectors. Annual Review of Genetics. 38, 819-845 (2004).
- Yang, Y., et al. A dual AAV system enables the Cas9-mediated correction of a metabolic liver disease in newborn mice. Nature Biotechnology. 34 (3), 334-338 (2016).
- Stacey, G. N. Cell culture contamination. Methods in Molecular Biology. 731, 79-91 (2011).
- Parks, W. P., Melnick, J. L., Rongey, R., Mayor, H. D. Physical assay and growth cycle studies of a defective adeno-satellite virus. Journal of Virology. 1 (1), 171-180 (1967).
- Bantel-Schaal, U., zur Hausen, H. Characterization of the DNA of a defective human parvovirus isolated from a genital site. Virology. 134 (1), 52-63 (1984).
- Gao, G. P., et al. Novel adeno-associated viruses from rhesus monkeys as vectors for human gene therapy. Proceedings of the National Academy of Science United States of America. 99 (18), 11854-11859 (2002).
- Gao, G., et al. Clades of Adeno-associated viruses are widely disseminated in human tissues. Journal of Virology. 78 (12), 6381-6388 (2004).
- Zinn, E., et al. In Silico Reconstruction of the Viral Evolutionary Lineage Yields a Potent Gene Therapy Vector. Cell Reports. 12 (6), 1056-1068 (2015).
- Grimm, D., et al. In vitro and in vivo gene therapy vector evolution via multispecies interbreeding and retargeting of adeno-associated viruses. Journal of Virology. 82 (12), 5887-5911 (2008).
- Lisowski, L., et al. Selection and evaluation of clinically relevant AAV variants in a xenograft liver model. Nature. 506 (7488), 382-386 (2014).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. JCI Insight. 4 (22), e131610 (2019).
- Colon-Thillet, R., Jerome, K. R., Stone, D. Optimization of AAV vectors to target persistent viral reservoirs. Virology Journal. 18 (1), 85 (2021).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Britton, S., Coates, J., Jackson, S. P. A new method for high-resolution imaging of Ku foci to decipher mechanisms of DNA double-strand break repair. The Journal of Cell Biology. 202 (3), 579-595 (2013).
- Bi, X., Liu, L. F. DNA rearrangement mediated by inverted repeats. Proceedings of the National Academy of Science United States of America. 93 (2), 819-823 (1996).
- Samulski, R. J., Berns, K. I., Tan, M., Muzyczka, N. Cloning of adeno-associated virus into pBR322: rescue of intact virus from the recombinant plasmid in human cells. Proceedings of the National Academy of Science United States of America. 79 (6), 2077-2081 (1982).
- Vandenberghe, L. H., et al. Efficient serotype-dependent release of functional vector into the culture medium during adeno-associated virus manufacturing. Human Gene Therapy. 21 (10), 1251-1257 (2010).
- Sommer, J. M., et al. Quantification of adeno-associated virus particles and empty capsids by optical density measurement. Molecular Therapy. 7 (1), 122-128 (2003).
- Zhu, J., Huang, X., Yang, Y. The TLR9-MyD88 pathway is critical for adaptive immune responses to adeno-associated virus gene therapy vectors in mice. The Journal of Clinical Investigation. 119 (8), 2388-2398 (2009).
- Wagner, H., Bauer, S. All is not Toll: new pathways in DNA recognition. The Journal of Experimental Medicine. 203 (2), 265-268 (2006).
- Sanmiguel, J., Gao, G., Vandenberghe, L. H. Quantitative and Digital Droplet-Based AAV Genome Titration. Methods in Molecular Biology. 1950, 51-83 (2019).
- Grimm, D., et al. Titration of AAV-2 particles via a novel capsid ELISA: packaging of genomes can limit production of recombinant AAV-2. Gene Therapy. 6 (7), 1322-1330 (1999).
- Summerford, C., Samulski, R. J. Membrane-associated heparan sulfate proteoglycan is a receptor for adeno-associated virus type 2 virions. Journal of Virology. 72 (2), 1438-1445 (1998).
- Bell, C. L., et al. The AAV9 receptor and its modification to improve in vivo lung gene transfer in mice. The Journal of Clinical Investigation. 121 (6), 2427-2435 (2011).
- McCown, T. J., Xiao, X., Li, J., Breese, G. R., Samulski, R. J. Differential and persistent expression patterns of CNS gene transfer by an adeno-associated virus (AAV) vector. Brain Research. 713 (1-2), 99-107 (1996).
- Weitzman, M. D., Kyostio, S. R., Kotin, R. M., Owens, R. A. Adeno-associated virus (AAV) Rep proteins mediate complex formation between AAV DNA and its integration site in human DNA. Proceedings of the National Academy of Science United States of America. 91 (13), 5808-5812 (1994).
- Miller, D. G., Petek, L. M., Russell, D. W. Adeno-associated virus vectors integrate at chromosome breakage sites. Nature Genetics. 36 (7), 767-773 (2004).
- Hanlon, K. S., et al. High levels of AAV vector integration into CRISPR-induced DNA breaks. Nature Communications. 10 (1), 4439 (2019).
- Porteus, M. H., Cathomen, T., Weitzman, M. D., Baltimore, D. Efficient gene targeting mediated by adeno-associated virus and DNA double-strand breaks. Molecular and Cellular Biology. 23 (10), 3558-3565 (2003).

