Realistisk membranmodellering ved hjælp af komplekse lipidblandinger i simuleringsstudier
Summary
Membranlipiddiversitet i struktur og sammensætning er en vigtig bidragyder til cellulære processer og kan være en markør for sygdom. Molekylære dynamiksimuleringer giver os mulighed for at studere membraner og deres interaktioner med biomolekyler ved atomistisk opløsning. Her leverer vi en protokol til at bygge, køre og analysere komplekse membransystemer.
Abstract
Lipider er strukturelle byggesten i cellemembraner; Lipidarter varierer på tværs af celleorganeller og på tværs af organismer. Denne variation resulterer i forskellige mekaniske og strukturelle egenskaber i membranen, der direkte påvirker molekylerne og processerne, der forekommer ved denne grænseflade. Lipidsammensætning er dynamisk og kan tjene til at modulere cellesignaleringsprocesser. Computational tilgange bruges i stigende grad til at forudsige interaktioner mellem biomolekyler og give molekylær indsigt i eksperimentelle observerbare. Molekylær dynamik (MD) er en teknik baseret på statistisk mekanik, der forudsiger bevægelsen af atomer baseret på de kræfter, der virker på dem. MD-simuleringer kan bruges til at karakterisere interaktionen mellem biomolekyler. Her introducerer vi kort teknikken, skitserer praktiske trin for begyndere, der er interesseret i at simulere lipiddobbeltlag, demonstrerer protokollen med begyndervenlig software og diskuterer alternativer, udfordringer og vigtige overvejelser om processen. Vi understreger især relevansen af at bruge komplekse lipidblandinger til at modellere en cellemembran af interesse for at fange de passende hydrofobe og mekaniske miljøer i simulering. Vi diskuterer også nogle eksempler, hvor membransammensætning og egenskaber modulerer interaktionerne mellem dobbeltlag og andre biomolekyler.
Introduction
Lipider er vigtige bestanddele af membraner, som giver grænser for celler og muliggør intracellulær ruminddeling 1,2,3. Lipider er amfifile, med en polær hovedgruppe og to hydrofobe fedtsyrehaler; Disse samles selv i et dobbeltlag for at minimere kontakt mellem de hydrofobe kæder og vand 3,4. Forskellige kombinationer af hydrofile hovedgrupper og hydrofobe haler resulterer i forskellige klasser af lipider i biologiske membraner, såsom glycerophospholipider, sfingolipider og steroler (figur 1)1,5,6. Glycerophospholipider er primære byggesten i eukaryote cellemembraner sammensat af glycerophosphat, langkædede fedtsyrer og hovedgrupper med lav molekylvægt7. Lipidnomenklatur er baseret på forskelle i hovedgrupper; eksempler omfatter phosphatidylcholin (PC), phosphatidylethanolamin (PE), phosphatidylserin (PS), phosphatidylglycerol (PG), phosphatidyl-inositol (PI) eller umodificeret phosphatidsyre (PA)5,6. Hvad angår hydrofobe haler, varierer længden og graden af mætning sammen med rygradstrukturen. De mulige kombinationer er talrige, hvilket resulterer i tusindvis af lipidarter i pattedyrceller6. Ændringer i membranlipidsammensætningen fører til forskellige mekaniske og strukturelle membranegenskaber, der påvirker aktiviteten af både integrerede membranproteiner og perifere proteiner 2,6.
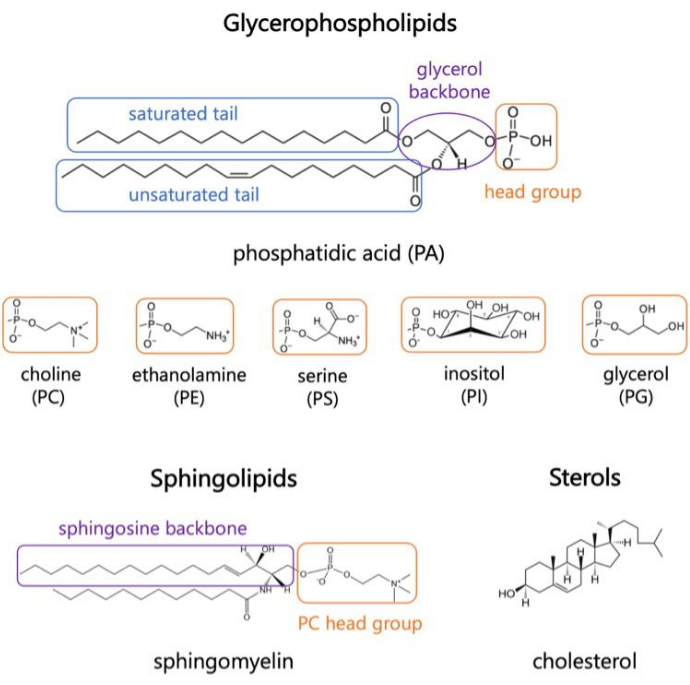
Figur 1. Repræsentative lipidstrukturer. Fedtsyrehaler vises i blå kasser, almindelige lipidhovedgrupper i orange og prøverygrad i lilla. Klik her for at se en større version af denne figur.
Lipider er aktive spillere i cellulære processer, proteinaktivering i signalkaskader og sund cellehomeostase 8,9. Ændret lipiddynamik er resultatet af infektion eller kan være markører for patogenese af sygdom 10,11,12,13,14,15. Som barrierer for cellen er undersøgelsen af membranlipider og deres rolle i gennemtrængning af små molekyler af relevans for lægemiddelafgivelsessystemer og membranforstyrrelsesmekanismer16,17. Kemisk mangfoldighed og forskellige forhold mellem lipidarter på tværs af organeller, væv og organismer giver anledning til kompleks membrandynamik2. Det er derfor vigtigt at bevare disse egenskaber i modelleringsstudier af lipiddobbeltlag, især når målet med en undersøgelse er at undersøge interaktioner mellem andre biomolekyler og membranen. De lipidarter, der skal overvejes i en model, afhænger af organismen og det cellulære rum af interesse. For eksempel er PG-lipider vigtige for elektronoverførsel i fotosyntetisk bateria18, mens phosphoryleret inositollipider (PIP’er) er vigtige aktører i plasmamembrandynamik (PM) og signalkaskader i pattedyrceller 19,20. Inde i cellen indeholder PM, endoplasmatisk retikulum (ER), Golgi og mitokondriemembraner unikke lipidmængder, der påvirker deres funktion. For eksempel er ER knudepunktet for lipidbiogenese og transporterer kolesterol ud til PM og Golgi; den indeholder en høj lipiddiversitet med en overflod af PC og PE, men lavt sterolindhold, hvilket fremmer membranfluiditet21,22,23,24. I modsætning hertil inkorporerer PM hundreder og endda tusinder af lipidarter afhængigt af organismen25, den indeholder høje niveauer af sphingolipider og kolesterol, der giver den en karakteristisk stivhed sammenlignet med andre membraner i cellen24. Brochureasymmetri bør overvejes for membraner som PM, som har en ydre folder rig på sphingomyelin, PC og kolesterol og en indre folder rig på PE, PI og PS, der er vigtige for signalering af kaskader24. Endelig beder lipiddiversitet også dannelsen af mikrodomæner, der adskiller sig i pakning og intern orden, kendt som lipidflåder24,26; Disse udviser lateral asymmetri, antages at spille vigtige roller i cellulær signalering26 og er svære at studere på grund af deres forbigående karakter.
Eksperimentelle teknikker såsom fluoroskopi, spektroskopi og modelmembransystemer som gigantiske unilamellære vesikler (GUV’er) er blevet brugt til at undersøge interaktioner mellem biomolekyler og membraner. Imidlertid er den komplekse og dynamiske karakter af de involverede komponenter vanskelig at fange med eksperimentelle metoder alene. For eksempel er der begrænsninger på billeddannelse af transmembrandomæner af proteiner, kompleksiteten af membraner, der anvendes i sådanne undersøgelser, og identifikation af mellemliggende eller forbigående tilstande under processen af interesse27,28,29. Siden fremkomsten af molekylær simulering af lipidmonolag og dobbeltlag i 1980’erne29 kan lipid-proteinsystemer og deres interaktioner nu kvantificeres på molekylært niveau. Simulering af molekylær dynamik (MD) er en almindelig beregningsteknik, der forudsiger partiklernes bevægelse baseret på deres intermolekylære kræfter. Et additivt interaktionspotentiale beskriver de bundne og ikke-bundne interaktioner mellem partikler i systemet30. Det sæt parametre, der bruges til at modellere disse interaktioner, kaldes simuleringskraftfeltet (FF). Disse parametre opnås fra ab initio-beregninger, semiempiriske og kvantemekaniske beregninger og optimeres til reproducerede data fra røntgen- og elektrondiffraktionseksperimenter, NMR, infrarød, Raman og neutronspektroskopi, blandt andre metoder31.
MD-simuleringer kan bruges til at studere systemer på forskellige niveauer af opløsning32,33,34. Systemer, der sigter mod at karakterisere specifikke biomolekylære interaktioner, hydrogenbindinger og andre detaljer med høj opløsning, studeres med all-atom (AA) simuleringer. I modsætning hertil klumper grovkornede (CG) simuleringer atomer i større funktionelle grupper for at reducere beregningsomkostningerne og undersøge dynamik i større skala33. Placeret imellem disse to er united-atom (UA) simuleringer, hvor hydrogenatomer kombineres med deres respektive tunge atomer for at fremskynde beregningen33,35. MD-simuleringer er et kraftfuldt værktøj til udforskning af dynamikken i lipidmembraner og deres interaktioner med andre molekyler og kan tjene til at tilvejebringe molekylære niveaumekanismer til processer af interesse ved membrangrænsefladen. Derudover kan MD-simuleringer tjene til at indsnævre eksperimentelle mål og forudsige makromolekylære egenskaber af et givet system baseret på mikroskopiske interaktioner.
Kort sagt, givet et sæt indledende koordinater, hastigheder og et sæt betingelser som konstant temperatur og tryk, beregnes positioner og hastigheder for hver partikel gennem numerisk integration af interaktionspotentialet og Newtons bevægelseslov. Dette gentages iterativt og genererer derved en simuleringsbane30. Disse beregninger udføres med en MD-motor; blandt flere open source-pakker er GROMACS36 en af de mest anvendte motorer og den, vi beskriver her. Det omfatter også værktøjer til analyse og konstruktion af indledende koordinater for systemer, der skal simuleres37. Andre MD-motorer omfatter NAMD38; CHARMM39 og AMBER40, som brugeren kan vælge efter eget skøn baseret på beregningsydelsen for et givet system. Det er afgørende at visualisere banerne under simuleringen samt til analyse og fortolkning af resultaterne. En række værktøjer er tilgængelige; her diskuterer vi visuel molekylær dynamik (VMD), der tilbyder en bred vifte af funktioner, herunder tredimensionel (3-D) visualisering med ekspansive tegnings- og farvelægningsmetoder, volumetrisk datavisualisering, opbygning, forberedelse og analyse af baner af MD-simuleringssystemer og banefilmfremstilling uden grænser for systemstørrelse, hvis hukommelsen er tilgængelig41,42,43.
Nøjagtigheden af forudsagt dynamik mellem systemkomponenter påvirkes direkte af FF valgt til udbredelse af banen. Empirisk FF-parametriseringsindsats forfølges af få forskningsgrupper. De mest etablerede og almindelige FF for MD omfatter CHARMM39, AMBER 40, Martini44, OPLS 45 og SIRAH 46. All-atom additiv CHARMM36 (C36) kraftfelt47 anvendes i vid udstrækning til AA MD af membransystemer, da det nøjagtigt gengiver eksperimentelle strukturelle data. Det blev oprindeligt udviklet af CHARMM-samfundet, og det er kompatibelt med flere MD-motorer som GROMACS og NAMD. På trods af forbedringer på tværs af almindelige FF’er er der en kontinuerlig indsats for at forbedre parametersættene for at muliggøre forudsigelser, der nøje gengiver eksperimentelle observerbare, drevet af interesser i bestemte undersøgelsessystemer48,49.
En udfordring ved simulering af lipidmembraner er at bestemme længden af simuleringsbanen. Dette afhænger i høj grad af de målinger, der skal analyseres, og den proces, man sigter mod at karakterisere. Typisk kræver komplekse lipidblandinger længere tid at nå ligevægt, da flere arter skal have tid nok til at diffundere på membranplanet og nå en stabil lateral organisation. En simulering siges at være i ligevægt, når egenskaben af interesse har nået et plateau og svinger omkring en konstant værdi. Det er almindelig praksis at opnå mindst 100-200 ns ligevægtsbane for at udføre passende statistisk analyse af egenskaber og interaktioner af interesse. Det er almindeligt at køre membransimuleringer mellem 200-500 ns, afhængigt af kompleksiteten af lipidblandingen og forskningsspørgsmålet. Protein-lipid-interaktioner kræver typisk længere simuleringstider, mellem 500-2000 ns. Nogle tilgange til at fremskynde prøveudtagning og observerbar dynamik med membransystemer er: (i) den meget mobile membranmimetiske (HMMM) model, som erstatter slutcarbonatomer af lipider i membranen med organisk opløsningsmiddel for at fremskynde prøveudtagning50; og ii) hydrogenmasseomfordeling (HMR), som kombinerer en brøkdel af masserne af tunge atomer i et system med masserne af hydrogenatomer for at muliggøre anvendelse af et større simuleringstidstrin51.
Følgende protokol diskuterer en begyndervenlig tilgang til at bygge, køre og analysere realistiske membranmodeller ved hjælp af AA MD. I betragtning af karakteren af MD-simuleringer skal der køres flere baner for at tage højde for reproducerbarhed og korrekt statistisk analyse af resultaterne. Det er almindelig praksis at køre mindst tre replikaer pr. interessesystem. Når lipidarterne er blevet udvalgt til organismen og processen af interesse, er grundlæggende trin til at opbygge, køre og analysere en simuleringsbane for et membran-eneste system skitseret og opsummeret i figur 2.
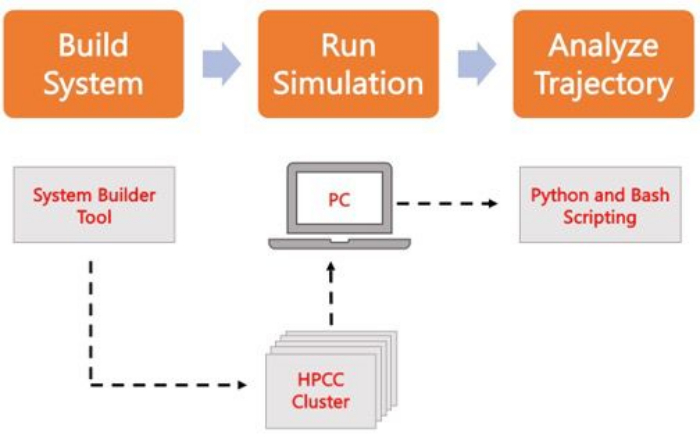
Figur 2. Skematisk til at køre MD-simuleringer. Orange bokse svarer til de tre hovedtrin, der er beskrevet i protokollen. Nedenunder er arbejdsgangen i simuleringsprocessen. Under systemopsætningen bygges systemet, der indeholder de indledende koordinater for et solvated membransystem, med en systemindgangsgenerator som CHARMM-GUI Membrane Builder. Efter overførsel af inputfilerne til en højtydende computerklynge formeres simuleringsbanen ved hjælp af en MD-motor, såsom GROMACS. Baneanalyse kan udføres på computerklyngen eller en lokal arbejdsstation sammen med visualisering. Analysen udføres derefter ved hjælp af enten pakker med indbygget analysekode såsom GROMACS og VMD eller ved hjælp af Bash-scripts eller forskellige Python-biblioteker. Klik her for at se en større version af denne figur.
Protocol
Representative Results
Discussion
Eksperimentelle teknikker kan visualisere biomolekyler ved høj opløsning ved hjælp af kryo-elektronmikroskopi (cryo-EM)58, fluorescensteknikker og atomkraftmikroskopi (AFM)59. Det er imidlertid udfordrende at fange samspillet og dynamikken i molekylære interaktioner, der ligger til grund for biologiske veje, sygdomspatogenese og terapeutisk levering på atom- eller aminosyreniveau. Her blev MD-simuleringers muligheder for at studere lipidmembraner og de vigtigste trin t…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Forfatterne takker Jinhui Li og Ricardo X. Ramirez for deres simuleringsbaner og diskussioner under skrivningen af dette manuskript. O.C. blev støttet af University at Buffalo Presidential Fellowship og National Institute of Health’s Initiative for Maximizing Student Development Training Grant 1T32GM144920-01 tildelt Margarita L. Dubocovich (PI).
Materials
| Anaconda3 | Anaconda Inc (Python & related libraries) | N/A | |
| CHARMM-GUI.org | Im lab, Lehigh University | N/A | |
| GROMACS | GROMACS development team | N/A | |
| Linux HPC Cluster | UB CCR | N/A | |
| MATLAB | MathWorks | N/A | |
| VMD | Theoretical and Computational Biophysics Group | N/A |
References
- Vanni, S., Riccardi, L., Palermo, G., De Vivo, M. Structure and Dynamics of the Acyl Chains in the Membrane Trafficking and Enzymatic Processing of Lipids. Accounts of Chemical Research. 52 (11), 3087-3096 (2019).
- Harayama, T., Riezman, H. Understanding the diversity of membrane lipid composition. Nature Reviews Molecular Cell Biology. 19 (5), 281-296 (2018).
- Tanaka, M., Edward, H. .. . E. g. e. l. m. a. n. . Comprehensive Biophysics. , 261-272 (2012).
- Bruce Alberts, A. J., Julian Lewis, , Martin Raff, , Keith Roberts, , Peter Walter, . Molecular Biology of the Cell. , (2002).
- Watson, H. Biological membranes. Essays in Biochemistry. 59, 43-69 (2015).
- Coskun, &. #. 2. 2. 0. ;., Simons, K. Cell Membranes: The Lipid Perspective. Structure. 19 (11), 1543-1548 (2011).
- Douglas G, H. a. y. e. s., Daniel, K. Y., Solaiman, D., Richard, . Biobased Surfactants (Second Edition) eds. , 515-529 (2019).
- González-Rubio, P., Gautier, R., Etchebest, C., Fuchs, P. F. J. Amphipathic-Lipid-Packing-Sensor interactions with lipids assessed by atomistic molecular dynamics. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808, 2119-2127 (2011).
- Halbleib, K., et al. Activation of the Unfolded Protein Response by Lipid Bilayer Stress. Molecular Cell. 67, 673-684 (2017).
- Andreasen, M., Lorenzen, N., Otzen, D. Interactions between misfolded protein oligomers and membranes: A central topic in neurodegenerative diseases. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1848 (9), 1897-1907 (2015).
- Calianese, D. C., Birge, R. B. Biology of phosphatidylserine (PS): basic physiology and implications in immunology, infectious disease, and cancer. Cell Commununication and Signaling. 18 (1), 41 (2020).
- Nieto-Garai, J. A., Contreras, F. X., Arboleya, A., Lorizate, M. Role of Protein-Lipid Interactions in Viral Entry. Advanced Biology. 6, 2101264 (2022).
- Mazzon, M., Mercer, J. Lipid interactions during virus entry and infection. Cell Microbiology. 16, 1493-1502 (2014).
- Colombelli, C., Aoun, M., Tiranti, V. Defective lipid metabolism in neurodegeneration with brain iron accumulation (NBIA) syndromes: not only a matter of iron. Journal of Inherited Metabolic Disease. 38 (1), 123-136 (2015).
- Saini-Chohan, H. K., Mitchell, R. W., Vaz, F. M., Zelinski, T., Hatch, G. M. Delineating the role of alterations in lipid metabolism to the pathogenesis of inherited skeletal and cardiac muscle disorders: Thematic Review Series: Genetics of Human Lipid Diseases. Journal of Lipid Research. 53 (1), 4-27 (2012).
- Martinotti, C., Ruiz-Perez, L., Deplazes, E., Mancera, R. L. Molecular Dynamics Simulation of Small Molecules Interacting with Biological Membranes. ChemPhysChem. 21 (14), 1486-1514 (2020).
- Li, J., Kalyanram, P., Rozati, S., Monje-Galvan, V., Gupta, A. Interaction of Cyanine-D112 with Binary Lipid Mixtures: Molecular Dynamics Simulation and Differential Scanning Calorimetry Study. ACS Omega. 7 (11), 9765-9774 (2022).
- Nagy, L., et al. Protein/Lipid Interaction in the Bacterial Photosynthetic Reaction Center: Phosphatidylcholine and Phosphatidylglycerol Modify the Free Energy Levels of the Quinones. Biochemistry. 43 (40), 12913-12923 (2004).
- Ramirez, R. X., Campbell, O., Pradhan, A. J., Atilla-Gokcumen, G. E., Monje-Galvan, V. Modeling the molecular fingerprint of protein-lipid interactions of MLKL on complex bilayers. Frontiers in Chemistry. 10, (2023).
- Dondelinger, Y., et al. MLKL Compromises Plasma Membrane Integrity by Binding to Phosphatidylinositol Phosphates. Cell Reports. 7 (4), 971-981 (2014).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews Molecular Cell Biology. 9 (2), 112-124 (2008).
- van Meer, G., de Kroon, A. I. P. M. Lipid map of the mammalian cell. Journal of Cell Science. 124 (1), 5 (2011).
- Lee, H. R., Lee, G. Y., You, D. G., Kim, H. K., Young, D. Y. Hepatitis C virus p7 induces membrane permeabilization by interacting with phosphatidylserine. International Journal of Molecular Sciences. 21 (3), 897 (2020).
- Casares, D., Escribá, P. V., Rosselló, C. A. Membrane Lipid Composition: Effect on Membrane and Organelle Structure, Function and Compartmentalization and Therapeutic Avenues. International Journal of Molecular Sciences. 20 (9), 2167 (2019).
- Marrink, S. J., et al. Computational Modeling of Realistic Cell Membranes. Chemical Reviews. 119 (9), 6184-6226 (2019).
- Janmey, P. A., Kinnunen, P. K. J. Biophysical properties of lipids and dynamic membranes. Trends in Cell Biology. 16 (10), 538-546 (2006).
- Brémaud, E., Favard, C., Muriaux, D. Deciphering the Assembly of Enveloped Viruses Using Model Lipid Membranes. Membranes. 12, 441 (2022).
- Campbell, O., Monje-Galvan, V. Protein-driven membrane remodeling: Molecular perspectives from Flaviviridae infections. Biophysical Journal. 122 (11), 1890-1899 (2022).
- Loschwitz, J., Olubiyi, O. O., Hub, J. S., Strodel, B., Poojari, C. S. Computer simulations of protein-membrane systems. Progress in molecular biology and translational science. 170, 273-403 (2020).
- Shell, M. S., Scott Shell, M. . Thermodynamics and Statistical Mechanics: An Integrated ApproachCambridge Series in Chemical Engineering. , 21-49 (2015).
- Yang, J., et al. Molecular Dynamic Simulation of Ni-Al Alloy-H2O Reactions Using the ReaxFF Reactive Force Field. ACS Omega. 8 (11), 9807-9814 (2023).
- Ingólfsson, H. I., Arnarez, C., Periole, X., Marrink, S. J. Computational ‘microscopy’ of cellular membranes. Journal of Cell Science. 129 (2), 257-268 (2016).
- Klauda, J. B. Perspective: Computational modeling of accurate cellular membranes with molecular resolution. The Journal of Chemical Physics. 149 (22), 220901 (2018).
- Chavent, M., Duncan, A. L., Sansom, M. S. P. Molecular dynamics simulations of membrane proteins and their interactions: from nanoscale to mesoscale. Current Opinion in Structural Biology. 40, 8-16 (2016).
- Khakbaz, P., Monje-Galvan, V., Zhuang, X., Klauda, J. B., Otto Geiger, . Biogenesis of Fatty Acids, Lipids and Membranes. , 1-19 (2017).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1, 19-25 (2015).
- Lemkul, J. A. From Proteins to Perturbed Hamiltonians: A Suite of Tutorials for the GROMACS-2018 Molecular Simulation Package. Living Journal of Computational Molecular Science. 1 (1), 5068 (2018).
- Phillips, J. C., et al. Scalable molecular dynamics on CPU and GPU architectures with NAMD. The Journal of Chemical Physics. 153 (4), 044130 (2020).
- Klauda, J. B., et al. Update of the CHARMM All-Atom Additive Force Field for Lipids: Validation on Six Lipid Types. The Journal of Physical Chemistry B. 114 (23), 7830-7843 (2010).
- Wang, J., Wolf, R. M., Caldwell, J. W., Kollman, P. A., Case, D. A. Development and testing of a general amber force field. Journal of Computational Chemistry. 25 (9), 1157-1174 (2004).
- John Stone, A. A., et al. . Using VMD. , (2011).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Hsin, J., Arkhipov, A., Yin, Y., Stone, J. E., Schulten, K. Using VMD: An Introductory Tutorial. Current Protocols in Bioinformatics. 24 (1), 5.7.1-5.7.48 (2008).
- Souza, P. C. T., et al. Martini 3: a general purpose force field for coarse-grained molecular dynamics. Nature Methods. 18 (4), 382-388 (2021).
- Jorgensen, W. L., Maxwell, D. S., Tirado-Rives, J. Development and Testing of the OPLS All-Atom Force Field on Conformational Energetics and Properties of Organic Liquids. Journal of the American Chemical Society. 118 (45), 11225-11236 (1996).
- Machado, M. R., et al. The SIRAH 2.0 Force Field: Altius, Fortius, Citius. Journal of Chemical Theory and Computation. 15 (4), 2719-2733 (2019).
- Huang, J., et al. CHARMM36m: an improved force field for folded and intrinsically disordered proteins. Nature Methods. 14 (1), 71-73 (2017).
- Mu, J., Liu, H., Zhang, J., Luo, R., Chen, H. F. Recent Force Field Strategies for Intrinsically Disordered Proteins. Journal of Chemical Information and Modeling. 61 (3), 1037-1047 (2021).
- Inakollu, V. S. S., Geerke, D. P., Rowley, C. N., Yu, H. Polarisable force fields: what do they add in biomolecular simulations. Current Opinion in Structural Biology. 61, 182-190 (2020).
- Ohkubo, Y. Z., et al. Accelerating Membrane Insertion of Peripheral Proteins with a Novel Membrane Mimetic Model. Biophysical Journal. 102 (9), 2130-2139 (2012).
- Hopkins, C. W., Le Grand, S., Walker, R. C., Roitberg, A. E. Long-Time-Step Molecular Dynamics through Hydrogen Mass Repartitioning. Journal of Chemical Theory and Computation. 11 (4), 1864-1874 (2015).
- Park, S., Beaven, A. H., Klauda, J. B., Im, W. How Tolerant are Membrane Simulations with Mismatch in Area per Lipid between Leaflets. Journal of Chemical Theory and Computation. 11 (7), 3466-3477 (2015).
- Park, S., Im, W., Pastor, R. W. Developing initial conditions for simulations of asymmetric membranes: a practical recommendation. Biophysical Journal. 120 (22), 5041-5059 (2021).
- Wu, E. L., et al. CHARMM-GUI Membrane Builder toward realistic biological membrane simulations. Journal of Computational Chemistry. 35 (27), 1997-2004 (2014).
- Center for Computational Research, U.a.B.. . CCR Facility Description. , (2019).
- Piggot, T. J., Allison, J. R., Sessions, R. B., Essex, J. W. On the Calculation of Acyl Chain Order Parameters from Lipid Simulations. Journal of Chemical Theory and Computation. 13 (11), 5683-5696 (2017).
- Li, J., Monje-Galvan, V. Effect of Glycone Diversity on the Interaction of Triterpenoid Saponins and Lipid Bilayers. ACS Applied Bio Materials. , (2023).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews Drug Discovery. 17 (7), 471-492 (2018).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Martínez, L., Andrade, R., Birgin, E. G., Martínez, J. M. PACKMOL: A package for building initial configurations for molecular dynamics simulations. Journal of Computational Chemistry. 30 (13), 2157-2164 (2009).
- Jewett, A. I., et al. Moltemplate: A Tool for Coarse-Grained Modeling of Complex Biological Matter and Soft Condensed Matter Physics. Journal of Molecular Biology. 433 (11), 166841 (2021).
- Jo, S., Kim, T., Iyer, V. G., Im, W. CHARMM-GUI: A web-based graphical user interface for CHARMM. Journal of Computational Chemistry. 29 (11), 1859-1865 (2008).
- Polêto, M. D., Lemkul, J. A. Integration of experimental data and use of automated fitting methods in developing protein force fields. Communications Chemistry. 5 (1), 38 (2022).
- Hynninen, A. P., Crowley, M. F. New faster CHARMM molecular dynamics engine. Journal of Computational Chemistry. 35 (5), 406-413 (2014).
- Kim, S. Issues on the Choice of a Proper Time Step in Molecular Dynamics. Physics Procedia. 53, 60-62 (2014).
- Grubmüller, H., Heller, H., Windemuth, A., Schulten, K. Generalized Verlet Algorithm for Efficient Molecular Dynamics Simulations with Long-range Interactions. Molecular Simulation. 6 (1-3), 121-142 (1991).
- Darden, T., York, D., Pedersen, L. Particle mesh Ewald: An N·log(N) method for Ewald sums in large systems. Journal of Chemical Physics. 98 (12), 10089-10092 (1993).
- . . Hepatitis C. , (2021).
- Braun, E., et al. Best Practices for Foundations in Molecular Simulations [Article v1.0]. Living Journal of Computational Molecular Science. 1 (1), 5957 (2018).
- Moradi, S., Nowroozi, A., Shahlaei, M. Shedding light on the structural properties of lipid bilayers using molecular dynamics simulation: a review study. RSC Advances. 9 (8), 4644-4658 (2019).
- Monje-Galvan, V., Klauda, J. B. Modeling Yeast Organelle Membranes and How Lipid Diversity Influences Bilayer Properties. Biochemistry. 54 (45), 6852-6861 (2015).
- Michaud-Agrawal, N., Denning, E. J., Woolf, T. B., Beckstein, O. MDAnalysis: A toolkit for the analysis of molecular dynamics simulations. Journal of Computational Chemistry. 32 (10), 2319-2327 (2011).
- Gowers, R., et al. MDAnalysis: A Python Package for the Rapid Analysis of Molecular Dynamics Simulations. SciPy. , (2016).
- McGibbon, R. o. b. e. r. t. . T., et al. MDTraj: A Modern Open Library for the Analysis of Molecular Dynamics Trajectories. Biophysical Journal. 109 (8), 1528-1532 (2015).
- Fortunato, M. E., Colina, C. M. pysimm: A python package for simulation of molecular systems. SoftwareX. 6, 7-12 (2017).
- Scherer, M. K., et al. PyEMMA 2: A Software Package for Estimation, Validation, and Analysis of Markov Models. Journal of Chemical Theory and Computation. 11 (11), 5525-5542 (2015).
- Song, W., et al. PyLipID: A Python Package for Analysis of Protein-Lipid Interactions from Molecular Dynamics Simulations. Journal of Chemical Theory and Computation. 18 (2), 1188-1201 (2022).
- Monje-Galvan, V., Klauda, J. B. Peripheral membrane proteins: Tying the knot between experiment and computation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1858 (7, Part B), 1584-1593 (2016).
- Monje-Galvan, V., Voth, G. A. Binding mechanism of the matrix domain of HIV-1 gag on lipid membranes. eLife. 9, e58621 (2020).
- Wang, B., Guo, C. Concentration-Dependent Effects of Cholesterol on the Dimerization of Amyloid-β Peptides in Lipid Bilayers. ACS Chemical Neuroscience. 13 (18), 2709-2718 (2022).

