Modelagem Realística de Membranas Utilizando Misturas Lipídicas Complexas em Estudos de Simulação
Summary
A diversidade lipídica da membrana em estrutura e composição é um importante contribuinte para os processos celulares e pode ser um marcador de doença. Simulações de dinâmica molecular permitem estudar membranas e suas interações com biomoléculas em resolução atomística. Aqui, fornecemos um protocolo para construir, executar e analisar sistemas complexos de membrana.
Abstract
Os lipídios são blocos de construção estruturais das membranas celulares; As espécies lipídicas variam entre organelas celulares e entre organismos. Essa variedade resulta em diferentes propriedades mecânicas e estruturais na membrana que impactam diretamente as moléculas e processos que ocorrem nessa interface. A composição lipídica é dinâmica e pode servir para modular processos de sinalização celular. Abordagens computacionais são cada vez mais usadas para prever interações entre biomoléculas e fornecer insights moleculares para observáveis experimentais. A dinâmica molecular (DM) é uma técnica baseada na mecânica estatística que prevê o movimento dos átomos com base nas forças que atuam sobre eles. Simulações MD podem ser usadas para caracterizar a interação de biomoléculas. Aqui, apresentamos brevemente a técnica, descrevemos passos práticos para iniciantes que estão interessados em simular bicamadas lipídicas, demonstramos o protocolo com software amigável para iniciantes e discutimos alternativas, desafios e considerações importantes do processo. Particularmente, enfatizamos a relevância do uso de misturas lipídicas complexas para modelar uma membrana celular de interesse para capturar os ambientes hidrofóbicos e mecânicos apropriados em simulação. Também discutimos alguns exemplos em que a composição e as propriedades da membrana modulam as interações das bicamadas com outras biomoléculas.
Introduction
Os lipídios são os principais constituintes das membranas, que fornecem limites para as células e permitem a compartimentalização intracelular 1,2,3. Os lipídios são anfifílicos, com um grupo de cabeça polar e duas caudas de ácidos graxos hidrofóbicos; Estes se auto-agrupam em uma bicamada para minimizar o contato das cadeias hidrofóbicas com a água 3,4. Várias combinações de grupos cabeçais hidrofílicos e caudas hidrofóbicas resultam em diferentes classes de lipídios nas membranas biológicas, como glicerofosfolipídios, esfingolipídios e esteróis (Figura 1)1,5,6. Os glicerofosfolipídios são blocos de construção primários das membranas celulares eucarióticas compostas por glicerofosfato, ácidos graxos de cadeia longa e grupos de cabeça de baixo peso molecular7. A nomenclatura lipídica é baseada em diferenças nos grupos de cabeça; exemplos incluem fosfatidil-colina (PC), fosfatidil-etanolamina (PE), fosfatidil-serina (PS), fosfatidil-glicerol (PG), fosfatidil-inositol (PI) ou ácido fosfatídico (PA) não modificado5,6. Quanto às caudas hidrofóbicas, o comprimento e o grau de saturação variam, juntamente com a estrutura da espinha dorsal. As combinações possíveis são inúmeras, resultando em milhares de espécies lipídicas em células demamíferos6. Alterações na composição lipídica da membrana levam a diferentes propriedades mecânicas e estruturais da membrana que afetam a atividade tanto das proteínas integrais de membrana quanto das proteínas periféricas 2,6.
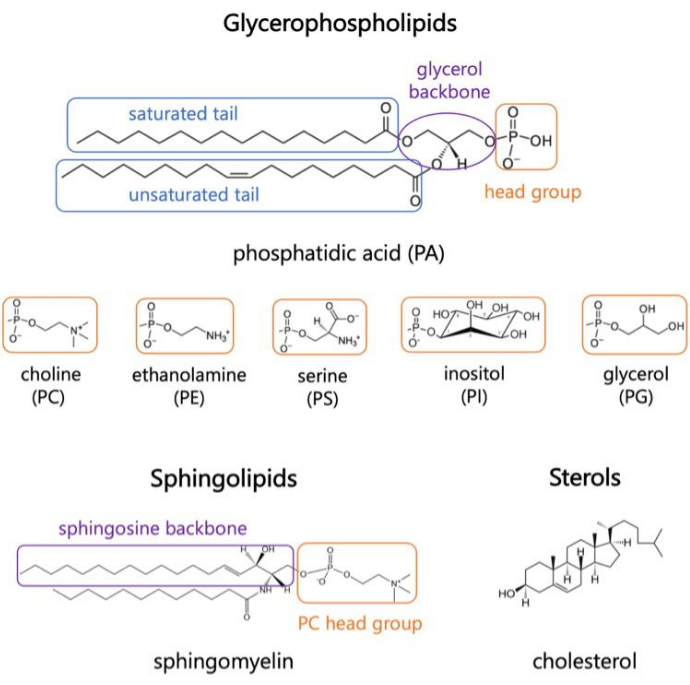
Gráfico 1. Estruturas lipídicas representativas. Caudas de ácidos graxos são mostradas em caixas azuis, grupos de cabeças lipídicas comuns em laranja e espinhas dorsais de amostra em roxo. Clique aqui para ver uma versão maior desta figura.
Os lipídios são atores ativos nos processos celulares, na ativação de proteínas em cascatas de sinalização e na homeostase de células saudáveis 8,9. Alterações na dinâmica lipídica são resultado de infecção ou podem ser marcadores de patogênese da doença10,11,12,13,14,15. Como barreiras para a célula, o estudo dos lipídios da membrana e seu papel na permeação de pequenas moléculas é de relevância para os sistemas de liberação de fármacos e mecanismos de ruptura de membrana16,17. A diversidade química e as diferentes proporções de espécies lipídicas entre organelas, tecidos e organismos dão origem a uma complexa dinâmica de membranas2. Portanto, é importante manter essas características em estudos de modelagem de bicamadas lipídicas, especialmente quando o objetivo de um estudo é examinar as interações de outras biomoléculas com a membrana. As espécies lipídicas a serem consideradas em um modelo dependem do organismo e do compartimento celular de interesse. Por exemplo, os lipídios PG são importantes para a transferência de elétrons na bateria fotossintética18, enquanto os lipídios inositol fosforilados (PIPs) são os principais agentes na dinâmica da membrana plasmática (PM) e cascatas de sinalização em células de mamíferos19,20. Dentro da célula, as membranas PM, retículo endoplasmático (RE), Golgi e mitocondrial contêm abundâncias lipídicas únicas que influenciam sua função. Por exemplo, o ER é o centro da biogênese lipídica e transporta o colesterol para o PM e Golgi; contém alta diversidade lipídica com abundância de PC e PE, mas baixo teor de esteróis, o que promove fluidez da membrana21,22,23,24. Em contrapartida, o MP incorpora centenas e até milhares de espécies lipídicas dependendo do organismo25, contém altos níveis de esfingolipídios e colesterol que lhe conferem rigidez característica em relação a outras membranas da célula24. A assimetria da cúspide deve ser considerada para membranas como a PM, que possui uma cúspide externa rica em esfingomielina, PC e colesterol, e uma cúspide interna rica em EP, PI e SP, importantes para a sinalização de cascatas24. Finalmente, a diversidade lipídica também propicia a formação de microdomínios que diferem em empacotamento e ordem interna, conhecidos como jangadas lipídicas24,26; Estes exibem assimetria lateral, são hipotetizados para desempenhar papéis importantes na sinalização celular26, e são difíceis de estudar devido à sua natureza transitória.
Técnicas experimentais como fluoroscopia, espectroscopia e sistemas de membrana modelo como vesículas unilamelares gigantes (GUVs) têm sido usadas para investigar interações de biomoléculas com membranas. No entanto, a natureza complexa e dinâmica dos componentes envolvidos é difícil de capturar apenas com métodos experimentais. Por exemplo, existem limitações na obtenção de imagens de domínios transmembrana de proteínas, na complexidade das membranas utilizadas nesses estudos e na identificação de estados intermediários ou transitórios durante o processo de interesse27,28,29. Desde o advento da simulação molecular de monocamadas e bicamadas lipídicas na década de 198029, os sistemas lipídico-proteicos e suas interações podem agora ser quantificados em nível molecular. A simulação de dinâmica molecular (DM) é uma técnica computacional comum que prevê o movimento de partículas com base em suas forças intermoleculares. Um potencial de interação aditivo descreve as interações ligadas e não ligadas entre partículas do sistema30. O conjunto de parâmetros usados para modelar essas interações é denominado campo de força de simulação (FF). Esses parâmetros são obtidos a partir de cálculos ab initio, semi-empíricos e de mecânica quântica, e otimizados para reproduzir dados de experimentos de difração de raios X e elétrons, RMN, infravermelho, espectroscopia Raman e de nêutrons, entre outros métodos31.
Simulações MD podem ser usadas para estudar sistemas em vários níveis de resolução32,33,34. Sistemas que visam caracterizar interações biomoleculares específicas, ligações de hidrogênio e outros detalhes de alta resolução são estudados com simulações de átomos (AA). Em contraste, simulações de grão grosso (CG) agrupam átomos em grupos funcionais maiores para reduzir o custo computacional e examinar a dinâmica em maior escala33. Entre esses dois estão as simulações de átomos unidos (UA), onde átomos de hidrogênio são combinados com seus respectivos átomos pesados para acelerar a computação33,35. Simulações MD são uma ferramenta poderosa para a exploração da dinâmica de membranas lipídicas e suas interações com outras moléculas e podem servir para fornecer mecanismos de nível molecular para processos de interesse na interface membrana. Adicionalmente, simulações MD podem servir para estreitar alvos experimentais e prever propriedades macromoleculares de um dado sistema com base em interações microscópicas.
Em resumo, dado um conjunto de coordenadas iniciais, velocidades e um conjunto de condições como temperatura e pressão constantes, as posições e velocidades de cada partícula são calculadas através da integração numérica do potencial de interação e da Lei do movimento de Newton. Isso se repete iterativamente, gerando uma trajetória de simulação30. Esses cálculos são realizados com um motor MD; entre vários pacotes de código aberto, o GROMACS36 é um dos mecanismos mais usados e o que descrevemos aqui. Inclui também ferramentas para análise e construção de coordenadas iniciais de sistemas a serem simulados37. Outros motores MD incluem NAMD38; CHARMM39 e AMBER40, que o usuário pode selecionar a seu próprio critério com base no desempenho computacional de um determinado sistema. É fundamental visualizar as trajetórias durante a simulação, bem como para análise e interpretação dos resultados. Uma variedade de ferramentas estão disponíveis; aqui discutimos a dinâmica molecular visual (VMD) que oferece uma ampla gama de recursos, incluindo visualização tridimensional (3D) com métodos expansivos de desenho e coloração, visualização volumétrica de dados, construção, preparação e análise de trajetórias de sistemas de simulação MD e produção de trajetória-filme sem limites no tamanho do sistema, se a memória estiver disponível41,42,43.
A precisão da dinâmica prevista entre os componentes do sistema é diretamente influenciada pelo FF escolhido para a propagação da trajetória. Os esforços empíricos de parametrização dos FF são perseguidos por poucos grupos de pesquisa. Os FF mais estabelecidos e comuns para MD incluem CHARMM39, AMBER 40, Martini44, OPLS 45 e SIRAH 46. O campo de força de CHARMM36 aditivo de todos os átomos (C36)47 é amplamente utilizado para DM AA de sistemas de membrana, pois reproduz com precisão dados estruturais experimentais. Foi originalmente desenvolvido pela comunidade CHARMM, e é compatível com vários motores MD como GROMACS e NAMD. Apesar das melhorias entre os FFs comuns, há um esforço contínuo para melhorar os conjuntos de parâmetros para permitir previsões que reproduzam de perto observáveis experimentais, impulsionados por interesses em sistemas particulares de estudo48,49.
Um desafio ao simular membranas lipídicas é determinar o comprimento da trajetória da simulação. Isso depende muito das métricas a serem analisadas e do processo que se pretende caracterizar. Tipicamente, misturas lipídicas complexas requerem maior tempo para atingir o equilíbrio, pois mais espécies devem ter tempo suficiente para se difundir no plano da membrana e alcançar uma organização lateral estável. Uma simulação é dita estar em equilíbrio quando a propriedade de interesse atingiu um platô e flutua em torno de um valor constante. É prática comum obter pelo menos 100-200 ns de trajetória equilibrada para realizar análises estatísticas apropriadas sobre as propriedades e interações de interesse. É comum executar simulações somente de membrana entre 200-500 ns, dependendo da complexidade da mistura lipídica e da questão de pesquisa. Interações proteína-lipídio tipicamente requerem tempos de simulação mais longos, entre 500-2000 ns. Algumas abordagens para acelerar a amostragem e a dinâmica observável com sistemas de membrana são: (i) o modelo mimético de membrana altamente móvel (HMMM), que substitui carbonos finais de lipídios na membrana por solvente orgânico para acelerar a amostragem50; e (ii) reparticionamento de massa de hidrogênio (HMR), que combina uma fração das massas de átomos pesados dentro de um sistema com as de átomos de hidrogênio para permitir o uso de um maior tempo de simulação51.
O protocolo a seguir discute uma abordagem amigável para iniciantes para construir, executar e analisar modelos de membrana realistas usando AA MD. Dada a natureza das simulações MD, múltiplas trajetórias devem ser executadas para dar conta da reprodutibilidade e da análise estatística adequada dos resultados. É prática corrente executar no mínimo três réplicas por sistema de interesse. Uma vez que as espécies lipídicas tenham sido selecionadas para o organismo e processo de interesse, as etapas básicas para construir, executar e analisar uma trajetória de simulação de um sistema somente de membrana são descritas e resumidas na Figura 2.
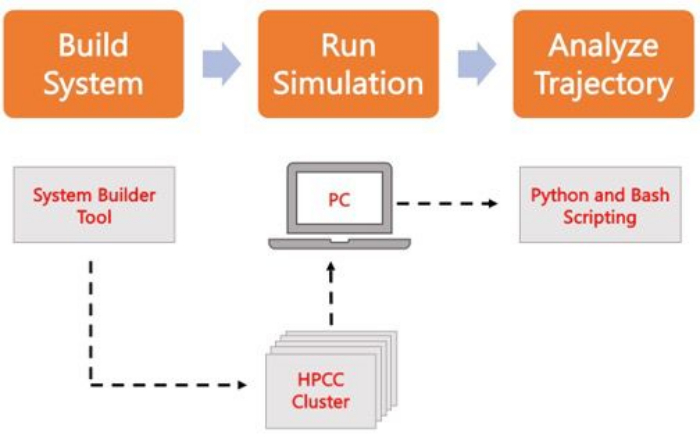
Gráfico 2. Esquema para executar simulações MD. As caixas laranja correspondem às três etapas principais descritas no protocolo. Abaixo está o fluxo de trabalho do processo de simulação. Durante a configuração do sistema, o sistema que contém as coordenadas iniciais de um sistema de membrana solvatada é construído com um gerador de entrada do sistema como o CHARMM-GUI Membrane Builder. Depois de transferir os arquivos de entrada para um cluster de computação de alto desempenho, a trajetória de simulação é propagada usando um mecanismo MD, como o GROMACS. A análise de trajetória pode ser feita no cluster de computadores ou em uma estação de trabalho local, juntamente com a visualização. A análise é então realizada, usando pacotes com código de análise interno, como GROMACS e VMD, ou usando scripts Bash ou várias bibliotecas Python. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
Técnicas experimentais podem visualizar biomoléculas em alta resolução usando microscopia crioeletrônica (cryo-EM)58, técnicas de fluorescência e microscopia de força atômica (AFM)59. No entanto, é desafiador capturar a interação e a dinâmica das interações moleculares subjacentes às vias biológicas, à patogênese da doença e à liberação terapêutica em nível atômico ou de aminoácidos. Aqui, as capacidades das simulações MD para estudar membranas …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Os autores agradecem a Jinhui Li e Ricardo X. Ramirez por suas trajetórias de simulação e discussões durante a redação deste manuscrito. O.C. foi apoiado pela University at Buffalo Presidential Fellowship e National Institute of Health’s Initiative for Maximizing Student Development Training Grant 1T32GM144920-01 concedido a Margarita L. Dubocovich (PI).
Materials
| Anaconda3 | Anaconda Inc (Python & related libraries) | N/A | |
| CHARMM-GUI.org | Im lab, Lehigh University | N/A | |
| GROMACS | GROMACS development team | N/A | |
| Linux HPC Cluster | UB CCR | N/A | |
| MATLAB | MathWorks | N/A | |
| VMD | Theoretical and Computational Biophysics Group | N/A |
References
- Vanni, S., Riccardi, L., Palermo, G., De Vivo, M. Structure and Dynamics of the Acyl Chains in the Membrane Trafficking and Enzymatic Processing of Lipids. Accounts of Chemical Research. 52 (11), 3087-3096 (2019).
- Harayama, T., Riezman, H. Understanding the diversity of membrane lipid composition. Nature Reviews Molecular Cell Biology. 19 (5), 281-296 (2018).
- Tanaka, M., Edward, H. .. . E. g. e. l. m. a. n. . Comprehensive Biophysics. , 261-272 (2012).
- Bruce Alberts, A. J., Julian Lewis, , Martin Raff, , Keith Roberts, , Peter Walter, . Molecular Biology of the Cell. , (2002).
- Watson, H. Biological membranes. Essays in Biochemistry. 59, 43-69 (2015).
- Coskun, &. #. 2. 2. 0. ;., Simons, K. Cell Membranes: The Lipid Perspective. Structure. 19 (11), 1543-1548 (2011).
- Douglas G, H. a. y. e. s., Daniel, K. Y., Solaiman, D., Richard, . Biobased Surfactants (Second Edition) eds. , 515-529 (2019).
- González-Rubio, P., Gautier, R., Etchebest, C., Fuchs, P. F. J. Amphipathic-Lipid-Packing-Sensor interactions with lipids assessed by atomistic molecular dynamics. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808, 2119-2127 (2011).
- Halbleib, K., et al. Activation of the Unfolded Protein Response by Lipid Bilayer Stress. Molecular Cell. 67, 673-684 (2017).
- Andreasen, M., Lorenzen, N., Otzen, D. Interactions between misfolded protein oligomers and membranes: A central topic in neurodegenerative diseases. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1848 (9), 1897-1907 (2015).
- Calianese, D. C., Birge, R. B. Biology of phosphatidylserine (PS): basic physiology and implications in immunology, infectious disease, and cancer. Cell Commununication and Signaling. 18 (1), 41 (2020).
- Nieto-Garai, J. A., Contreras, F. X., Arboleya, A., Lorizate, M. Role of Protein-Lipid Interactions in Viral Entry. Advanced Biology. 6, 2101264 (2022).
- Mazzon, M., Mercer, J. Lipid interactions during virus entry and infection. Cell Microbiology. 16, 1493-1502 (2014).
- Colombelli, C., Aoun, M., Tiranti, V. Defective lipid metabolism in neurodegeneration with brain iron accumulation (NBIA) syndromes: not only a matter of iron. Journal of Inherited Metabolic Disease. 38 (1), 123-136 (2015).
- Saini-Chohan, H. K., Mitchell, R. W., Vaz, F. M., Zelinski, T., Hatch, G. M. Delineating the role of alterations in lipid metabolism to the pathogenesis of inherited skeletal and cardiac muscle disorders: Thematic Review Series: Genetics of Human Lipid Diseases. Journal of Lipid Research. 53 (1), 4-27 (2012).
- Martinotti, C., Ruiz-Perez, L., Deplazes, E., Mancera, R. L. Molecular Dynamics Simulation of Small Molecules Interacting with Biological Membranes. ChemPhysChem. 21 (14), 1486-1514 (2020).
- Li, J., Kalyanram, P., Rozati, S., Monje-Galvan, V., Gupta, A. Interaction of Cyanine-D112 with Binary Lipid Mixtures: Molecular Dynamics Simulation and Differential Scanning Calorimetry Study. ACS Omega. 7 (11), 9765-9774 (2022).
- Nagy, L., et al. Protein/Lipid Interaction in the Bacterial Photosynthetic Reaction Center: Phosphatidylcholine and Phosphatidylglycerol Modify the Free Energy Levels of the Quinones. Biochemistry. 43 (40), 12913-12923 (2004).
- Ramirez, R. X., Campbell, O., Pradhan, A. J., Atilla-Gokcumen, G. E., Monje-Galvan, V. Modeling the molecular fingerprint of protein-lipid interactions of MLKL on complex bilayers. Frontiers in Chemistry. 10, (2023).
- Dondelinger, Y., et al. MLKL Compromises Plasma Membrane Integrity by Binding to Phosphatidylinositol Phosphates. Cell Reports. 7 (4), 971-981 (2014).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews Molecular Cell Biology. 9 (2), 112-124 (2008).
- van Meer, G., de Kroon, A. I. P. M. Lipid map of the mammalian cell. Journal of Cell Science. 124 (1), 5 (2011).
- Lee, H. R., Lee, G. Y., You, D. G., Kim, H. K., Young, D. Y. Hepatitis C virus p7 induces membrane permeabilization by interacting with phosphatidylserine. International Journal of Molecular Sciences. 21 (3), 897 (2020).
- Casares, D., Escribá, P. V., Rosselló, C. A. Membrane Lipid Composition: Effect on Membrane and Organelle Structure, Function and Compartmentalization and Therapeutic Avenues. International Journal of Molecular Sciences. 20 (9), 2167 (2019).
- Marrink, S. J., et al. Computational Modeling of Realistic Cell Membranes. Chemical Reviews. 119 (9), 6184-6226 (2019).
- Janmey, P. A., Kinnunen, P. K. J. Biophysical properties of lipids and dynamic membranes. Trends in Cell Biology. 16 (10), 538-546 (2006).
- Brémaud, E., Favard, C., Muriaux, D. Deciphering the Assembly of Enveloped Viruses Using Model Lipid Membranes. Membranes. 12, 441 (2022).
- Campbell, O., Monje-Galvan, V. Protein-driven membrane remodeling: Molecular perspectives from Flaviviridae infections. Biophysical Journal. 122 (11), 1890-1899 (2022).
- Loschwitz, J., Olubiyi, O. O., Hub, J. S., Strodel, B., Poojari, C. S. Computer simulations of protein-membrane systems. Progress in molecular biology and translational science. 170, 273-403 (2020).
- Shell, M. S., Scott Shell, M. . Thermodynamics and Statistical Mechanics: An Integrated ApproachCambridge Series in Chemical Engineering. , 21-49 (2015).
- Yang, J., et al. Molecular Dynamic Simulation of Ni-Al Alloy-H2O Reactions Using the ReaxFF Reactive Force Field. ACS Omega. 8 (11), 9807-9814 (2023).
- Ingólfsson, H. I., Arnarez, C., Periole, X., Marrink, S. J. Computational ‘microscopy’ of cellular membranes. Journal of Cell Science. 129 (2), 257-268 (2016).
- Klauda, J. B. Perspective: Computational modeling of accurate cellular membranes with molecular resolution. The Journal of Chemical Physics. 149 (22), 220901 (2018).
- Chavent, M., Duncan, A. L., Sansom, M. S. P. Molecular dynamics simulations of membrane proteins and their interactions: from nanoscale to mesoscale. Current Opinion in Structural Biology. 40, 8-16 (2016).
- Khakbaz, P., Monje-Galvan, V., Zhuang, X., Klauda, J. B., Otto Geiger, . Biogenesis of Fatty Acids, Lipids and Membranes. , 1-19 (2017).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1, 19-25 (2015).
- Lemkul, J. A. From Proteins to Perturbed Hamiltonians: A Suite of Tutorials for the GROMACS-2018 Molecular Simulation Package. Living Journal of Computational Molecular Science. 1 (1), 5068 (2018).
- Phillips, J. C., et al. Scalable molecular dynamics on CPU and GPU architectures with NAMD. The Journal of Chemical Physics. 153 (4), 044130 (2020).
- Klauda, J. B., et al. Update of the CHARMM All-Atom Additive Force Field for Lipids: Validation on Six Lipid Types. The Journal of Physical Chemistry B. 114 (23), 7830-7843 (2010).
- Wang, J., Wolf, R. M., Caldwell, J. W., Kollman, P. A., Case, D. A. Development and testing of a general amber force field. Journal of Computational Chemistry. 25 (9), 1157-1174 (2004).
- John Stone, A. A., et al. . Using VMD. , (2011).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Hsin, J., Arkhipov, A., Yin, Y., Stone, J. E., Schulten, K. Using VMD: An Introductory Tutorial. Current Protocols in Bioinformatics. 24 (1), 5.7.1-5.7.48 (2008).
- Souza, P. C. T., et al. Martini 3: a general purpose force field for coarse-grained molecular dynamics. Nature Methods. 18 (4), 382-388 (2021).
- Jorgensen, W. L., Maxwell, D. S., Tirado-Rives, J. Development and Testing of the OPLS All-Atom Force Field on Conformational Energetics and Properties of Organic Liquids. Journal of the American Chemical Society. 118 (45), 11225-11236 (1996).
- Machado, M. R., et al. The SIRAH 2.0 Force Field: Altius, Fortius, Citius. Journal of Chemical Theory and Computation. 15 (4), 2719-2733 (2019).
- Huang, J., et al. CHARMM36m: an improved force field for folded and intrinsically disordered proteins. Nature Methods. 14 (1), 71-73 (2017).
- Mu, J., Liu, H., Zhang, J., Luo, R., Chen, H. F. Recent Force Field Strategies for Intrinsically Disordered Proteins. Journal of Chemical Information and Modeling. 61 (3), 1037-1047 (2021).
- Inakollu, V. S. S., Geerke, D. P., Rowley, C. N., Yu, H. Polarisable force fields: what do they add in biomolecular simulations. Current Opinion in Structural Biology. 61, 182-190 (2020).
- Ohkubo, Y. Z., et al. Accelerating Membrane Insertion of Peripheral Proteins with a Novel Membrane Mimetic Model. Biophysical Journal. 102 (9), 2130-2139 (2012).
- Hopkins, C. W., Le Grand, S., Walker, R. C., Roitberg, A. E. Long-Time-Step Molecular Dynamics through Hydrogen Mass Repartitioning. Journal of Chemical Theory and Computation. 11 (4), 1864-1874 (2015).
- Park, S., Beaven, A. H., Klauda, J. B., Im, W. How Tolerant are Membrane Simulations with Mismatch in Area per Lipid between Leaflets. Journal of Chemical Theory and Computation. 11 (7), 3466-3477 (2015).
- Park, S., Im, W., Pastor, R. W. Developing initial conditions for simulations of asymmetric membranes: a practical recommendation. Biophysical Journal. 120 (22), 5041-5059 (2021).
- Wu, E. L., et al. CHARMM-GUI Membrane Builder toward realistic biological membrane simulations. Journal of Computational Chemistry. 35 (27), 1997-2004 (2014).
- Center for Computational Research, U.a.B.. . CCR Facility Description. , (2019).
- Piggot, T. J., Allison, J. R., Sessions, R. B., Essex, J. W. On the Calculation of Acyl Chain Order Parameters from Lipid Simulations. Journal of Chemical Theory and Computation. 13 (11), 5683-5696 (2017).
- Li, J., Monje-Galvan, V. Effect of Glycone Diversity on the Interaction of Triterpenoid Saponins and Lipid Bilayers. ACS Applied Bio Materials. , (2023).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews Drug Discovery. 17 (7), 471-492 (2018).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Martínez, L., Andrade, R., Birgin, E. G., Martínez, J. M. PACKMOL: A package for building initial configurations for molecular dynamics simulations. Journal of Computational Chemistry. 30 (13), 2157-2164 (2009).
- Jewett, A. I., et al. Moltemplate: A Tool for Coarse-Grained Modeling of Complex Biological Matter and Soft Condensed Matter Physics. Journal of Molecular Biology. 433 (11), 166841 (2021).
- Jo, S., Kim, T., Iyer, V. G., Im, W. CHARMM-GUI: A web-based graphical user interface for CHARMM. Journal of Computational Chemistry. 29 (11), 1859-1865 (2008).
- Polêto, M. D., Lemkul, J. A. Integration of experimental data and use of automated fitting methods in developing protein force fields. Communications Chemistry. 5 (1), 38 (2022).
- Hynninen, A. P., Crowley, M. F. New faster CHARMM molecular dynamics engine. Journal of Computational Chemistry. 35 (5), 406-413 (2014).
- Kim, S. Issues on the Choice of a Proper Time Step in Molecular Dynamics. Physics Procedia. 53, 60-62 (2014).
- Grubmüller, H., Heller, H., Windemuth, A., Schulten, K. Generalized Verlet Algorithm for Efficient Molecular Dynamics Simulations with Long-range Interactions. Molecular Simulation. 6 (1-3), 121-142 (1991).
- Darden, T., York, D., Pedersen, L. Particle mesh Ewald: An N·log(N) method for Ewald sums in large systems. Journal of Chemical Physics. 98 (12), 10089-10092 (1993).
- . . Hepatitis C. , (2021).
- Braun, E., et al. Best Practices for Foundations in Molecular Simulations [Article v1.0]. Living Journal of Computational Molecular Science. 1 (1), 5957 (2018).
- Moradi, S., Nowroozi, A., Shahlaei, M. Shedding light on the structural properties of lipid bilayers using molecular dynamics simulation: a review study. RSC Advances. 9 (8), 4644-4658 (2019).
- Monje-Galvan, V., Klauda, J. B. Modeling Yeast Organelle Membranes and How Lipid Diversity Influences Bilayer Properties. Biochemistry. 54 (45), 6852-6861 (2015).
- Michaud-Agrawal, N., Denning, E. J., Woolf, T. B., Beckstein, O. MDAnalysis: A toolkit for the analysis of molecular dynamics simulations. Journal of Computational Chemistry. 32 (10), 2319-2327 (2011).
- Gowers, R., et al. MDAnalysis: A Python Package for the Rapid Analysis of Molecular Dynamics Simulations. SciPy. , (2016).
- McGibbon, R. o. b. e. r. t. . T., et al. MDTraj: A Modern Open Library for the Analysis of Molecular Dynamics Trajectories. Biophysical Journal. 109 (8), 1528-1532 (2015).
- Fortunato, M. E., Colina, C. M. pysimm: A python package for simulation of molecular systems. SoftwareX. 6, 7-12 (2017).
- Scherer, M. K., et al. PyEMMA 2: A Software Package for Estimation, Validation, and Analysis of Markov Models. Journal of Chemical Theory and Computation. 11 (11), 5525-5542 (2015).
- Song, W., et al. PyLipID: A Python Package for Analysis of Protein-Lipid Interactions from Molecular Dynamics Simulations. Journal of Chemical Theory and Computation. 18 (2), 1188-1201 (2022).
- Monje-Galvan, V., Klauda, J. B. Peripheral membrane proteins: Tying the knot between experiment and computation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1858 (7, Part B), 1584-1593 (2016).
- Monje-Galvan, V., Voth, G. A. Binding mechanism of the matrix domain of HIV-1 gag on lipid membranes. eLife. 9, e58621 (2020).
- Wang, B., Guo, C. Concentration-Dependent Effects of Cholesterol on the Dimerization of Amyloid-β Peptides in Lipid Bilayers. ACS Chemical Neuroscience. 13 (18), 2709-2718 (2022).

