Реалистичное моделирование мембран с использованием сложных липидных смесей в имитационных исследованиях
Summary
Липидное разнообразие мембран по структуре и составу является важным фактором, влияющим на клеточные процессы, и может быть маркером заболевания. Молекулярно-динамическое моделирование позволяет изучать мембраны и их взаимодействие с биомолекулами с атомистическим разрешением. Здесь мы предоставляем протокол для создания, запуска и анализа сложных мембранных систем.
Abstract
Липиды являются структурными строительными блоками клеточных мембран; Виды липидов различаются в разных клеточных органеллах и в разных организмах. Это разнообразие приводит к различным механическим и структурным свойствам мембраны, которые непосредственно влияют на молекулы и процессы, происходящие на этой границе. Липидный состав динамичен и может служить для модуляции клеточных сигнальных процессов. Вычислительные подходы все чаще используются для прогнозирования взаимодействий между биомолекулами и получения молекулярной информации для экспериментальных наблюдений. Молекулярная динамика (МД) — это метод, основанный на статистической механике, который предсказывает движение атомов на основе сил, которые на них действуют. МД-моделирование может быть использовано для характеристики взаимодействия биомолекул. Здесь мы кратко представим технику, опишем практические шаги для начинающих, заинтересованных в моделировании липидных бислоев, продемонстрируем протокол с помощью программного обеспечения, удобного для начинающих, и обсудим альтернативы, проблемы и важные соображения процесса. В частности, мы подчеркиваем актуальность использования сложных липидных смесей для моделирования интересующей клеточной мембраны для захвата соответствующих гидрофобных и механических сред при моделировании. Мы также обсудим некоторые примеры, когда состав и свойства мембраны модулируют взаимодействие бислоев с другими биомолекулами.
Introduction
Липиды являются основными компонентами мембран, которые обеспечивают границы для клеток и обеспечивают внутриклеточную компартментализацию 1,2,3. Липиды амфифильные, с полярной головной группой и двумя гидрофобными хвостами жирных кислот; Они самоорганизуются в бислой, чтобы свести к минимуму контакт гидрофобных цепей с водой 3,4. Различные комбинации гидрофильных головных групп и гидрофобных хвостов приводят к образованию различных классов липидов в биологических мембранах, таких как глицерофосфолипиды, сфинголипиды и стерины (рис. 1)1,5,6. Глицерофосфолипиды являются первичными строительными блоками мембран эукариотических клеток, состоящих из глицерофосфата, длинноцепочечных жирных кислот и головных групп с низкой молекулярной массой7. Липидная номенклатура основана на различиях в головных группах; Например, фосфатидилхолин (ФК), фосфатидилэтаноламин (ПЭ), фосфатидилсерин (ФС), фосфатидилглицерин (ПГ), фосфатидил-инозитол (ПИ) или немодифицированная фосфатидная кислота (ПА)5,6. Что касается гидрофобных хвостов, то длина и степень насыщения варьируются, как и структура позвоночника. Возможные комбинации многочисленны, в результате чего в клетках млекопитающих образуются тысячи видов липидов6. Изменения липидного состава мембран приводят к различным механическим и структурным свойствам мембраны, которые влияют на активность как интегральных мембранных белков, так и периферических белков 2,6.
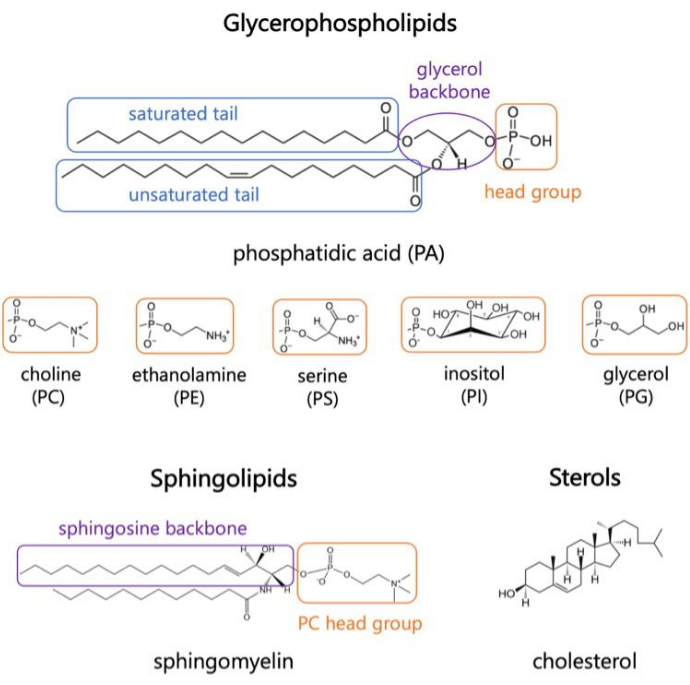
Рисунок 1. Репрезентативные липидные структуры. Хвосты жирных кислот показаны синими прямоугольниками, общие липидные группы головок — оранжевым, а костяк образцов — фиолетовым. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
Липиды являются активными игроками в клеточных процессах, активации белков в сигнальных каскадах и здоровом клеточном гомеостазе 8,9. Измененная липидная динамика является следствием инфекции или может быть маркерами патогенеза заболевания 10,11,12,13,14,15. Изучение мембранных липидов и их роли в проникновении малых молекул в качестве барьеров для клетки имеет значение для систем доставки лекарств и механизмов разрушения мембран16,17. Химическое разнообразие и различное соотношение видов липидов в органеллах, тканях и организмах приводят к сложной мембранной динамике. Поэтому важно сохранить эти характеристики при моделировании липидных бислоев, особенно когда целью исследования является изучение взаимодействия других биомолекул с мембраной. Виды липидов, которые следует учитывать в модели, зависят от организма и интересующего клеточного компартмента. Например, PG-липиды важны для переноса электронов в фотосинтетических bateria18, в то время как фосфорилированные инозитоловые липиды (PIPs) играют важную роль в динамике плазматической мембраны (PM) и сигнальных каскадах в клетках млекопитающих 19,20. Внутри клетки PM, эндоплазматический ретикулум (ER), мембраны Гольджи и митохондрии содержат уникальное количество липидов, которые влияют на их функцию. Например, ER является центром липидного биогенеза и транспортирует холестерин к PM и Гольджи; он содержит высокое липидное разнообразие с обилием ПК и ПЭ, но низкое содержание стеринов, что способствует текучести мембран21,22,23,24. Напротив, PM включает в себя сотни и даже тысячи видов липидов в зависимости от организма25, он содержит высокие уровни сфинголипидов и холестерина, которые придают ему характерную жесткость по сравнению с другими мембранами в клетке24. Асимметрию створок следует учитывать для мембран, таких как ПМ, которая имеет внешнюю створку, богатую сфингомиелином, ПК и холестерином, и внутреннюю створку, богатую ПЭ, ПИ и ФС, которые важны для сигнальных каскадов24. Наконец, липидное разнообразие также приводит к образованию микродоменов, различающихся по упаковке и внутреннему порядку, известных как липидные рафты24,26; Они демонстрируют латеральную асимметрию, предположительно играют важную роль в клеточнойсигнализации, и их трудно изучать из-за их переходной природы.
Экспериментальные методы, такие как рентгеноскопия, спектроскопия и модельные мембранные системы, такие как гигантские одноламеллярные везикулы (ГУВ), были использованы для исследования взаимодействия биомолекул с мембранами. Тем не менее, сложную и динамичную природу задействованных компонентов трудно охватить только экспериментальными методами. Например, существуют ограничения на визуализацию трансмембранных доменов белков, сложность мембран, используемых в таких исследованиях, и идентификацию промежуточных или переходных состояний во время интересующего процесса27,28,29. С момента появления молекулярного моделирования липидных монослоев и бислоев в 1980-х годах29 липидно-белковые системы и их взаимодействия теперь могут быть количественно оценены на молекулярном уровне. Моделирование молекулярной динамики (МД) — это распространенный вычислительный метод, который предсказывает движение частиц на основе их межмолекулярных сил. Потенциал аддитивного взаимодействия описывает связанные и несвязанные взаимодействия между частицами системы30. Набор параметров, используемых для моделирования этих взаимодействий, называется силовым полем моделирования (FF). Эти параметры получены в результате вычислений ab initio, полуэмпирических и квантово-механических расчетов и оптимизированы для воспроизведения данных рентгеновских и электронных дифракционных экспериментов, ЯМР, инфракрасной, комбинационной и нейтронной спектроскопии, а также других методов31.
МД-моделирование может быть использовано для исследования систем с различными уровнями разрешения32,33,34. Системы, целью которых является характеристика конкретных биомолекулярных взаимодействий, водородных связей и других деталей с высоким разрешением, изучаются с помощью моделирования всех атомов (АА). В отличие от этого, крупнозернистое (CG) моделирование объединяет атомы в более крупные функциональные группы для снижения вычислительных затрат иизучения крупномасштабной динамики. Между этими двумя моделями находятся модели объединенных атомов (UA), в которых атомы водорода объединяются с соответствующими тяжелыми атомами для ускорения вычислений33,35. Моделирование МД является мощным инструментом для изучения динамики липидных мембран и их взаимодействия с другими молекулами и может служить для создания механизмов на молекулярном уровне для интересующих процессов на границе раздела мембран. Кроме того, МД-моделирование может служить для сужения экспериментальных мишеней и прогнозирования макромолекулярных свойств данной системы на основе микроскопических взаимодействий.
Короче говоря, имея набор начальных координат, скоростей и набор условий, таких как постоянная температура и давление, положения и скорости каждой частицы вычисляются путем численного интегрирования потенциала взаимодействия и закона движения Ньютона. Это повторяется итеративно, тем самым генерируя траекториюмоделирования 30. Эти вычисления выполняются с помощью движка MD; Среди нескольких пакетов с открытым исходным кодом GROMACS36 является одним из наиболее часто используемых движков, и именно его мы опишем здесь. Он также включает в себя инструменты для анализа и построения начальных координат моделируемых систем37. Другие двигатели MD включают NAMD38; CHARMM39 и AMBER40, которые пользователь может выбрать по своему усмотрению в зависимости от вычислительной производительности данной системы. Очень важно визуализировать траектории во время моделирования, а также для анализа и интерпретации результатов. Доступны различные инструменты; Здесь мы обсудим визуальную молекулярную динамику (VMD), которая предлагает широкий спектр возможностей, включая трехмерную (3-D) визуализацию с расширенными методами рисования и раскрашивания, визуализацию объемных данных, построение, подготовку и анализ траекторий систем моделирования МД, а также создание траекторных фильмов без ограничений по размеру системы, если доступна память41,42,43.
На точность предсказанной динамики между компонентами системы напрямую влияет ФФ, выбранный для распространения траектории. Эмпирическая параметризация ФФ предпринимается несколькими исследовательскими группами. Наиболее распространенными и распространенными FF для MD являются CHARMM39, AMBER 40, Martini44, OPLS 45 и SIRAH 46. Силовое поле47 из всех атомов CHARMM36 (C36) широко используется для АА МД мембранных систем, так как оно точно воспроизводит экспериментальные структурные данные. Первоначально он был разработан сообществом CHARMM и совместим с несколькими движками MD, такими как GROMACS и NAMD. Несмотря на усовершенствования во всех распространенных FF, предпринимаются постоянные усилия по совершенствованию наборов параметров, позволяющих делать прогнозы, точно воспроизводящие экспериментальные наблюдаемые объекты, что обусловлено интересами к конкретным системам исследования48,49.
Сложность при моделировании липидных мембран заключается в определении длины траектории моделирования. Это во многом зависит от анализируемых показателей и процесса, который мы хотим охарактеризовать. Как правило, сложным липидным смесям требуется больше времени для достижения равновесия, так как большее количество видов должно иметь достаточно времени, чтобы диффундировать в плоскости мембраны и достичь стабильной латеральной организации. Моделирование называется равновесным, когда интересующее свойство достигло плато и колеблется около постоянной величины. Общепринятой практикой является получение по крайней мере 100-200 нс равновесной траектории для проведения соответствующего статистического анализа интересующих свойств и взаимодействий. Обычно моделирование только мембраны выполняется в диапазоне от 200 до 500 нс, в зависимости от сложности липидной смеси и исследовательского вопроса. Белок-липидные взаимодействия обычно требуют более длительного времени моделирования, от 500 до 2000 нс. Некоторые подходы к ускорению отбора проб и наблюдаемой динамике с помощью мембранных систем включают: (i) высокоподвижную мембранно-миметическую модель (HMMM), в которой конечные углероды липидов в мембране заменяются органическим растворителем для ускорения отбора проб50; и (ii) перераспределение массы водорода (HMR), которое объединяет часть масс тяжелых атомов в системе с массами атомов водорода, что позволяет использовать больший временной шаг51 моделирования.
В следующем протоколе описывается удобный для начинающих подход к построению, запуску и анализу реалистичных мембранных моделей с использованием AA MD. Учитывая природу моделирования МД, необходимо использовать несколько траекторий для обеспечения воспроизводимости и надлежащего статистического анализа результатов. В настоящее время рекомендуется запускать не менее трех реплик на интересующую систему. После того, как липидные формы были выбраны для интересующего организма и процесса, основные шаги по построению, запуску и анализу траектории моделирования мембранной системы описаны и обобщены на рисунке 2.
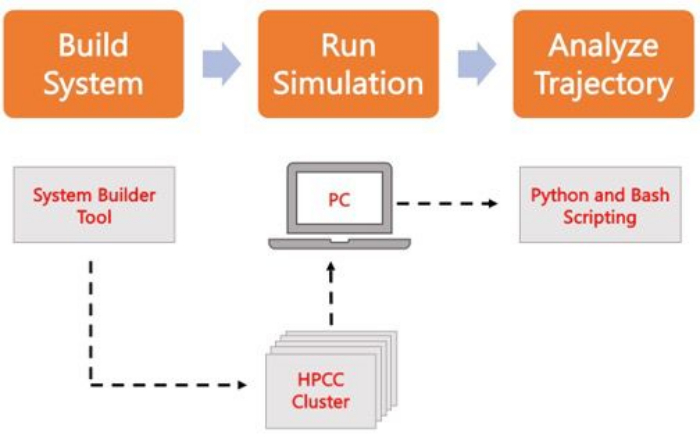
Рисунок 2. Schematic для запуска MD-моделирования. Оранжевые прямоугольники соответствуют трем основным шагам, описанным в протоколе. Ниже показан рабочий процесс моделирования. Во время настройки системы система, содержащая начальные координаты сольватированной мембранной системы, строится с помощью системного генератора входных данных, такого как CHARMM-GUI Membrane Builder. После передачи входных файлов в кластер высокопроизводительных вычислений траектория моделирования распространяется с помощью движка MD, такого как GROMACS. Анализ траектории может быть выполнен на вычислительном кластере или локальной рабочей станции вместе с визуализацией. Затем анализ выполняется либо с помощью пакетов со встроенным кодом анализа, таких как GROMACS и VMD, либо с помощью сценариев Bash или различных библиотек Python. Пожалуйста, нажмите здесь, чтобы увидеть увеличенную версию этого рисунка.
Protocol
Representative Results
Discussion
Экспериментальные методы позволяют визуализировать биомолекулы с высоким разрешением с помощью криоэлектронной микроскопии (крио-ЭМ)58, флуоресцентных методов и атомно-силовой микроскопии (АСМ)59. Тем не менее, сложно уловить взаимодействие и динамику молекул…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Авторы благодарят Цзиньхуэй Ли (Jinhui Li) и Рикардо Х. Рамиреса (Ricardo X. Ramirez) за их моделирование траекторий и дискуссии во время написания этой статьи. O.C. был поддержан Президентской стипендией Университета в Буффало и грантом 1T32GM144920-01 Национального института здравоохранения по максимальному развитию студентов, присужденным Маргарите Л. Дубокович (PI).
Materials
| Anaconda3 | Anaconda Inc (Python & related libraries) | N/A | |
| CHARMM-GUI.org | Im lab, Lehigh University | N/A | |
| GROMACS | GROMACS development team | N/A | |
| Linux HPC Cluster | UB CCR | N/A | |
| MATLAB | MathWorks | N/A | |
| VMD | Theoretical and Computational Biophysics Group | N/A |
References
- Vanni, S., Riccardi, L., Palermo, G., De Vivo, M. Structure and Dynamics of the Acyl Chains in the Membrane Trafficking and Enzymatic Processing of Lipids. Accounts of Chemical Research. 52 (11), 3087-3096 (2019).
- Harayama, T., Riezman, H. Understanding the diversity of membrane lipid composition. Nature Reviews Molecular Cell Biology. 19 (5), 281-296 (2018).
- Tanaka, M., Edward, H. .. . E. g. e. l. m. a. n. . Comprehensive Biophysics. , 261-272 (2012).
- Bruce Alberts, A. J., Julian Lewis, , Martin Raff, , Keith Roberts, , Peter Walter, . Molecular Biology of the Cell. , (2002).
- Watson, H. Biological membranes. Essays in Biochemistry. 59, 43-69 (2015).
- Coskun, &. #. 2. 2. 0. ;., Simons, K. Cell Membranes: The Lipid Perspective. Structure. 19 (11), 1543-1548 (2011).
- Douglas G, H. a. y. e. s., Daniel, K. Y., Solaiman, D., Richard, . Biobased Surfactants (Second Edition) eds. , 515-529 (2019).
- González-Rubio, P., Gautier, R., Etchebest, C., Fuchs, P. F. J. Amphipathic-Lipid-Packing-Sensor interactions with lipids assessed by atomistic molecular dynamics. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808, 2119-2127 (2011).
- Halbleib, K., et al. Activation of the Unfolded Protein Response by Lipid Bilayer Stress. Molecular Cell. 67, 673-684 (2017).
- Andreasen, M., Lorenzen, N., Otzen, D. Interactions between misfolded protein oligomers and membranes: A central topic in neurodegenerative diseases. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1848 (9), 1897-1907 (2015).
- Calianese, D. C., Birge, R. B. Biology of phosphatidylserine (PS): basic physiology and implications in immunology, infectious disease, and cancer. Cell Commununication and Signaling. 18 (1), 41 (2020).
- Nieto-Garai, J. A., Contreras, F. X., Arboleya, A., Lorizate, M. Role of Protein-Lipid Interactions in Viral Entry. Advanced Biology. 6, 2101264 (2022).
- Mazzon, M., Mercer, J. Lipid interactions during virus entry and infection. Cell Microbiology. 16, 1493-1502 (2014).
- Colombelli, C., Aoun, M., Tiranti, V. Defective lipid metabolism in neurodegeneration with brain iron accumulation (NBIA) syndromes: not only a matter of iron. Journal of Inherited Metabolic Disease. 38 (1), 123-136 (2015).
- Saini-Chohan, H. K., Mitchell, R. W., Vaz, F. M., Zelinski, T., Hatch, G. M. Delineating the role of alterations in lipid metabolism to the pathogenesis of inherited skeletal and cardiac muscle disorders: Thematic Review Series: Genetics of Human Lipid Diseases. Journal of Lipid Research. 53 (1), 4-27 (2012).
- Martinotti, C., Ruiz-Perez, L., Deplazes, E., Mancera, R. L. Molecular Dynamics Simulation of Small Molecules Interacting with Biological Membranes. ChemPhysChem. 21 (14), 1486-1514 (2020).
- Li, J., Kalyanram, P., Rozati, S., Monje-Galvan, V., Gupta, A. Interaction of Cyanine-D112 with Binary Lipid Mixtures: Molecular Dynamics Simulation and Differential Scanning Calorimetry Study. ACS Omega. 7 (11), 9765-9774 (2022).
- Nagy, L., et al. Protein/Lipid Interaction in the Bacterial Photosynthetic Reaction Center: Phosphatidylcholine and Phosphatidylglycerol Modify the Free Energy Levels of the Quinones. Biochemistry. 43 (40), 12913-12923 (2004).
- Ramirez, R. X., Campbell, O., Pradhan, A. J., Atilla-Gokcumen, G. E., Monje-Galvan, V. Modeling the molecular fingerprint of protein-lipid interactions of MLKL on complex bilayers. Frontiers in Chemistry. 10, (2023).
- Dondelinger, Y., et al. MLKL Compromises Plasma Membrane Integrity by Binding to Phosphatidylinositol Phosphates. Cell Reports. 7 (4), 971-981 (2014).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews Molecular Cell Biology. 9 (2), 112-124 (2008).
- van Meer, G., de Kroon, A. I. P. M. Lipid map of the mammalian cell. Journal of Cell Science. 124 (1), 5 (2011).
- Lee, H. R., Lee, G. Y., You, D. G., Kim, H. K., Young, D. Y. Hepatitis C virus p7 induces membrane permeabilization by interacting with phosphatidylserine. International Journal of Molecular Sciences. 21 (3), 897 (2020).
- Casares, D., Escribá, P. V., Rosselló, C. A. Membrane Lipid Composition: Effect on Membrane and Organelle Structure, Function and Compartmentalization and Therapeutic Avenues. International Journal of Molecular Sciences. 20 (9), 2167 (2019).
- Marrink, S. J., et al. Computational Modeling of Realistic Cell Membranes. Chemical Reviews. 119 (9), 6184-6226 (2019).
- Janmey, P. A., Kinnunen, P. K. J. Biophysical properties of lipids and dynamic membranes. Trends in Cell Biology. 16 (10), 538-546 (2006).
- Brémaud, E., Favard, C., Muriaux, D. Deciphering the Assembly of Enveloped Viruses Using Model Lipid Membranes. Membranes. 12, 441 (2022).
- Campbell, O., Monje-Galvan, V. Protein-driven membrane remodeling: Molecular perspectives from Flaviviridae infections. Biophysical Journal. 122 (11), 1890-1899 (2022).
- Loschwitz, J., Olubiyi, O. O., Hub, J. S., Strodel, B., Poojari, C. S. Computer simulations of protein-membrane systems. Progress in molecular biology and translational science. 170, 273-403 (2020).
- Shell, M. S., Scott Shell, M. . Thermodynamics and Statistical Mechanics: An Integrated ApproachCambridge Series in Chemical Engineering. , 21-49 (2015).
- Yang, J., et al. Molecular Dynamic Simulation of Ni-Al Alloy-H2O Reactions Using the ReaxFF Reactive Force Field. ACS Omega. 8 (11), 9807-9814 (2023).
- Ingólfsson, H. I., Arnarez, C., Periole, X., Marrink, S. J. Computational ‘microscopy’ of cellular membranes. Journal of Cell Science. 129 (2), 257-268 (2016).
- Klauda, J. B. Perspective: Computational modeling of accurate cellular membranes with molecular resolution. The Journal of Chemical Physics. 149 (22), 220901 (2018).
- Chavent, M., Duncan, A. L., Sansom, M. S. P. Molecular dynamics simulations of membrane proteins and their interactions: from nanoscale to mesoscale. Current Opinion in Structural Biology. 40, 8-16 (2016).
- Khakbaz, P., Monje-Galvan, V., Zhuang, X., Klauda, J. B., Otto Geiger, . Biogenesis of Fatty Acids, Lipids and Membranes. , 1-19 (2017).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1, 19-25 (2015).
- Lemkul, J. A. From Proteins to Perturbed Hamiltonians: A Suite of Tutorials for the GROMACS-2018 Molecular Simulation Package. Living Journal of Computational Molecular Science. 1 (1), 5068 (2018).
- Phillips, J. C., et al. Scalable molecular dynamics on CPU and GPU architectures with NAMD. The Journal of Chemical Physics. 153 (4), 044130 (2020).
- Klauda, J. B., et al. Update of the CHARMM All-Atom Additive Force Field for Lipids: Validation on Six Lipid Types. The Journal of Physical Chemistry B. 114 (23), 7830-7843 (2010).
- Wang, J., Wolf, R. M., Caldwell, J. W., Kollman, P. A., Case, D. A. Development and testing of a general amber force field. Journal of Computational Chemistry. 25 (9), 1157-1174 (2004).
- John Stone, A. A., et al. . Using VMD. , (2011).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Hsin, J., Arkhipov, A., Yin, Y., Stone, J. E., Schulten, K. Using VMD: An Introductory Tutorial. Current Protocols in Bioinformatics. 24 (1), 5.7.1-5.7.48 (2008).
- Souza, P. C. T., et al. Martini 3: a general purpose force field for coarse-grained molecular dynamics. Nature Methods. 18 (4), 382-388 (2021).
- Jorgensen, W. L., Maxwell, D. S., Tirado-Rives, J. Development and Testing of the OPLS All-Atom Force Field on Conformational Energetics and Properties of Organic Liquids. Journal of the American Chemical Society. 118 (45), 11225-11236 (1996).
- Machado, M. R., et al. The SIRAH 2.0 Force Field: Altius, Fortius, Citius. Journal of Chemical Theory and Computation. 15 (4), 2719-2733 (2019).
- Huang, J., et al. CHARMM36m: an improved force field for folded and intrinsically disordered proteins. Nature Methods. 14 (1), 71-73 (2017).
- Mu, J., Liu, H., Zhang, J., Luo, R., Chen, H. F. Recent Force Field Strategies for Intrinsically Disordered Proteins. Journal of Chemical Information and Modeling. 61 (3), 1037-1047 (2021).
- Inakollu, V. S. S., Geerke, D. P., Rowley, C. N., Yu, H. Polarisable force fields: what do they add in biomolecular simulations. Current Opinion in Structural Biology. 61, 182-190 (2020).
- Ohkubo, Y. Z., et al. Accelerating Membrane Insertion of Peripheral Proteins with a Novel Membrane Mimetic Model. Biophysical Journal. 102 (9), 2130-2139 (2012).
- Hopkins, C. W., Le Grand, S., Walker, R. C., Roitberg, A. E. Long-Time-Step Molecular Dynamics through Hydrogen Mass Repartitioning. Journal of Chemical Theory and Computation. 11 (4), 1864-1874 (2015).
- Park, S., Beaven, A. H., Klauda, J. B., Im, W. How Tolerant are Membrane Simulations with Mismatch in Area per Lipid between Leaflets. Journal of Chemical Theory and Computation. 11 (7), 3466-3477 (2015).
- Park, S., Im, W., Pastor, R. W. Developing initial conditions for simulations of asymmetric membranes: a practical recommendation. Biophysical Journal. 120 (22), 5041-5059 (2021).
- Wu, E. L., et al. CHARMM-GUI Membrane Builder toward realistic biological membrane simulations. Journal of Computational Chemistry. 35 (27), 1997-2004 (2014).
- Center for Computational Research, U.a.B.. . CCR Facility Description. , (2019).
- Piggot, T. J., Allison, J. R., Sessions, R. B., Essex, J. W. On the Calculation of Acyl Chain Order Parameters from Lipid Simulations. Journal of Chemical Theory and Computation. 13 (11), 5683-5696 (2017).
- Li, J., Monje-Galvan, V. Effect of Glycone Diversity on the Interaction of Triterpenoid Saponins and Lipid Bilayers. ACS Applied Bio Materials. , (2023).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews Drug Discovery. 17 (7), 471-492 (2018).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Martínez, L., Andrade, R., Birgin, E. G., Martínez, J. M. PACKMOL: A package for building initial configurations for molecular dynamics simulations. Journal of Computational Chemistry. 30 (13), 2157-2164 (2009).
- Jewett, A. I., et al. Moltemplate: A Tool for Coarse-Grained Modeling of Complex Biological Matter and Soft Condensed Matter Physics. Journal of Molecular Biology. 433 (11), 166841 (2021).
- Jo, S., Kim, T., Iyer, V. G., Im, W. CHARMM-GUI: A web-based graphical user interface for CHARMM. Journal of Computational Chemistry. 29 (11), 1859-1865 (2008).
- Polêto, M. D., Lemkul, J. A. Integration of experimental data and use of automated fitting methods in developing protein force fields. Communications Chemistry. 5 (1), 38 (2022).
- Hynninen, A. P., Crowley, M. F. New faster CHARMM molecular dynamics engine. Journal of Computational Chemistry. 35 (5), 406-413 (2014).
- Kim, S. Issues on the Choice of a Proper Time Step in Molecular Dynamics. Physics Procedia. 53, 60-62 (2014).
- Grubmüller, H., Heller, H., Windemuth, A., Schulten, K. Generalized Verlet Algorithm for Efficient Molecular Dynamics Simulations with Long-range Interactions. Molecular Simulation. 6 (1-3), 121-142 (1991).
- Darden, T., York, D., Pedersen, L. Particle mesh Ewald: An N·log(N) method for Ewald sums in large systems. Journal of Chemical Physics. 98 (12), 10089-10092 (1993).
- . . Hepatitis C. , (2021).
- Braun, E., et al. Best Practices for Foundations in Molecular Simulations [Article v1.0]. Living Journal of Computational Molecular Science. 1 (1), 5957 (2018).
- Moradi, S., Nowroozi, A., Shahlaei, M. Shedding light on the structural properties of lipid bilayers using molecular dynamics simulation: a review study. RSC Advances. 9 (8), 4644-4658 (2019).
- Monje-Galvan, V., Klauda, J. B. Modeling Yeast Organelle Membranes and How Lipid Diversity Influences Bilayer Properties. Biochemistry. 54 (45), 6852-6861 (2015).
- Michaud-Agrawal, N., Denning, E. J., Woolf, T. B., Beckstein, O. MDAnalysis: A toolkit for the analysis of molecular dynamics simulations. Journal of Computational Chemistry. 32 (10), 2319-2327 (2011).
- Gowers, R., et al. MDAnalysis: A Python Package for the Rapid Analysis of Molecular Dynamics Simulations. SciPy. , (2016).
- McGibbon, R. o. b. e. r. t. . T., et al. MDTraj: A Modern Open Library for the Analysis of Molecular Dynamics Trajectories. Biophysical Journal. 109 (8), 1528-1532 (2015).
- Fortunato, M. E., Colina, C. M. pysimm: A python package for simulation of molecular systems. SoftwareX. 6, 7-12 (2017).
- Scherer, M. K., et al. PyEMMA 2: A Software Package for Estimation, Validation, and Analysis of Markov Models. Journal of Chemical Theory and Computation. 11 (11), 5525-5542 (2015).
- Song, W., et al. PyLipID: A Python Package for Analysis of Protein-Lipid Interactions from Molecular Dynamics Simulations. Journal of Chemical Theory and Computation. 18 (2), 1188-1201 (2022).
- Monje-Galvan, V., Klauda, J. B. Peripheral membrane proteins: Tying the knot between experiment and computation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1858 (7, Part B), 1584-1593 (2016).
- Monje-Galvan, V., Voth, G. A. Binding mechanism of the matrix domain of HIV-1 gag on lipid membranes. eLife. 9, e58621 (2020).
- Wang, B., Guo, C. Concentration-Dependent Effects of Cholesterol on the Dimerization of Amyloid-β Peptides in Lipid Bilayers. ACS Chemical Neuroscience. 13 (18), 2709-2718 (2022).

