Realistisk membranmodellering med hjälp av komplexa lipidblandningar i simuleringsstudier
Summary
Membranlipiddiversitet i struktur och sammansättning är en viktig bidragsgivare till cellulära processer och kan vara en markör för sjukdom. Molekyldynamiksimuleringar gör det möjligt för oss att studera membran och deras interaktioner med biomolekyler med atomistisk upplösning. Här tillhandahåller vi ett protokoll för att bygga, köra och analysera komplexa membransystem.
Abstract
Lipider är strukturella byggstenar i cellmembran; Lipidarter varierar mellan cellorganeller och mellan organismer. Denna variation resulterar i olika mekaniska och strukturella egenskaper i membranet som direkt påverkar de molekyler och processer som sker vid detta gränsskikt. Lipidsammansättningen är dynamisk och kan tjäna till att modulera cellsignaleringsprocesser. Beräkningsmetoder används i allt högre grad för att förutsäga interaktioner mellan biomolekyler och ge molekylära insikter till experimentella observabler. Molekyldynamik (MD) är en teknik baserad på statistisk mekanik som förutsäger atomers rörelse baserat på de krafter som verkar på dem. MD-simuleringar kan användas för att karakterisera interaktionen mellan biomolekyler. Här introducerar vi kortfattat tekniken, beskriver praktiska steg för nybörjare som är intresserade av att simulera lipiddubbellager, demonstrerar protokollet med nybörjarvänlig programvara och diskuterar alternativ, utmaningar och viktiga överväganden i processen. Speciellt betonar vi relevansen av att använda komplexa lipidblandningar för att modellera ett cellmembran av intresse för att fånga lämpliga hydrofoba och mekaniska miljöer i simulering. Vi diskuterar också några exempel där membransammansättning och egenskaper modulerar interaktionen mellan dubbellager och andra biomolekyler.
Introduction
Lipider är viktiga beståndsdelar i membran, som ger gränser för celler och möjliggör intracellulär kompartmentalisering 1,2,3. Lipider är amfifila, med en polär huvudgrupp och två hydrofoba fettsyrasvansar; Dessa självorganiserar sig i ett tvåskikt för att minimera kontakten mellan de hydrofoba kedjorna och vatten 3,4. Olika kombinationer av hydrofila huvudgrupper och hydrofoba svansar resulterar i olika klasser av lipider i biologiska membran, såsom glycerofosfolipider, sfingolipider och steroler (Figur 1)1,5,6. Glycerofosfolipider är primära byggstenar i eukaryota cellmembran som består av glycerofosfat, långkedjiga fettsyror och huvudgrupper med låg molekylvikt7. Lipidnomenklaturen baseras på skillnader i huvudgrupper; Exempel på detta är fosfatidylkolin (PC), fosfatidyletanolamin (PE), fosfatidylserin (PS), fosfatidylglycerol (PG), fosfatidylinositol (PI) eller omodifierad fosfatidinsyra (PA)5,6. När det gäller hydrofoba svansar varierar längden och mättnadsgraden, tillsammans med ryggradsstrukturen. De möjliga kombinationerna är många, vilket resulterar i tusentals lipidarter i däggdjursceller6. Förändringar i membranlipidsammansättningen leder till olika mekaniska och strukturella membranegenskaper som påverkar aktiviteten hos både integrerade membranproteiner och perifera proteiner 2,6.
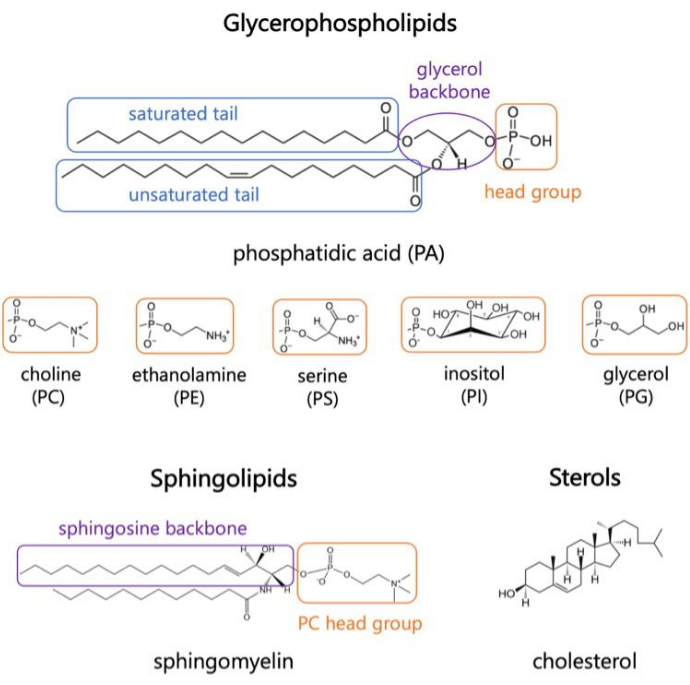
Figur 1. Representativa lipidstrukturer. Fettsyrasvansar visas i blå lådor, vanliga lipidhuvudgrupper i orange och provryggrad i lila. Klicka här för att se en större version av denna figur.
Lipider är aktiva aktörer i cellulära processer, proteinaktivering i signalkaskader och hälsosam cellhomeostas 8,9. Förändrad lipiddynamik är resultatet av infektion eller kan vara markörer för patogenes av sjukdom 10,11,12,13,14,15. Som barriärer för cellen är studiet av membranlipider och deras roll i genomträngning av små molekyler av relevans för läkemedelsleveranssystem och membranstörningsmekanismer16,17. Kemisk mångfald och olika förhållanden av lipidarter mellan organeller, vävnader och organismer ger upphov till komplex membrandynamik2. Det är därför viktigt att behålla dessa egenskaper i modelleringsstudier av lipiddubbellager, särskilt när målet med en studie är att undersöka interaktioner mellan andra biomolekyler och membranet. Vilka lipidarter som ska beaktas i en modell beror på organismen och det cellulära facket av intresse. Till exempel är PG-lipider viktiga för elektronöverföring i fotosyntetisk bateria18, medan fosforylerade inositollipider (PIP) är viktiga aktörer i plasmamembrandynamik (PM) och signalkaskader i däggdjursceller 19,20. Inuti cellen innehåller PM, endoplasmatiskt retikulum (ER), GoLgi och mitokondriella membran unika lipidöverflöd som påverkar deras funktion. Till exempel är ER navet för lipidbiogenes och transporterar kolesterol ut till PM och Golgi; den innehåller en hög lipiddiversitet med ett överflöd av PC och PE, men lågt sterolinnehåll, vilket främjar membranfluiditet21,22,23,24. Däremot innehåller PM hundratals och till och med tusentals lipidarter beroende på organismen25, den innehåller höga halter av sfingolipider och kolesterol som ger den en karakteristisk styvhet jämfört med andra membran i cellen24. Bladasymmetri bör övervägas för membran som PM, som har en yttre broschyr rik på sfingomyelin, PC och kolesterol, och en inre broschyr rik på PE, PI och PS som är viktiga för signalkaskader24. Slutligen leder lipiddiversitet också till bildandet av mikrodomäner som skiljer sig åt i packning och inre ordning, kända som lipidflottar24,26; Dessa uppvisar lateral asymmetri, antas spela viktiga roller i cellulär signalering26 och är svåra att studera på grund av deras övergående natur.
Experimentella tekniker som fluoroskopi, spektroskopi och modellmembransystem som gigantiska unilamellära vesiklar (GUV) har använts för att undersöka interaktioner mellan biomolekyler och membran. Den komplexa och dynamiska karaktären hos de inblandade komponenterna är dock svår att fånga med enbart experimentella metoder. Till exempel finns det begränsningar för avbildning av transmembrandomäner av proteiner, komplexiteten hos membran som används i sådana studier och identifiering av intermediära eller övergående tillstånd under processen av intresse27,28,29. Sedan tillkomsten av molekylär simulering av lipidmonolager och dubbellager på 1980-talet29 kan lipid-proteinsystem och deras interaktioner nu kvantifieras på molekylär nivå. Simulering av molekyldynamik (MD) är en vanlig beräkningsteknik som förutsäger partiklars rörelse baserat på deras intermolekylära krafter. En additiv interaktionspotential beskriver de bundna och icke-bundna interaktionerna mellan partiklar i systemet30. Den uppsättning parametrar som används för att modellera dessa interaktioner kallas simuleringskraftfält (FF). Dessa parametrar erhålls från ab initio-beräkningar, semi-empiriska och kvantmekaniska beräkningar och optimeras för att reproducera data från röntgen- och elektrondiffraktionsexperiment, NMR, infraröd, Raman och neutronspektroskopi, bland andra metoder31.
MD-simuleringar kan användas för att studera system på olika upplösningsnivåer32,33,34. System som syftar till att karakterisera specifika biomolekylära interaktioner, vätebindningar och andra högupplösta detaljer studeras med simuleringar av alla atomer (AA). Grovkorniga (CG) simuleringar klumpar däremot ihop atomer i större funktionella grupper för att minska beräkningskostnaderna och undersöka dynamiken i större skala33. Mellan dessa två finns UA-simuleringar (united-atoms), där väteatomer kombineras med sina respektive tunga atomer för att påskynda beräkningen33,35. MD-simuleringar är ett kraftfullt verktyg för att utforska dynamiken hos lipidmembran och deras interaktioner med andra molekyler och kan tjäna till att tillhandahålla mekanismer på molekylär nivå för processer av intresse vid membrangränssnittet. Dessutom kan MD-simuleringar användas för att begränsa experimentella mål och förutsäga makromolekylära egenskaper hos ett givet system baserat på mikroskopiska interaktioner.
I korthet, givet en uppsättning initiala koordinater, hastigheter och en uppsättning villkor som konstant temperatur och tryck, beräknas positioner och hastigheter för varje partikel genom numerisk integration av interaktionspotentialen och Newtons rörelselag. Detta upprepas iterativt och genererar därmed en simuleringsbana30. Dessa beräkningar utförs med en MD-motor; bland flera paket med öppen källkod är GROMACS36 en av de mest använda motorerna och den vi beskriver här. Den innehåller också verktyg för analys och konstruktion av initiala koordinater för system som ska simuleras37. Andra MD-motorer inkluderar NAMD38; CHARMM39 och AMBER40, som användaren kan välja efter eget gottfinnande baserat på beräkningsprestanda för ett givet system. Det är viktigt att visualisera banorna under simuleringen samt för analys och tolkning av resultaten. En mängd olika verktyg finns tillgängliga; här diskuterar vi visuell molekyldynamik (VMD) som erbjuder ett brett utbud av funktioner, inklusive tredimensionell (3-D) visualisering med expansiva ritnings- och färgläggningsmetoder, volymetrisk datavisualisering, byggande, förberedelse och analys av banor för MD-simuleringssystem och banfilmsskapande utan begränsningar för systemstorlek, om minnet är tillgängligt41,42,43.
Noggrannheten i den förutsagda dynamiken mellan systemkomponenter påverkas direkt av den FF som valts för banans utbredning. Empiriska FF-parametriseringsinsatser bedrivs av ett fåtal forskargrupper. De mest etablerade och vanliga FF för MD inkluderar CHARMM39, AMBER 40, Martini44, OPLS 45 och SIRAH 46. Kraftfältet47 för additiv CHARMM36 (C36) med alla atomer används i stor utsträckning för AA MD av membransystem eftersom det exakt reproducerar experimentella strukturdata. Den utvecklades ursprungligen av CHARMM-communityt, och den är kompatibel med flera MD-motorer som GROMACS och NAMD. Trots förbättringar i vanliga FF finns det ett kontinuerligt arbete med att förbättra parameteruppsättningarna för att möjliggöra förutsägelser som nära reproducerar experimentella observerbara värden, drivet av intressen i vissa studiesystem48,49.
En utmaning vid simulering av lipidmembran är att bestämma längden på simuleringsbanan. Detta beror till stor del på de mätvärden som ska analyseras och den process som man vill karakterisera. Vanligtvis kräver komplexa lipidblandningar längre tid för att nå jämvikt, eftersom fler arter måste ha tillräckligt med tid för att diffundera på membranplanet och nå en stabil lateral organisation. En simulering sägs vara i jämvikt när egenskapen av intresse har nått en platå och fluktuerar kring ett konstant värde. Det är vanligt att erhålla minst 100-200 ns jämviktsbana för att utföra lämplig statistisk analys av egenskaper och interaktioner av intresse. Det är vanligt att köra membransimuleringar mellan 200-500 ns, beroende på lipidblandningens komplexitet och frågeställning. Protein-lipidinteraktioner kräver vanligtvis längre simuleringstider, mellan 500-2000 ns. Några metoder för att påskynda provtagning och observerbar dynamik med membransystem är: (i) den mycket rörliga membranmimetiska (HMMM) modellen, som ersätter ändkol av lipider i membranet med organiskt lösningsmedel för att påskynda provtagning50; och (ii) vätemassepartitionering (HMR), som kombinerar en bråkdel av massan av tunga atomer i ett system med massan av väteatomer för att möjliggöra användning av ett större simuleringstidssteg51.
Följande protokoll beskriver en nybörjarvänlig metod för att bygga, köra och analysera realistiska membranmodeller med hjälp av AA MD. Med tanke på MD-simuleringarnas karaktär måste flera trajektorier köras för att ta hänsyn till reproducerbarhet och korrekt statistisk analys av resultaten. Det är aktuell praxis att köra minst tre repliker per system av intresse. När lipidarterna har valts ut för organismen och processen av intresse, beskrivs och sammanfattas grundläggande steg för att bygga, köra och analysera en simuleringsbana för ett membransystem i figur 2.
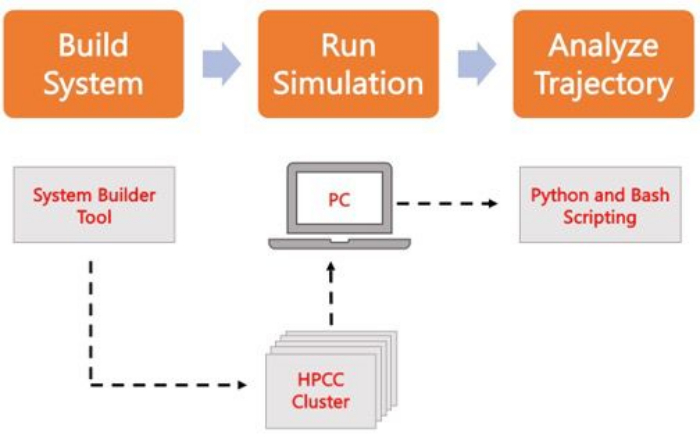
Figur 2. Schematisk för att köra MD-simuleringar. Orange rutor motsvarar de tre huvudstegen som beskrivs i protokollet. Nedanför finns arbetsflödet för simuleringsprocessen. Under systeminstallationen byggs systemet som innehåller de initiala koordinaterna för ett löst membransystem med en systemingångsgenerator som CHARMM-GUI Membrane Builder. Efter att ha överfört indatafilerna till ett högpresterande datorkluster sprids simuleringsbanan med hjälp av en MD-motor, till exempel GROMACS. Bananalys kan göras på datorklustret eller en lokal arbetsstation tillsammans med visualisering. Analysen utförs sedan antingen med hjälp av paket med inbyggd analyskod som GROMACS och VMD, eller med hjälp av Bash-skript eller olika Python-bibliotek. Klicka här för att se en större version av denna figur.
Protocol
Representative Results
Discussion
Experimentella tekniker kan visualisera biomolekyler med hög upplösning med hjälp av kryoelektronmikroskopi (cryo-EM)58, fluorescenstekniker och atomkraftsmikroskopi (AFM)59. Det är dock utmanande att fånga samspelet och dynamiken i molekylära interaktioner som ligger till grund för biologiska vägar, sjukdomspatogenes och terapeutisk leverans på atom- eller aminosyranivå. Här diskuterades MD-simuleringars möjligheter att studera lipidmembran och de viktigaste st…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Författarna tackar Jinhui Li och Ricardo X. Ramirez för deras simuleringsbanor och diskussioner under skrivandet av detta manuskript. OC stöddes av University at Buffalo Presidential Fellowship och National Institute of Health’s Initiative for Maximizing Student Development Training Grant 1T32GM144920-01 som tilldelades Margarita L. Dubocovich (PI).
Materials
| Anaconda3 | Anaconda Inc (Python & related libraries) | N/A | |
| CHARMM-GUI.org | Im lab, Lehigh University | N/A | |
| GROMACS | GROMACS development team | N/A | |
| Linux HPC Cluster | UB CCR | N/A | |
| MATLAB | MathWorks | N/A | |
| VMD | Theoretical and Computational Biophysics Group | N/A |
References
- Vanni, S., Riccardi, L., Palermo, G., De Vivo, M. Structure and Dynamics of the Acyl Chains in the Membrane Trafficking and Enzymatic Processing of Lipids. Accounts of Chemical Research. 52 (11), 3087-3096 (2019).
- Harayama, T., Riezman, H. Understanding the diversity of membrane lipid composition. Nature Reviews Molecular Cell Biology. 19 (5), 281-296 (2018).
- Tanaka, M., Edward, H. .. . E. g. e. l. m. a. n. . Comprehensive Biophysics. , 261-272 (2012).
- Bruce Alberts, A. J., Julian Lewis, , Martin Raff, , Keith Roberts, , Peter Walter, . Molecular Biology of the Cell. , (2002).
- Watson, H. Biological membranes. Essays in Biochemistry. 59, 43-69 (2015).
- Coskun, &. #. 2. 2. 0. ;., Simons, K. Cell Membranes: The Lipid Perspective. Structure. 19 (11), 1543-1548 (2011).
- Douglas G, H. a. y. e. s., Daniel, K. Y., Solaiman, D., Richard, . Biobased Surfactants (Second Edition) eds. , 515-529 (2019).
- González-Rubio, P., Gautier, R., Etchebest, C., Fuchs, P. F. J. Amphipathic-Lipid-Packing-Sensor interactions with lipids assessed by atomistic molecular dynamics. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808, 2119-2127 (2011).
- Halbleib, K., et al. Activation of the Unfolded Protein Response by Lipid Bilayer Stress. Molecular Cell. 67, 673-684 (2017).
- Andreasen, M., Lorenzen, N., Otzen, D. Interactions between misfolded protein oligomers and membranes: A central topic in neurodegenerative diseases. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1848 (9), 1897-1907 (2015).
- Calianese, D. C., Birge, R. B. Biology of phosphatidylserine (PS): basic physiology and implications in immunology, infectious disease, and cancer. Cell Commununication and Signaling. 18 (1), 41 (2020).
- Nieto-Garai, J. A., Contreras, F. X., Arboleya, A., Lorizate, M. Role of Protein-Lipid Interactions in Viral Entry. Advanced Biology. 6, 2101264 (2022).
- Mazzon, M., Mercer, J. Lipid interactions during virus entry and infection. Cell Microbiology. 16, 1493-1502 (2014).
- Colombelli, C., Aoun, M., Tiranti, V. Defective lipid metabolism in neurodegeneration with brain iron accumulation (NBIA) syndromes: not only a matter of iron. Journal of Inherited Metabolic Disease. 38 (1), 123-136 (2015).
- Saini-Chohan, H. K., Mitchell, R. W., Vaz, F. M., Zelinski, T., Hatch, G. M. Delineating the role of alterations in lipid metabolism to the pathogenesis of inherited skeletal and cardiac muscle disorders: Thematic Review Series: Genetics of Human Lipid Diseases. Journal of Lipid Research. 53 (1), 4-27 (2012).
- Martinotti, C., Ruiz-Perez, L., Deplazes, E., Mancera, R. L. Molecular Dynamics Simulation of Small Molecules Interacting with Biological Membranes. ChemPhysChem. 21 (14), 1486-1514 (2020).
- Li, J., Kalyanram, P., Rozati, S., Monje-Galvan, V., Gupta, A. Interaction of Cyanine-D112 with Binary Lipid Mixtures: Molecular Dynamics Simulation and Differential Scanning Calorimetry Study. ACS Omega. 7 (11), 9765-9774 (2022).
- Nagy, L., et al. Protein/Lipid Interaction in the Bacterial Photosynthetic Reaction Center: Phosphatidylcholine and Phosphatidylglycerol Modify the Free Energy Levels of the Quinones. Biochemistry. 43 (40), 12913-12923 (2004).
- Ramirez, R. X., Campbell, O., Pradhan, A. J., Atilla-Gokcumen, G. E., Monje-Galvan, V. Modeling the molecular fingerprint of protein-lipid interactions of MLKL on complex bilayers. Frontiers in Chemistry. 10, (2023).
- Dondelinger, Y., et al. MLKL Compromises Plasma Membrane Integrity by Binding to Phosphatidylinositol Phosphates. Cell Reports. 7 (4), 971-981 (2014).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews Molecular Cell Biology. 9 (2), 112-124 (2008).
- van Meer, G., de Kroon, A. I. P. M. Lipid map of the mammalian cell. Journal of Cell Science. 124 (1), 5 (2011).
- Lee, H. R., Lee, G. Y., You, D. G., Kim, H. K., Young, D. Y. Hepatitis C virus p7 induces membrane permeabilization by interacting with phosphatidylserine. International Journal of Molecular Sciences. 21 (3), 897 (2020).
- Casares, D., Escribá, P. V., Rosselló, C. A. Membrane Lipid Composition: Effect on Membrane and Organelle Structure, Function and Compartmentalization and Therapeutic Avenues. International Journal of Molecular Sciences. 20 (9), 2167 (2019).
- Marrink, S. J., et al. Computational Modeling of Realistic Cell Membranes. Chemical Reviews. 119 (9), 6184-6226 (2019).
- Janmey, P. A., Kinnunen, P. K. J. Biophysical properties of lipids and dynamic membranes. Trends in Cell Biology. 16 (10), 538-546 (2006).
- Brémaud, E., Favard, C., Muriaux, D. Deciphering the Assembly of Enveloped Viruses Using Model Lipid Membranes. Membranes. 12, 441 (2022).
- Campbell, O., Monje-Galvan, V. Protein-driven membrane remodeling: Molecular perspectives from Flaviviridae infections. Biophysical Journal. 122 (11), 1890-1899 (2022).
- Loschwitz, J., Olubiyi, O. O., Hub, J. S., Strodel, B., Poojari, C. S. Computer simulations of protein-membrane systems. Progress in molecular biology and translational science. 170, 273-403 (2020).
- Shell, M. S., Scott Shell, M. . Thermodynamics and Statistical Mechanics: An Integrated ApproachCambridge Series in Chemical Engineering. , 21-49 (2015).
- Yang, J., et al. Molecular Dynamic Simulation of Ni-Al Alloy-H2O Reactions Using the ReaxFF Reactive Force Field. ACS Omega. 8 (11), 9807-9814 (2023).
- Ingólfsson, H. I., Arnarez, C., Periole, X., Marrink, S. J. Computational ‘microscopy’ of cellular membranes. Journal of Cell Science. 129 (2), 257-268 (2016).
- Klauda, J. B. Perspective: Computational modeling of accurate cellular membranes with molecular resolution. The Journal of Chemical Physics. 149 (22), 220901 (2018).
- Chavent, M., Duncan, A. L., Sansom, M. S. P. Molecular dynamics simulations of membrane proteins and their interactions: from nanoscale to mesoscale. Current Opinion in Structural Biology. 40, 8-16 (2016).
- Khakbaz, P., Monje-Galvan, V., Zhuang, X., Klauda, J. B., Otto Geiger, . Biogenesis of Fatty Acids, Lipids and Membranes. , 1-19 (2017).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1, 19-25 (2015).
- Lemkul, J. A. From Proteins to Perturbed Hamiltonians: A Suite of Tutorials for the GROMACS-2018 Molecular Simulation Package. Living Journal of Computational Molecular Science. 1 (1), 5068 (2018).
- Phillips, J. C., et al. Scalable molecular dynamics on CPU and GPU architectures with NAMD. The Journal of Chemical Physics. 153 (4), 044130 (2020).
- Klauda, J. B., et al. Update of the CHARMM All-Atom Additive Force Field for Lipids: Validation on Six Lipid Types. The Journal of Physical Chemistry B. 114 (23), 7830-7843 (2010).
- Wang, J., Wolf, R. M., Caldwell, J. W., Kollman, P. A., Case, D. A. Development and testing of a general amber force field. Journal of Computational Chemistry. 25 (9), 1157-1174 (2004).
- John Stone, A. A., et al. . Using VMD. , (2011).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Hsin, J., Arkhipov, A., Yin, Y., Stone, J. E., Schulten, K. Using VMD: An Introductory Tutorial. Current Protocols in Bioinformatics. 24 (1), 5.7.1-5.7.48 (2008).
- Souza, P. C. T., et al. Martini 3: a general purpose force field for coarse-grained molecular dynamics. Nature Methods. 18 (4), 382-388 (2021).
- Jorgensen, W. L., Maxwell, D. S., Tirado-Rives, J. Development and Testing of the OPLS All-Atom Force Field on Conformational Energetics and Properties of Organic Liquids. Journal of the American Chemical Society. 118 (45), 11225-11236 (1996).
- Machado, M. R., et al. The SIRAH 2.0 Force Field: Altius, Fortius, Citius. Journal of Chemical Theory and Computation. 15 (4), 2719-2733 (2019).
- Huang, J., et al. CHARMM36m: an improved force field for folded and intrinsically disordered proteins. Nature Methods. 14 (1), 71-73 (2017).
- Mu, J., Liu, H., Zhang, J., Luo, R., Chen, H. F. Recent Force Field Strategies for Intrinsically Disordered Proteins. Journal of Chemical Information and Modeling. 61 (3), 1037-1047 (2021).
- Inakollu, V. S. S., Geerke, D. P., Rowley, C. N., Yu, H. Polarisable force fields: what do they add in biomolecular simulations. Current Opinion in Structural Biology. 61, 182-190 (2020).
- Ohkubo, Y. Z., et al. Accelerating Membrane Insertion of Peripheral Proteins with a Novel Membrane Mimetic Model. Biophysical Journal. 102 (9), 2130-2139 (2012).
- Hopkins, C. W., Le Grand, S., Walker, R. C., Roitberg, A. E. Long-Time-Step Molecular Dynamics through Hydrogen Mass Repartitioning. Journal of Chemical Theory and Computation. 11 (4), 1864-1874 (2015).
- Park, S., Beaven, A. H., Klauda, J. B., Im, W. How Tolerant are Membrane Simulations with Mismatch in Area per Lipid between Leaflets. Journal of Chemical Theory and Computation. 11 (7), 3466-3477 (2015).
- Park, S., Im, W., Pastor, R. W. Developing initial conditions for simulations of asymmetric membranes: a practical recommendation. Biophysical Journal. 120 (22), 5041-5059 (2021).
- Wu, E. L., et al. CHARMM-GUI Membrane Builder toward realistic biological membrane simulations. Journal of Computational Chemistry. 35 (27), 1997-2004 (2014).
- Center for Computational Research, U.a.B.. . CCR Facility Description. , (2019).
- Piggot, T. J., Allison, J. R., Sessions, R. B., Essex, J. W. On the Calculation of Acyl Chain Order Parameters from Lipid Simulations. Journal of Chemical Theory and Computation. 13 (11), 5683-5696 (2017).
- Li, J., Monje-Galvan, V. Effect of Glycone Diversity on the Interaction of Triterpenoid Saponins and Lipid Bilayers. ACS Applied Bio Materials. , (2023).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews Drug Discovery. 17 (7), 471-492 (2018).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Martínez, L., Andrade, R., Birgin, E. G., Martínez, J. M. PACKMOL: A package for building initial configurations for molecular dynamics simulations. Journal of Computational Chemistry. 30 (13), 2157-2164 (2009).
- Jewett, A. I., et al. Moltemplate: A Tool for Coarse-Grained Modeling of Complex Biological Matter and Soft Condensed Matter Physics. Journal of Molecular Biology. 433 (11), 166841 (2021).
- Jo, S., Kim, T., Iyer, V. G., Im, W. CHARMM-GUI: A web-based graphical user interface for CHARMM. Journal of Computational Chemistry. 29 (11), 1859-1865 (2008).
- Polêto, M. D., Lemkul, J. A. Integration of experimental data and use of automated fitting methods in developing protein force fields. Communications Chemistry. 5 (1), 38 (2022).
- Hynninen, A. P., Crowley, M. F. New faster CHARMM molecular dynamics engine. Journal of Computational Chemistry. 35 (5), 406-413 (2014).
- Kim, S. Issues on the Choice of a Proper Time Step in Molecular Dynamics. Physics Procedia. 53, 60-62 (2014).
- Grubmüller, H., Heller, H., Windemuth, A., Schulten, K. Generalized Verlet Algorithm for Efficient Molecular Dynamics Simulations with Long-range Interactions. Molecular Simulation. 6 (1-3), 121-142 (1991).
- Darden, T., York, D., Pedersen, L. Particle mesh Ewald: An N·log(N) method for Ewald sums in large systems. Journal of Chemical Physics. 98 (12), 10089-10092 (1993).
- . . Hepatitis C. , (2021).
- Braun, E., et al. Best Practices for Foundations in Molecular Simulations [Article v1.0]. Living Journal of Computational Molecular Science. 1 (1), 5957 (2018).
- Moradi, S., Nowroozi, A., Shahlaei, M. Shedding light on the structural properties of lipid bilayers using molecular dynamics simulation: a review study. RSC Advances. 9 (8), 4644-4658 (2019).
- Monje-Galvan, V., Klauda, J. B. Modeling Yeast Organelle Membranes and How Lipid Diversity Influences Bilayer Properties. Biochemistry. 54 (45), 6852-6861 (2015).
- Michaud-Agrawal, N., Denning, E. J., Woolf, T. B., Beckstein, O. MDAnalysis: A toolkit for the analysis of molecular dynamics simulations. Journal of Computational Chemistry. 32 (10), 2319-2327 (2011).
- Gowers, R., et al. MDAnalysis: A Python Package for the Rapid Analysis of Molecular Dynamics Simulations. SciPy. , (2016).
- McGibbon, R. o. b. e. r. t. . T., et al. MDTraj: A Modern Open Library for the Analysis of Molecular Dynamics Trajectories. Biophysical Journal. 109 (8), 1528-1532 (2015).
- Fortunato, M. E., Colina, C. M. pysimm: A python package for simulation of molecular systems. SoftwareX. 6, 7-12 (2017).
- Scherer, M. K., et al. PyEMMA 2: A Software Package for Estimation, Validation, and Analysis of Markov Models. Journal of Chemical Theory and Computation. 11 (11), 5525-5542 (2015).
- Song, W., et al. PyLipID: A Python Package for Analysis of Protein-Lipid Interactions from Molecular Dynamics Simulations. Journal of Chemical Theory and Computation. 18 (2), 1188-1201 (2022).
- Monje-Galvan, V., Klauda, J. B. Peripheral membrane proteins: Tying the knot between experiment and computation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1858 (7, Part B), 1584-1593 (2016).
- Monje-Galvan, V., Voth, G. A. Binding mechanism of the matrix domain of HIV-1 gag on lipid membranes. eLife. 9, e58621 (2020).
- Wang, B., Guo, C. Concentration-Dependent Effects of Cholesterol on the Dimerization of Amyloid-β Peptides in Lipid Bilayers. ACS Chemical Neuroscience. 13 (18), 2709-2718 (2022).

