Analyse av proteinkompleksdannelse ved mikromolære konsentrasjoner ved kobling av mikrofluidikk med massefotometri
Summary
Denne protokollen kombinerer massefotometri med et nytt mikrofluidikksystem for å undersøke protein-protein-interaksjoner med lav affinitet. Denne tilnærmingen er basert på rask fortynning av høykonsentrerte komplekser i løsning, noe som muliggjør målinger med lav affinitet og utvider anvendeligheten av massefotometri.
Abstract
Massefotometri er en allsidig massemålingsteknologi som muliggjør studier av biomolekylære interaksjoner og kompleks dannelse i løsning uten etiketter. Massefotometri er generelt egnet til å analysere prøver i konsentrasjonsområdet 100 pM-100 nM. I mange biologiske systemer er det imidlertid nødvendig å måle mer konsentrerte prøver for å studere interaksjoner med lav affinitet eller forbigående affinitet. Her demonstrerer vi en metode som effektivt utvider rekkevidden av prøvekonsentrasjoner som kan analyseres ved massefotometri fra nanomolar til titalls mikromolar.
I denne protokollen kombineres massefotometri med et nytt mikrofluidikksystem for å undersøke dannelsen av proteinkomplekser i oppløsning i det mikromolære konsentrasjonsområdet. Med mikrofluidikksystemet kan brukerne opprettholde en prøve ved en ønsket høyere konsentrasjon etterfulgt av fortynning til nanomolærområdet – flere millisekunder før massefotometrimålingen. På grunn av fortynningshastigheten oppnås data før prøvens likevekt har forskjøvet seg (dvs. dissosiasjon av komplekset).
Teknikken brukes til å måle interaksjoner mellom et immunoglobulin G (IgG) antistoff og den neonatale Fc-reseptoren, som viser dannelsen av høyordenskomplekser som ikke var kvantifiserbare med statiske massefotometrimålinger.
Avslutningsvis gjør kombinasjonen av massefotometri og mikrofluidikk det mulig å karakterisere prøver i det mikromolære konsentrasjonsområdet og er dyktig i å måle biomolekylære interaksjoner med svakere affiniteter. Disse evnene kan brukes i en rekke sammenhenger – inkludert utvikling og design av bioterapi – noe som muliggjør grundig karakterisering av ulike protein-protein-interaksjoner.
Introduction
Protein-protein-interaksjoner understreker de fleste cellulære funksjoner, fra immunregulering til DNA-replikasjon og oversettelse. Som et resultat er det et grunnleggende behov i hele biovitenskapen for å undersøke et stort spekter av interaksjoner på tvers av ulike heterogene komplekser som ofte dannes. Imidlertid er deteksjon, karakterisering og kvantifisering ofte utfordrende, spesielt for interaksjoner med lav affinitet1.
Immunutfellingsanalyser brukes ofte til å oppdage interaksjoner med høy affinitet, men for interaksjoner med lav affinitet og forbigående er deteksjon stort sett ikke mulig2. Fluorescensteknikker kan også brukes, men krever potensielt forstyrrende tillegg av fluorescerende etiketter2. Cryo-EM kan gi et strukturelt øyeblikksbilde og en ensemblesning av proteinkompleksene dannet med høy romlig oppløsning, men krever også vanligvis arbeid ved konsentrasjoner som er for lave for avbildning av lavaffinitetsinteraksjoner. Cryo-EM gir også utfordringer knyttet til kostnad, tilgjengelighet, prøvepreparering og analysetid3.
I tillegg har overflateplasmonresonans (SPR) blitt en populær måte å kvantifisere protein-protein-interaksjoner på, selv om det krever proteinimmobilisering, noe som kan påvirke bindingslikevekten og resultere i variable frekvenser, og dermed redusere målenøyaktigheten 4,5. Det innebærer også flere analysetrinn før datainnsamling og analyse6.
Massefotometri er en enkeltmolekylteknikk som har blitt brukt til å analysere protein-protein-interaksjoner 5,6,7. Det fungerer ved å måle massen av enkeltmolekyler eller komplekser basert på lyset de sprer når de lander på overflaten av et glassdeksel8. Massefotometrimålinger har blitt brukt til å kvantifisere bindingsaffiniteter fra den relative overflod av bindingspartnere og kompleksene de danner5. Likevel, som andre enkeltmolekylteknikker, bør konsentrasjonen av prøven som skal måles, typisk være mindre enn 100 nM. Hvis konsentrasjonen er høyere, vil molekylene som lander på glassoverflaten overlappe romlig, noe som resulterer i dårlig datakvalitet7. Følgelig kan svakere interaksjoner (KD ~ mikromolarer), som dissosieres ved disse lavere konsentrasjonene, ikke måles pålitelig siden det ikke er mulig å observere den nødvendige blandingen av ubundne og bundne arter5.
Her beskriver vi en tilnærming som overvinner denne begrensningen basert på en ny koblet mikrofluidikk massefotometri enhet. Spesielt brukes et mikrofluidikksystem i kombinasjon med massefotometeret for effektivt å utvide rekkevidden av interaksjoner som kan kvantifiseres ved massefotometri. Mikrofluidikk har vist seg å tilby en rekke muligheter for å undersøke protein-protein-interaksjoner, inkludert rask fortynning for å oppdage svake interaksjoner 1,9. Systemet beskrevet her fungerer ved raskt å fortynne prøven opptil 10 000 ganger på en mikrofluidisk chip og umiddelbart strømme den over observasjonsområdet til brikken, slik at massefotometrimålingen kan starte innen 50 ms fra når molekylene begynte fortynningsprosessen10. Fortynningen skjer når prøven og bufferen kombineres i en omvendt Tesla-ventilblander på brikken, med de relative strømningshastighetene til de to løsningene som bestemmer mengden fortynning som oppstår (se protokolltrinn 8). Strømningshastigheten kan kontrolleres med programvaren for mikrofluidisk kontroll. Endring av strømningshastigheten kan endre den relative populasjonen av arten, da det kan påvirke antall landingshendelser på overflaten av glasset, som er det som måles av massefotometeret.
Prosessens hastighet er rask nok til at målingen kan fullføres før integriteten til samhandlingen har blitt forstyrret (for ytterligere detaljer, se også diskusjonen). Dette kan forstås gjennom en kort titt på teorien om førsteordens reaksjoner, hvor 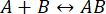 . Forward (assosiasjons) rate konstant er kf, bakover (dissosiasjon) rate konstant er kb, og likevektsdissosiasjonskonstanten (KD) er definert som
. Forward (assosiasjons) rate konstant er kf, bakover (dissosiasjon) rate konstant er kb, og likevektsdissosiasjonskonstanten (KD) er definert som
KD = kb / kf
For proteinbinding er kfgenerelt begrenset av reaktantens diffusjon11 og er derfor begrenset til området 106-10 7 M-1 · s-1. Fordi rekkevidden av er begrenset, vil en reaksjon med lav affinitet (KD ~ mikromolarer) ha kb≈ 1 s-1. Det vil si kb = kf · KD = (106 M-1·s-1) (10-6 M) = 1 s-1, med kompleksets halveringstid rundt 0,7 s11,12.
Vårt eksempelsystem er binding av IgG monoklonalt antistoff trastuzumab til løselig domene av IgG neonatal Fc-reseptor (FcRn), som er kjente vekselvirkende partnere13. Tidligere publiserte data oppnådd ved bruk av konvensjonell massefotometri alene (dvs. med manuell fortynning av prøver) viste at proteinene danner flere arter. FcRn-monomerer, FcRn-dimerer og ubundet IgG var tydelig synlige, mens IgG-FcRn-komplekser (ved forholdet 1:1 og 1:2) også ble detektert (ved pH 5,0), men bare med svært lav overflod5. Denne observasjonen stiller spørsmål ved om dannelsen av IgG-FcRn-komplekser kan oppdages tydeligere hvis den måles i en høyere konsentrasjon. Faktisk ga kombinering av massefotometri med en kombinert rask fortynningstilnærming beskrevet her mer robust bevis på kompleksdannelse ved en økning i deres målte partikler.
Massefotometri- og mikrofluidikkprotokollen beskrevet her gjør det mulig å karakterisere dannelsen av komplekser med en K, D opp til mikromolærområdet. En empirisk bestemmelse av KD vil kreve ytterligere forbedringer på strømningssensorens nøyaktighet, pumpestabilitet, chip-to-chip-variasjoner og måleplassering inne i observasjonsvinduet, da alle disse faktorene vil påvirke tiden fra prøven fortynnes til den måles.
Den samme tilnærmingen kan brukes til å undersøke binding blant oppløselige proteiner, forutsatt at de har forskjellige molekylvekter (separert med minst 25 kDa) som faller inn i området som er egnet for analyse med et massefotometer (30 kDa til 6 MDa). Innsikten som er oppnådd, kan være nyttig for studier i en rekke sammenhenger – fra å få en mekanistisk forståelse av cellulære funksjoner til utforming av nye bioterapeutiske stoffer.
Protocol
Representative Results
Discussion
Protokollen som er skissert her, gir en metode for å oppdage og kvantifisere protein-protein-interaksjoner med lav affinitet. Den bruker et massefotometer koblet til et hurtigfortynnende mikrofluidikksystem. Massefotometri er et etikettfritt, bioanalytisk verktøy som pålitelig kan måle molekylær masse i løsning for biomolekyler16, for de innenfor området 30 kDa til 6 MDa. Siden massefotometri er en enkeltmolekylteknikk som analyserer prøver en etter en, er den generelt begrenset til prøver i konsentrasjonsområdet 100 pM-100 nM. Over dette området vil molekylene som lander på glassoverflaten overlappe romlig, noe som resulterer i dårlig datakvalitet; Under dette området oppnås for lite data til å gjøre robust analyse7. En viktig konsekvens er at det kan begrense undersøkelsen av proteininteraksjoner til de som danner en blanding av bundne og ubundne arter innenfor dette området.
Her detaljerte vi en trinnvis protokoll for bruk av et hurtigfortynnende mikrofluidikksystem for effektivt å utvide rekkevidden av prøvekonsentrasjoner som er mottagelige for massefotometri. Ved å fortynne prøven på den mikrofluidiske brikken og deretter strømme den over detektorobservasjonsvinduet innen 50 ms, fanger systemet kompleksene som er tilstede i den ufortynnede prøven før interaksjonslikevekten skifter. Prøven leveres kontinuerlig til detektoren under individuelle målinger. Under disse forholdene vil 95% av komplekset forbli intakt når prøven måles, selv for lavaffinitetsinteraksjoner – med en KD i størrelsesorden mikromolarer og dissosiasjonshastigheter så raskt som 1 s-1.
Dette kan beregnes som følger: For en reaksjon 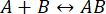 med en terminrente kfog bakoverkurs kb,
med en terminrente kfog bakoverkurs kb,
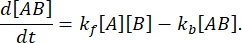
Ved likevekt forblir konsentrasjonene av alle tre artene (A, B og komplekset AB) konstant, så 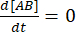 og
og 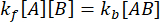 . Under den konservative antagelsen om at forstyrrelsen (fortynning, i dette tilfellet) kan føre til at komplekset dissosierer, men den fremre (assosiasjons) reaksjonen ikke fortsetter, kan begrepet kf [A] [B] behandles som ubetydelig, og følgende forenkling kan gjøres:
. Under den konservative antagelsen om at forstyrrelsen (fortynning, i dette tilfellet) kan føre til at komplekset dissosierer, men den fremre (assosiasjons) reaksjonen ikke fortsetter, kan begrepet kf [A] [B] behandles som ubetydelig, og følgende forenkling kan gjøres:
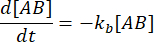
Integrering gir følgende uttrykk for konsentrasjonen av kompleks på tid etter forstyrrelse av likevekt:
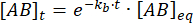
Fraksjonen av komplekset som forblir bundet på tidspunkt t etter forstyrrelsen av likevekt er således:
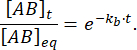
Ved = 50 ms, for en reaksjon med kb≈ 1 s-1, er fraksjonen bundet 0,95 eller 95%11,12.
Her og tidligere5 ble massefotometri brukt for å undersøke bindingen av det monoklonale antistoffet IgG trastuzumab til det løselige domenet til FcRn. De to bindingspartnerne er rapportert å binde seg med nanomolar affinitet ved sur pH17. Massefotometri ble brukt til å kvalitativt vurdere overflod av kompleksene som ble dannet mens bindingspartnerne var ved pH 5,0, og prøvene ble raskt fortynnet gjennom et ekstra mikrofluidisk system. Prosedyren ble optimalisert for den spesielle protein-protein-interaksjonen basert på tidligere rapporterte resultater5. Den samme prosedyren kan brukes til å studere andre interaksjoner, forutsatt at brukerne har forkunnskaper eller optimaliserer de eksperimentelle forholdene for det aktuelle systemet, for eksempel hvilke buffere som skal brukes, den opprinnelige proteinkonsentrasjonen, forventet støkiometri og mengden inkubasjon som trengs for å tillate samspillet å nå en likevekt.
Når IgG-FcRn-blandingen ble fortynnet manuelt, var det vanskelig å påvise tilstedeværelse av IgG-FcRn-komplekser, selv om disse proteinene er kjent for å interagere5. Denne artikkelen viser at den raske fortynningsmetoden resulterer i en betydelig økt mengde av disse kompleksene. For den samme prøven, når rask fortynning ble brukt, ble både 1: 1 FcRn-IgG-komplekser og 2: 1 FcRn-IgG-komplekser tydelig observert. Disse forskjellene i kompleks dannelse demonstrerer viktigheten av å studere biomolekylære interaksjonssystemer over et bredt spekter av konsentrasjoner.
I tillegg viser disse resultatene også at det er enkelt å bruke mikrofluidikk med enkeltmolekylanalyse for å fange svake interaksjoner – fylle et betydelig gap i metoden. Kombinasjonen av hurtigfortynnende mikrofluidikk med massefotometri gir attraktive fordeler på grunn av fordelene med massefotometri som analytisk teknikk. Det vil si at massefotometri ikke krever etiketter, innebærer minimal prøvepreparering, og målingene gjøres i løsning. For denne protokollen er en annen viktig fordel ved massefotometri dens evne til å skille og kvantifisere alle artene som dannes (forutsatt at de har en distinkt masse på >30 kDa). Dette er i motsetning til for eksempel SPR, som kan måle bindings- og ubindingshastigheter, men ikke lett kan gi støkiometriinformasjon8.
For denne protokollen, så vel som massefotometrieksperimenter mer generelt, er flere hensyn nyttige. For det første bør den endelige proteinkonsentrasjonen ligge innenfor grensen for hva massefotometri kan måle (100 pM-100 nM). Startinkubasjonskonsentrasjonen bør også være innenfor området for det mikrofluidiske systemet (opptil 90 μM) og teoretiseres til å være over den faktiske KD av interaksjonen10. Det anbefalte utgangspunktet er et blandingsforhold på 1:1 mellom de vekselvirkende artene ved μM-konsentrasjon. Forholdet kan da varieres til 1:2, 1:5, eller, som i tilfellet med denne interaksjonen, 1:10. Hvis det ikke er noen tidligere informasjon om proteininteraksjonene, må brukeren optimalisere eksperimentet, med utgangspunkt i en høy konsentrasjon (anbefalt 20 μM) for hver partner for å avgjøre om affiniteten til komponentene er innenfor konsentrasjonsområdet opprettholdt av metoden presentert (dvs. komplekser dannes). Optimalisering kan også innebære å velge andre bufferbetingelser for å fremme interaksjoner eller titrering av en av interaksjonskomponentene for å bestemme riktig blandingsforhold. Når disse er bestemt, er det mulig å optimalisere konsentrasjoner og strømmer for å tillate optimale forhold for studien og metoden, for eksempel å redusere konsentrasjonene for å muliggjøre bedre toppoppløsning.
For det andre, for å kunne replikere dette eksperimentet, bør urenheter minimeres. Vanlige kilder til urenheter som er kjent for å påvirke massefotometrimålinger negativt, inkluderer andre proteiner eller cellulært rusk som forblir etter rensing, ufiltrerte buffere, micelledannende vaskemidler (hvis de er tilstede i for høy konsentrasjon) og buffere som inneholder høye konsentrasjoner av salt, glyserol eller andre komponenter. Som omtalt i protokollen ovenfor, bør bobler i mikrofluidikksystemet fjernes. Bobler kan dannes i slangesystemet eller hvis prøvene har høy overflatespenning og er utsatt for skumdannelse. Det kan også dannes bobler i nedsenkningsoljen, som kan påvises fra fokusringen (figur 3). Hvis bobler ikke kan fjernes ved hjelp av trinnene beskrevet i protokollen, er en annen løsning å degas prøven ved hjelp av en tørketrommel og en vakuumpumpe, slik at prøven blir redusert i noen minutter. Vortexing eller risting av høykonsentrerte proteinløsninger anbefales ikke, da disse virkningene kan fremme bobledannelse.
Mens måling av en spesifikk protein-protein-interaksjon er demonstrert her, kan den samme protokollen brukes på andre protein-protein-interaksjonssystemer uten signifikant modifikasjon. En videre fremtidig retning av denne protokollen vil være å bruke målingene til å beregneKD-verdier for kompleksene som er identifisert, som det har blitt beskrevet andre steder i sammenheng med massefotometri 5,7. Mens de tidligere studiene brukte data fra eksperimenter som involverte manuell fortynning og sterkere interaksjoner, kunne analyseprinsippet lett brukes i denne sammenhengen – forutsatt at ytterligere forbedringer i den mikrofluidiske enheten implementeres (for eksempel økt strømningssensornøyaktighet og pumpestabilitet).
Utover protein-protein-interaksjoner vil det sannsynligvis være bredere anvendelser for kombinert massefotometri og hurtigfortynning mikrofluidikk tilnærming. Massefotometri kan brukes til å vurdere prøvens renhet, aggregering og homogenitet18,19; studere protein oligomerisering20, makromolekylær montering21 eller polymerisering22; og på andre områder. Massefotometrianalyse strekker seg også utover proteiner; Det har blitt brukt til å undersøke interaksjoner mellom nukleinsyrer og proteiner23, virale partikler24 og nanopartikler25. Denne protokollen beskriver dermed en viktig anvendelse av et kombinert massefotometri mikrofluidikksystem – det muliggjør direkte måling av svake protein-protein-interaksjoner på nivået av individuelle molekyler og komplekser. Verdien av den foreliggende anvendelsen er høy, da den åpner muligheten for enkel karakterisering av interaksjoner som generelt har vært vanskelige å studere – med relevans på tvers av kritiske terapeutiske områder. Denne kombinerte tilnærmingen kan også tjene som grunnlag for et bredere spekter av undersøkelser for prøver med konsentrasjoner opp til titalls mikromolar.
Disclosures
The authors have nothing to disclose.
Acknowledgements
WS støttes av et UKRI Future Leaders Fellowship [MR / V02213X / 1]. Manuskriptteksten og grafikken ble utarbeidet med støtte fra medlemmer av Refeyns vitenskapelige kommunikasjonsteam (Panagiota Paganopoulou, Neus Torres Tamarit og Catherine Lichten). Vi takker også for verdifulle tilbakemeldinger fra Camille Hetez, Sofia Ferreira og Matthias Langhorst.
Materials
| 2-Propanol (Isopropanol) | VWR International LLC | 20880.320 | |
| Data acquisition software | Refeyn | AcquireMP (v2022 R1) | |
| Data analysis software | Refeyn | DiscoverMP (v2022 R1) | |
| FCRN, His-Tag | Sigma | SRP0624 | |
| Herceptin (IgG) | Cambridge Bioscience | HY-P9907-1mg | |
| Mass photometer | Refeyn | TwoMP | |
| Microfluidics box | Refeyn | MassFluidix HC system | |
| Microfluidics chip | Refeyn | MassFluidix HC chip | |
| Microfluidics control software | Fluigent | OxyGEN | |
| Phosphate Buffered Saline (PBS), 1x Ultra Pure | VWR International LLC | K812 | |
| Sodium Hydroxide (NaOH) | Sigma | S2770 | |
| β-Amylase, from sweet potato | Sigma | A8781 |
References
- Arter, W. E., Levin, A., Krainer, G., Knowles, T. P. J. Microfluidic approaches for the analysis of protein-protein interactions in solution. Biophysical Reviews. 12 (2), 575-585 (2020).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry. B. 122 (49), 11554-11560 (2018).
- Li, Z. Editorial: Methods in structural biology: Cryo-electron microscopy. Frontiers in Molecular Biosciences. 9, 1041386 (2022).
- Herling, T. W., et al. A microfluidic platform for real-time detection and quantification of protein-ligand interactions. Biophysical Journal. 110 (9), 1957-1966 (2016).
- Soltermann, F., et al. Quantifying protein-protein interactions by molecular counting with mass photometry. Angewandte Chemie International Edition. 59 (27), 10774-10779 (2020).
- Wu, D., Piszczek, G. Rapid determination of antibody-antigen affinity by mass photometry. Journal of Visualized Experiments: JoVE. 168, 61784 (2021).
- Wu, D., Piszczek, G. Measuring the affinity of protein-protein interactions on a single-molecule level by mass photometry. Analytical Biochemistry. 592, 113575 (2020).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- MassFluidix® HC system for rapid dilution via microfluidics. Available from: https://www.refeyn.com/massfluidix-hc-system (2023)
- Pollard, T. D. A guide to simple and informative binding assays. Molecular Biology of the Cell. 21 (23), 4061-4067 (2010).
- Jarmoskaite, I., AlSadhan, I., Vaidyanathan, P. P., Herschlag, D. How to measure and evaluate binding affinities. eLife. 9, e57264 (2020).
- Monnet, C., et al. Selection of IgG variants with increased FcRn binding using random and directed mutagenesis: Impact on effector functions. Frontiers in Immunology. 6, 39 (2015).
- . Refeyn TwoMP: Transforming biomolecular characterisation Available from: https://www.refeyn.com/twomp-mass-photometer (2022)
- Lai, S. -. H., Tamara, S., Heck, A. J. R. Single-particle mass analysis of intact ribosomes by mass photometry and Orbitrap-based charge detection mass spectrometry. iScience. 24 (11), 103211 (2021).
- Wu, D., Piszczek, G. Standard protocol for mass photometry experiments. European Biophysics Journal. 50 (3-4), 403-409 (2021).
- Vaughn, D. E., Bjorkman, P. J. Structural basis of pH-dependent antibody binding by the neonatal Fc receptor. Structure. 6 (1), 63-73 (1998).
- Niebling, S., et al. Biophysical screening pipeline for Cryo-EM grid preparation of membrane proteins. Frontiers in Molecular Biosciences. 9, 882288 (2022).
- Paul, S. S., Lyons, A., Kirchner, R., Woodside, M. T. Quantifying oligomer populations in real time during protein aggregation using single-molecule mass photometry. ACS Nano. 16 (10), 16462-16470 (2022).
- Schulz, L., et al. Evolution of increased complexity and specificity at the dawn of form I Rubiscos. Science. 378 (6616), 155-160 (2022).
- Malay, A. D., et al. An ultra-stable gold-coordinated protein cage displaying reversible assembly. Nature. 569 (7756), 438-442 (2019).
- Hundt, N., Cole, D., Hantke, M. F., Miller, J. J., Struwe, W. B., Kukura, P. Direct observation of the molecular mechanism underlying protein polymerization. Science Advances. 8 (35), eabm7935 (2022).
- Acharya, A., et al. Distinct RPA domains promote recruitment and the helicase-nuclease activities of Dna2. Nature Communications. 12, 6521 (2021).
- Ebberink, E. H. T. M., Ruisinger, A., Nuebel, M., Thomann, M., Heck, A. J. R. Assessing production variability in empty and filled adeno-associated viruses by single molecule mass analyses. Molecular Therapy – Methods & Clinical Development. 27, 491-501 (2022).
- Melo, L., et al. Size distributions of gold nanoparticles in solution measured by single-particle mass photometry. The Journal of Physical Chemistry B. 125 (45), 12466-12475 (2021).

