Análisis de la formación de complejos proteicos a concentraciones micromolares mediante el acoplamiento de microfluídica con fotometría de masas
Summary
Este protocolo combina la fotometría de masas con un novedoso sistema de microfluídica para investigar las interacciones proteína-proteína de baja afinidad. Este enfoque se basa en la dilución rápida de complejos altamente concentrados en solución, lo que permite mediciones de baja afinidad y amplía la aplicabilidad de la fotometría masiva.
Abstract
La fotometría de masas es una tecnología versátil de medición de masas que permite el estudio de las interacciones biomoleculares y la formación de complejos en solución sin etiquetas. La fotometría de masas es generalmente adecuada para analizar muestras en el rango de concentración de 100 pM-100 nM. Sin embargo, en muchos sistemas biológicos, es necesario medir muestras más concentradas para estudiar interacciones transitorias o de baja afinidad. Aquí, demostramos un método que expande de manera efectiva el rango de concentraciones de muestras que se pueden analizar por fotometría de masas desde nanomolares hasta decenas de micromolares.
En este protocolo, la fotometría de masas se combina con un novedoso sistema de microfluídica para investigar la formación de complejos proteicos en solución en el rango de concentración micromolar. Con el sistema de microfluídica, los usuarios pueden mantener una muestra a una concentración deseada más alta, seguida de una dilución hasta el rango nanomolar, varios milisegundos antes de la medición de la fotometría de masas. Debido a la velocidad de la dilución, los datos se obtienen antes de que el equilibrio de la muestra haya cambiado (es decir, disociación del complejo).
La técnica se aplica para medir las interacciones entre un anticuerpo de inmunoglobulina G (IgG) y el receptor Fc neonatal, mostrando la formación de complejos de alto orden que no fueron cuantificables con mediciones de fotometría de masa estática.
En conclusión, la combinación de fotometría de masas y microfluídica permite caracterizar muestras en el rango de concentración micromolar y es competente en la medición de interacciones biomoleculares con afinidades más débiles. Estas capacidades se pueden aplicar en una variedad de contextos, incluido el desarrollo y el diseño de bioterapéuticos, lo que permite una caracterización exhaustiva de diversas interacciones proteína-proteína.
Introduction
Las interacciones proteína-proteína subrayan la mayoría de las funciones celulares, desde la regulación inmunitaria hasta la replicación y traducción del ADN. Como resultado, existe una necesidad fundamental en todas las ciencias de la vida para investigar una amplia gama de interacciones a través de diversos complejos heterogéneos que se forman comúnmente. Sin embargo, su detección, caracterización y cuantificación suelen ser un reto, especialmente en el caso de las interacciones de baja afinidad1.
Los ensayos de inmunoprecipitación se utilizan a menudo para detectar interacciones de alta afinidad, pero para interacciones transitorias y de baja afinidad, la detección es en gran medida inviable2. También se pueden utilizar técnicas de fluorescencia, pero requieren la adición potencialmente disruptiva de etiquetasfluorescentes 2. Cryo-EM puede proporcionar una instantánea estructural y una lectura de conjunto de los complejos de proteínas formados con alta resolución espacial, pero también suele requerir trabajar a concentraciones que son demasiado bajas para obtener imágenes de interacciones de baja afinidad. Cryo-EM también presenta desafíos relacionados con el costo, la accesibilidad, la preparación de muestras y el tiempo de análisis3.
Además, la resonancia de plasmón de superficie (SPR) se ha convertido en una forma popular de cuantificar las interacciones proteína-proteína, aunque requiere la inmovilización de proteínas, lo que puede afectar el equilibrio de unión y dar lugar a tasas de activación variables, reduciendo así la precisión de la medición 4,5. También implica varios pasos de ensayo antes de la recopilación y el análisis de datos6.
La fotometría masiva es una técnica de una sola molécula que se ha utilizado para analizar las interacciones proteína-proteína 5,6,7. Funciona midiendo la masa de moléculas individuales o complejos en función de la luz que dispersan cuando aterrizan en la superficie de un cubreobjetos de vidrio8. Las mediciones de fotometría masiva se han utilizado para cuantificar las afinidades de unión a partir de la abundancia relativa de los socios de unión y los complejos que forman5. Sin embargo, al igual que otras técnicas de una sola molécula, la concentración de la muestra que se va a medir suele ser inferior a 100 nM. Si la concentración es mayor, las moléculas que aterrizan en la superficie del vidrio se superpondrán espacialmente, lo que dará como resultado una mala calidad de los datos7. En consecuencia, las interacciones más débiles (KD ~ micromolares), que se disocian a estas concentraciones más bajas, no pueden medirse de manera confiable ya que no es posible observar la mezcla necesaria de especies unidas y no unidas5.
Aquí, describimos un enfoque que supera esta limitación basado en un nuevo dispositivo de fotometría masiva de microfluídica acoplada. Específicamente, un sistema de microfluídica se utiliza en combinación con el fotómetro de masas para expandir de manera efectiva la gama de interacciones que se pueden cuantificar mediante fotometría de masas. Se ha demostrado que la microfluídica ofrece una gama de posibilidades para investigar las interacciones proteína-proteína, incluida la dilución rápida para detectar interacciones débiles 1,9. El sistema descrito en este documento funciona diluyendo rápidamente la muestra hasta 10.000 veces en un chip microfluídico y fluyendo inmediatamente a través del área de observación del chip, lo que permite que la medición de la fotometría de masas comience dentro de los 50 ms desde que las moléculas comenzaron el proceso de dilución10. La dilución se produce cuando la muestra y el tampón se combinan en un mezclador de válvula Tesla inversa en el chip, y los caudales relativos de las dos soluciones determinan la cantidad de dilución que se produce (consulte el paso 8 del protocolo). El caudal es controlable con el software de control microfluídico. La alteración del caudal puede cambiar la población relativa de la especie, ya que puede afectar el número de eventos de aterrizaje en la superficie del vidrio, que es lo que mide el fotómetro de masa.
La velocidad del proceso es lo suficientemente rápida como para que la medición se complete antes de que se interrumpa la integridad de la interacción (para más detalles, consulte también la Discusión). Esto se puede entender a través de una breve mirada a la teoría de las reacciones de primer orden, donde 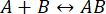 . La constante de velocidad hacia adelante (asociación) es kf, la constante de tasa hacia atrás (disociación) es kb y la constante de disociación de equilibrio (KD) se define como
. La constante de velocidad hacia adelante (asociación) es kf, la constante de tasa hacia atrás (disociación) es kb y la constante de disociación de equilibrio (KD) se define como
KD= kb/ kf
Para la unión a proteínas, kfestá generalmente limitada por la difusión de los reactivos11 y, por lo tanto, está restringida al rango de 10 6-107 M-1·s-1. Debido a que el rango de es limitado, una reacción de baja afinidad (KD ~ micromolares) tendrá kb≈ 1 s-1. Es decir, kb= kf · KD= (106 M-1·s-1) (10-6 M) = 1 s-1, con una vida media del complejo de alrededor de 0,7 s11,12.
Nuestro sistema de ejemplo es la unión del anticuerpo monoclonal IgG trastuzumab al dominio soluble del receptor Fc neonatal de IgG (FcRn), que se sabe que interactúan13. Los datos publicados anteriormente obtenidos utilizando solo fotometría masiva convencional (es decir, con dilución manual de muestras) mostraron que las proteínas forman múltiples especies. Los monómeros de FcRn, los dímeros de FcRn y la IgG no unida fueron claramente visibles, mientras que los complejos IgG-FcRn (en proporciones 1:1 y 1:2) también se detectaron (a pH 5,0) pero solo con una abundancia muy baja5. Esta observación plantea la cuestión de si la formación del complejo IgG-FcRn podría detectarse más claramente si se mide a una concentración más alta. De hecho, la combinación de la fotometría masiva con un enfoque de dilución rápida acoplada descrito aquí proporcionó pruebas más sólidas de la formación de complejos mediante un aumento de las partículas medidas.
El protocolo de fotometría de masas y microfluídica aquí descrito permite caracterizar la formación de complejos con un KD hasta el rango micromolar. Una determinación empírica del KD requerirá mejoras adicionales en la precisión del sensor de flujo, la estabilidad de la bomba, las variaciones de viruta a viruta y la ubicación de la medición dentro de la ventana de observación, ya que todos estos factores influirían en el tiempo desde que la muestra se diluye hasta que se mide.
El mismo enfoque podría aplicarse para investigar la unión entre cualquier proteína soluble, siempre que tenga pesos moleculares distintos (separados por al menos 25 kDa) que se encuentren en el rango adecuado para el análisis con un fotómetro de masa (30 kDa a 6 MDa). Los conocimientos obtenidos podrían ser útiles para estudios en una variedad de contextos, desde la obtención de una comprensión mecanicista de las funciones celulares hasta el diseño de nuevos medicamentos bioterapéuticos.
Protocol
Representative Results
Discussion
El protocolo descrito aquí proporciona un método para detectar y cuantificar las interacciones proteína-proteína de baja afinidad. Utiliza un fotómetro de masas acoplado a un sistema de microfluídica de dilución rápida. La fotometría de masas es una herramienta bioanalítica sin marcadores que puede medir de forma fiable la masa molecular en solución para biomoléculas16, para aquellas dentro del rango de 30 kDa a 6 MDa. Como la fotometría de masas es una técnica de una sola molécula que analiza las muestras una por una, generalmente se limita a muestras en el rango de concentración de 100 pM-100 nM. Por encima de este rango, las moléculas que aterrizan en la superficie del vidrio se superpondrán espacialmente, lo que dará lugar a una mala calidad de los datos; Por debajo de este rango, se obtienen muy pocos datos para realizar un análisis robusto7. Una consecuencia importante es que puede limitar la investigación de las interacciones de las proteínas a aquellas que forman una mezcla de especies unidas y no unidas dentro de ese rango.
Aquí, detallamos un protocolo paso a paso para usar un sistema de microfluídica de dilución rápida para expandir de manera efectiva el rango de concentraciones de muestras que son susceptibles de fotometría masiva. Al diluir la muestra en el chip microfluídico y luego hacerla fluir a través de la ventana de observación del detector dentro de los 50 ms, el sistema captura los complejos presentes en la muestra sin diluir antes de que cambie el equilibrio de interacción. La muestra se suministra continuamente al detector durante las mediciones individuales. En estas condiciones, el 95% del complejo permanecerá intacto cuando se mida la muestra, incluso para interacciones de baja afinidad, con un KD del orden de los micromolares y tasas de disociación tan rápidas como 1 s-1.
Esto se puede calcular de la siguiente manera: Para una reacción 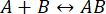 con una velocidad hacia adelante kfy una velocidad hacia atrás kb,
con una velocidad hacia adelante kfy una velocidad hacia atrás kb,
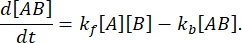
En equilibrio, las concentraciones de las tres especies (A, B y el complejo AB) permanecen constantes, por lo que 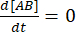 y
y 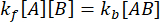 . Bajo la suposición conservadora de que la perturbación (dilución, en este caso) puede hacer que el complejo se disocie, pero la reacción directa (asociación) no procede, el término kf [A] [B] puede tratarse como despreciable, y se puede hacer la siguiente simplificación:
. Bajo la suposición conservadora de que la perturbación (dilución, en este caso) puede hacer que el complejo se disocie, pero la reacción directa (asociación) no procede, el término kf [A] [B] puede tratarse como despreciable, y se puede hacer la siguiente simplificación:
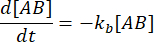
La integración da la siguiente expresión para la concentración de complejo en el tiempo después de la perturbación del equilibrio:
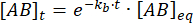
La fracción del complejo que permanece ligada en el tiempo t después de la perturbación del equilibrio es así:
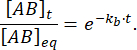
A = 50 ms, para una reacción con kb≈ 1 s-1, el enlace de la fracción es 0,95, o 95%11,12.
La fotometría masiva se utilizó aquí y anteriormente5 para investigar la unión del anticuerpo monoclonal IgG trastuzumab al dominio soluble del FcRn. Se ha informado que los dos socios de unión se unen con afinidad nanomolar a pHácido 17. Se utilizó fotometría masiva para evaluar cualitativamente la abundancia de los complejos formados mientras los socios de unión estaban a pH 5.0, y las muestras se diluyeron rápidamente a través de un sistema microfluídico adicional. El procedimiento fue optimizado para la interacción proteína-proteína particular con base en los resultados reportados previamente5. El mismo procedimiento se puede utilizar para estudiar otras interacciones, siempre que los usuarios tengan conocimientos previos u optimicen las condiciones experimentales para el sistema en cuestión, como qué tampones utilizar, la concentración inicial de proteínas, la estequiometría esperada y la cantidad de incubación necesaria para permitir que la interacción alcance un equilibrio.
Cuando la mezcla IgG-FcRn se diluyó manualmente, fue difícil detectar la presencia de complejos IgG-FcRn, a pesar de que se sabe que estas proteínas interactúan5. Este trabajo muestra que el enfoque de dilución rápida da como resultado una cantidad notablemente mayor de estos complejos. Para la misma muestra, cuando se utilizó una dilución rápida, se observaron claramente complejos FcRn-IgG 1:1 y 2:1 FcRn-IgG. Estas diferencias en la formación de complejos demuestran la importancia de estudiar los sistemas de interacción biomolecular en una amplia gama de concentraciones.
Además, estos resultados también demuestran que es sencillo utilizar la microfluídica con el análisis de una sola molécula para capturar interacciones débiles, llenando un vacío significativo en el método. La combinación de la microfluídica de dilución rápida con la fotometría de masas ofrece ventajas atractivas debido a las ventajas de la fotometría de masas como técnica analítica. Es decir, la fotometría masiva no requiere etiquetas, implica una preparación mínima de la muestra y las mediciones se realizan en solución. Para este protocolo, otra ventaja clave de la fotometría masiva es su capacidad para distinguir y cuantificar todas las especies formadas (siempre que tengan una masa distinta de >30 kDa). Esto contrasta con la SPR, por ejemplo, que puede medir las tasas de unión y desunión, pero no puede proporcionar fácilmente información estequiométrica8.
Para este protocolo, así como para los experimentos de fotometría masiva en general, son útiles varias consideraciones. En primer lugar, la concentración final de proteínas debe estar dentro del límite de lo que la fotometría de masas puede medir (100 pM-100 nM). La concentración de incubación inicial también debe estar dentro del rango del sistema microfluídico (hasta 90 μM) y se teoriza que está por encima del KD real de la interacción10. El punto de partida recomendado es una proporción de mezcla de concentración de 1:1 entre las especies que interactúan a una concentración de μM. La relación podría entonces variarse a 1:2, 1:5 o, como en el caso de esta interacción, 1:10. Si no hay información previa sobre las interacciones de las proteínas, el usuario tendría que optimizar el experimento, comenzando con una concentración alta (recomendada 20 μM) para cada socio para determinar si la afinidad de los componentes está dentro del rango de concentración sostenido por el método presentado (es decir, se forman complejos). La optimización también puede implicar la elección de otras condiciones de búfer para promover las interacciones o la valoración de uno de los componentes de interacción para determinar la proporción de mezcla correcta. Una vez determinados, es posible optimizar las concentraciones y los caudales para permitir condiciones óptimas para el estudio y el método, por ejemplo, disminuyendo las concentraciones para permitir una mejor resolución de los picos.
En segundo lugar, para replicar con éxito este experimento, se deben minimizar las impurezas. Las fuentes comunes de impurezas que se sabe que afectan negativamente a las mediciones de fotometría masiva incluyen otras proteínas o desechos celulares que permanecen después de la purificación, tampones sin filtrar, detergentes formadores de micelas (si están presentes en una concentración demasiado alta) y tampones que contienen altas concentraciones de sal, glicerol u otros componentes. Como se discutió en el Protocolo anterior, las burbujas en el sistema microfluídico deben eliminarse. Se pueden formar burbujas en el sistema de tubos o si las muestras tienen una alta tensión superficial y son propensas a la formación de espuma. También se pueden formar burbujas en el aceite de inmersión, que se pueden detectar desde el anillo de enfoque (Figura 3). Si las burbujas no se pueden eliminar siguiendo los pasos descritos en el protocolo, otra solución es desgasificar la muestra con un desecador y una bomba de vacío, dejando la muestra a presión reducida durante unos minutos. No se recomienda agitar o agitar soluciones de proteínas altamente concentradas, ya que estas acciones pueden promover la formación de burbujas.
Si bien aquí se demuestra la medición de una interacción proteína-proteína específica, el mismo protocolo se puede aplicar a otros sistemas de interacción proteína-proteína sin modificaciones significativas. Otra dirección futura de este protocolo sería utilizar las mediciones para calcular los valores deK D para los complejos identificados, como se ha descrito en otro lugar en el contexto de la fotometría de masas 5,7. Si bien los estudios anteriores utilizaron datos de experimentos que involucraban dilución manual e interacciones más fuertes, el principio de análisis podría aplicarse fácilmente en este contexto, siempre que se implementen mejoras adicionales en el dispositivo microfluídico (como una mayor precisión del sensor de flujo y estabilidad de la bomba).
Más allá de las interacciones proteína-proteína, es probable que haya aplicaciones más amplias para el enfoque combinado de fotometría de masas y microfluídica de dilución rápida. La fotometría masiva se puede utilizar para evaluar la pureza, agregación y homogeneidad de la muestra 18,19; estudiar la oligomerizaciónde proteínas 20, el ensamblaje macromolecular21 o la polimerización22; y en otros ámbitos. El análisis de fotometría masiva también se extiende más allá de las proteínas; Se ha utilizado para investigar las interacciones entre los ácidos nucleicos y las proteínas23, las partículas virales24 y las nanopartículas25. Por lo tanto, este protocolo describe una aplicación importante de un sistema combinado de microfluídica de fotometría de masas: permite la medición directa de interacciones débiles proteína-proteína a nivel de moléculas y complejos individuales. El valor de la presente aplicación es alto, ya que abre la posibilidad de caracterizar directamente interacciones que generalmente han sido difíciles de estudiar, con relevancia en áreas terapéuticas críticas. Este enfoque combinado también podría servir como base para una gama más amplia de investigaciones para muestras con concentraciones de hasta decenas de micromolares.
Disclosures
The authors have nothing to disclose.
Acknowledgements
W.S. cuenta con el apoyo de una beca de Futuros Líderes de UKRI [MR/V02213X/1]. El texto y los gráficos del manuscrito se prepararon con el apoyo de los miembros del equipo de comunicación científica de Refeyn (Panagiota Paganopoulou, Neus Torres Tamarit y Catherine Lichten). También agradecemos los valiosos comentarios de Camille Hetez, Sofia Ferreira y Matthias Langhorst.
Materials
| 2-Propanol (Isopropanol) | VWR International LLC | 20880.320 | |
| Data acquisition software | Refeyn | AcquireMP (v2022 R1) | |
| Data analysis software | Refeyn | DiscoverMP (v2022 R1) | |
| FCRN, His-Tag | Sigma | SRP0624 | |
| Herceptin (IgG) | Cambridge Bioscience | HY-P9907-1mg | |
| Mass photometer | Refeyn | TwoMP | |
| Microfluidics box | Refeyn | MassFluidix HC system | |
| Microfluidics chip | Refeyn | MassFluidix HC chip | |
| Microfluidics control software | Fluigent | OxyGEN | |
| Phosphate Buffered Saline (PBS), 1x Ultra Pure | VWR International LLC | K812 | |
| Sodium Hydroxide (NaOH) | Sigma | S2770 | |
| β-Amylase, from sweet potato | Sigma | A8781 |
References
- Arter, W. E., Levin, A., Krainer, G., Knowles, T. P. J. Microfluidic approaches for the analysis of protein-protein interactions in solution. Biophysical Reviews. 12 (2), 575-585 (2020).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry. B. 122 (49), 11554-11560 (2018).
- Li, Z. Editorial: Methods in structural biology: Cryo-electron microscopy. Frontiers in Molecular Biosciences. 9, 1041386 (2022).
- Herling, T. W., et al. A microfluidic platform for real-time detection and quantification of protein-ligand interactions. Biophysical Journal. 110 (9), 1957-1966 (2016).
- Soltermann, F., et al. Quantifying protein-protein interactions by molecular counting with mass photometry. Angewandte Chemie International Edition. 59 (27), 10774-10779 (2020).
- Wu, D., Piszczek, G. Rapid determination of antibody-antigen affinity by mass photometry. Journal of Visualized Experiments: JoVE. 168, 61784 (2021).
- Wu, D., Piszczek, G. Measuring the affinity of protein-protein interactions on a single-molecule level by mass photometry. Analytical Biochemistry. 592, 113575 (2020).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- MassFluidix® HC system for rapid dilution via microfluidics. Available from: https://www.refeyn.com/massfluidix-hc-system (2023)
- Pollard, T. D. A guide to simple and informative binding assays. Molecular Biology of the Cell. 21 (23), 4061-4067 (2010).
- Jarmoskaite, I., AlSadhan, I., Vaidyanathan, P. P., Herschlag, D. How to measure and evaluate binding affinities. eLife. 9, e57264 (2020).
- Monnet, C., et al. Selection of IgG variants with increased FcRn binding using random and directed mutagenesis: Impact on effector functions. Frontiers in Immunology. 6, 39 (2015).
- . Refeyn TwoMP: Transforming biomolecular characterisation Available from: https://www.refeyn.com/twomp-mass-photometer (2022)
- Lai, S. -. H., Tamara, S., Heck, A. J. R. Single-particle mass analysis of intact ribosomes by mass photometry and Orbitrap-based charge detection mass spectrometry. iScience. 24 (11), 103211 (2021).
- Wu, D., Piszczek, G. Standard protocol for mass photometry experiments. European Biophysics Journal. 50 (3-4), 403-409 (2021).
- Vaughn, D. E., Bjorkman, P. J. Structural basis of pH-dependent antibody binding by the neonatal Fc receptor. Structure. 6 (1), 63-73 (1998).
- Niebling, S., et al. Biophysical screening pipeline for Cryo-EM grid preparation of membrane proteins. Frontiers in Molecular Biosciences. 9, 882288 (2022).
- Paul, S. S., Lyons, A., Kirchner, R., Woodside, M. T. Quantifying oligomer populations in real time during protein aggregation using single-molecule mass photometry. ACS Nano. 16 (10), 16462-16470 (2022).
- Schulz, L., et al. Evolution of increased complexity and specificity at the dawn of form I Rubiscos. Science. 378 (6616), 155-160 (2022).
- Malay, A. D., et al. An ultra-stable gold-coordinated protein cage displaying reversible assembly. Nature. 569 (7756), 438-442 (2019).
- Hundt, N., Cole, D., Hantke, M. F., Miller, J. J., Struwe, W. B., Kukura, P. Direct observation of the molecular mechanism underlying protein polymerization. Science Advances. 8 (35), eabm7935 (2022).
- Acharya, A., et al. Distinct RPA domains promote recruitment and the helicase-nuclease activities of Dna2. Nature Communications. 12, 6521 (2021).
- Ebberink, E. H. T. M., Ruisinger, A., Nuebel, M., Thomann, M., Heck, A. J. R. Assessing production variability in empty and filled adeno-associated viruses by single molecule mass analyses. Molecular Therapy – Methods & Clinical Development. 27, 491-501 (2022).
- Melo, L., et al. Size distributions of gold nanoparticles in solution measured by single-particle mass photometry. The Journal of Physical Chemistry B. 125 (45), 12466-12475 (2021).

