إنشاء طريقة عرض تنظيم النسخ لميزات النسخ لمهمة التنبؤ واكتشاف العلامات الحيوية المظلمة على مجموعات البيانات الصغيرة
Summary
هنا ، نقدم بروتوكولا لتحويل البيانات النسخية إلى عرض mqTrans ، مما يتيح تحديد المؤشرات الحيوية المظلمة. على الرغم من عدم التعبير عنها بشكل تفاضلي في التحليلات النسخية التقليدية ، إلا أن هذه المؤشرات الحيوية تظهر تعبيرا تفاضليا في عرض mqTrans. يعمل هذا النهج كتقنية تكميلية للطرق التقليدية ، حيث يكشف النقاب عن المؤشرات الحيوية التي تم تجاهلها سابقا.
Abstract
يمثل Transcriptome مستويات التعبير للعديد من الجينات في العينة وقد استخدم على نطاق واسع في الأبحاث البيولوجية والممارسة السريرية. ركز الباحثون عادة على المؤشرات الحيوية النسخية ذات التمثيلات التفاضلية بين مجموعة النمط الظاهري ومجموعة التحكم من العينات. قدمت هذه الدراسة إطار عمل تعليمي متعدد المهام لشبكة الانتباه البياني (GAT) لمعرفة التفاعلات المعقدة بين الجينات للعينات المرجعية. تم تدريب نموذج مرجعي توضيحي مسبقا على العينات الصحية (HealthModel) ، والتي يمكن استخدامها مباشرة لتوليد عرض تنظيم النسخ الكمي القائم على النموذج (mqTrans) لنسخ الاختبار المستقلة. تم عرض عرض mqTrans الذي تم إنشاؤه للنسخ من خلال مهام التنبؤ واكتشاف العلامات الحيوية المظلمة. نشأ المصطلح “العلامة الحيوية الداكنة” من تعريفه بأن العلامة الحيوية المظلمة أظهرت تمثيلا تفاضليا في عرض mqTrans ولكن لا يوجد تعبير تفاضلي في مستوى التعبير الأصلي. تم دائما تجاهل العلامات الحيوية الداكنة في دراسات الكشف عن العلامات الحيوية التقليدية بسبب عدم وجود تعبير تفاضلي. يمكن تنزيل الكود المصدري ودليل خط الأنابيب HealthModelPipe من http://www.healthinformaticslab.org/supp/resources.php.
Introduction
يتكون Transcriptome من تعبيرات جميع الجينات في العينة ويمكن توصيفه بواسطة تقنيات عالية الإنتاجية مثل microarray و RNA-seq1. تسمى مستويات التعبير لجين واحد في مجموعة البيانات ميزة النسخ ، والتمثيل التفاضلي لميزة النسخ بين النمط الظاهري والمجموعات الضابطة يعرف هذا الجين كعلامة حيوية لهذا النمط الظاهري 2,3. تم استخدام المؤشرات الحيوية النسخية على نطاق واسع في التحقيقات في تشخيص المرض4 ، والآلية البيولوجية5 ، وتحليل البقاء على قيد الحياة6،7 ، إلخ.
تحمل أنماط نشاط الجينات في الأنسجة السليمة معلومات مهمة عن حياة 8,9. تقدم هذه الأنماط رؤى لا تقدر بثمن وتعمل كمراجع مثالية لفهم المسارات التنموية المعقدة للاضطرابات الحميدة10,11 والأمراض الفتاكة12. تتفاعل الجينات مع بعضها البعض ، وتمثل النسخ مستويات التعبير النهائية بعد تفاعلاتها المعقدة. تتم صياغة هذه الأنماط مثل شبكة تنظيم النسخ13 وشبكة التمثيل الغذائي14 ، إلخ. يمكن تنظيم تعبيرات الحمض النووي الريبي المرسال (mRNAs) نسخيا بواسطة عوامل النسخ (TFs) والحمض النووي الريبي الطويل غير المشفر بين الجينات (lincRNAs)15،16،17. تجاهل تحليل التعبير التفاضلي التقليدي مثل هذه التفاعلات الجينية المعقدة مع افتراض الاستقلال بين الميزات18,19.
تظهر التطورات الحديثة في الشبكات العصبية للرسم البياني (GNNs) إمكانات غير عادية في استخراج المعلومات المهمة من البيانات المستندة إلى OMIC لدراسات السرطان20 ، على سبيل المثال ، تحديد وحدات التعبير المشترك21. القدرة الفطرية ل GNNs تجعلها مثالية لنمذجة العلاقات المعقدة والتبعيات بين الجينات22,23.
غالبا ما تركز الدراسات الطبية الحيوية على التنبؤ بدقة بالنمط الظاهري مقابل المجموعة الضابطة. عادة ما تصاغ هذه المهام كتصنيفات ثنائية24،25،26. هنا ، عادة ما يتم ترميز تسميات الفئتين على أنها 1 و 0 ، صواب وخطأ ، أو حتى موجب وسالب27.
تهدف هذه الدراسة إلى توفير بروتوكول سهل الاستخدام لتوليد عرض تنظيم النسخ (mqTrans) لمجموعة بيانات النسخ بناء على النموذج المرجعي لشبكة انتباه الرسم البياني (GAT) المدرب مسبقا. تم استخدام إطار عمل GAT متعدد المهام من عمل منشور سابقا26 لتحويل ميزات النسخ إلى ميزات mqTrans. تم استخدام مجموعة بيانات كبيرة من النسخ الصحية من جامعة كاليفورنيا ، سانتا كروز (UCSC) منصة Xena28 للتدريب المسبق للنموذج المرجعي (HealthModel) ، والذي يقيس كميا لوائح النسخ من العوامل التنظيمية (TFs و lincRNAs) إلى mRNAs المستهدفة. يمكن استخدام عرض mqTrans الذي تم إنشاؤه لبناء نماذج التنبؤ واكتشاف المؤشرات الحيوية المظلمة. يستخدم هذا البروتوكول مجموعة بيانات مرضى سرطان القولون الغدي (COAD) من قاعدة بيانات أطلس جينوم السرطان (TCGA)29 كمثال توضيحي. في هذا السياق ، يتم تصنيف المرضى في المرحلتين الأولى أو الثانية على أنهم عينات سلبية ، بينما يعتبر المرضى في المرحلتين الثالثة أو الرابعة عينات إيجابية. كما تتم مقارنة توزيعات المؤشرات الحيوية الداكنة والتقليدية عبر 26 نوعا من أنواع السرطان TCGA.
وصف خط أنابيب HealthModel
تستند المنهجية المستخدمة في هذا البروتوكول إلى الإطار26 المنشور سابقا ، كما هو موضح في الشكل 1. للبدء، يطلب من المستخدمين إعداد مجموعة بيانات الإدخال، وإدخالها في خط أنابيب HealthModel المقترح، والحصول على ميزات mqTrans. وترد تعليمات مفصلة لإعداد البيانات في القسم 2 من قسم البروتوكول. بعد ذلك، يتوفر للمستخدمين خيار دمج ميزات mqTrans مع ميزات النسخ الأصلية أو متابعة ميزات mqTrans التي تم إنشاؤها فقط. ثم تخضع مجموعة البيانات المنتجة لعملية اختيار الميزات ، حيث يتمتع المستخدمون بالمرونة في اختيار القيمة المفضلة لديهم ل k في التحقق المتقاطع k-fold للتصنيف. مقياس التقييم الأساسي المستخدم في هذا البروتوكول هو الدقة.
يصنف HealthModel26 ميزات النسخ إلى ثلاث مجموعات متميزة: TF (عامل النسخ) ، lincRNA (الحمض النووي الريبي الطويل غير المشفر بين الجينات) ، و mRNA (الحمض النووي الريبي المرسال). يتم تعريف ميزات TF بناء على التعليقات التوضيحية المتوفرة في أطلس البروتين البشري30,31. يستخدم هذا العمل التعليقات التوضيحية ل lincRNAs من مجموعة بياناتGTEx 32. تعتبر الجينات التي تنتمي إلى مسارات المستوى الثالث في قاعدة بيانات KEGG33 من ميزات mRNA. تجدر الإشارة إلى أنه إذا أظهرت ميزة mRNA أدوارا تنظيمية للجين المستهدف كما هو موثق في قاعدة بيانات TRRUST34 ، إعادة تصنيفها في فئة TF.
يقوم هذا البروتوكول أيضا بإنشاء ملفين نموذجيين يدويا لمعرفات الجينات للعوامل التنظيمية (regulatory_geneIDs.csv) و mRNA المستهدف (target_geneIDs.csv). يتم حساب مصفوفة المسافة الزوجية بين السمات التنظيمية (TFs و lincRNAs) بواسطة معاملات ارتباط بيرسون ويتم تجميعها بواسطة أداة تحليل شبكة التعبير المشترك للجين المرجح (WGCNA) 36 (adjacent_matrix.csv). يمكن للمستخدمين استخدام خط أنابيب HealthModel مباشرة مع ملفات التكوين النموذجية هذه لإنشاء عرض mqTrans لمجموعة بيانات نسخية.
التفاصيل الفنية لنموذج الصحة
يمثل HealthModel العلاقات المعقدة بين TFs و lincRNAs كرسم بياني ، مع ميزات الإدخال التي تعمل كرؤوس يشار إليها بواسطة V ومصفوفة حافة بين الرؤوس تسمى E. تتميز كل عينة بميزات تنظيمية K ، يرمز لها ب VK ×1. على وجه التحديد ، شملت مجموعة البيانات 425 TFs و 375 lincRNAs ، مما أدى إلى أبعاد عينة من K = 425 + 375 = 800. لإنشاء مصفوفة الحافة E ، استخدم هذا العمل الأداة الشعبية WGCNA35. يتم تحديد الوزن الزوجي الذي يربط رأسين ممثلين ك 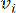 و
و 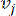 ، بواسطة معامل ارتباط بيرسون. تعرض شبكة تنظيم الجينات طوبولوجيا خالية من المقياس36 ، تتميز بوجود جينات محورية ذات أدوار وظيفية محورية. نحسب الارتباط بين ميزتين أو رأسين ،
، بواسطة معامل ارتباط بيرسون. تعرض شبكة تنظيم الجينات طوبولوجيا خالية من المقياس36 ، تتميز بوجود جينات محورية ذات أدوار وظيفية محورية. نحسب الارتباط بين ميزتين أو رأسين ، 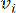 و
و 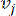 ، باستخدام مقياس التداخل الطوبولوجي (TOM) على النحو التالي:
، باستخدام مقياس التداخل الطوبولوجي (TOM) على النحو التالي:
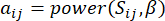 (1)
(1)
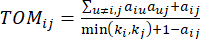 (2)
(2)
يتم حساب β العتبة اللينة باستخدام وظيفة “عتبة pickSoft” من حزمة WGCNA. يتم تطبيق دالة أس القدرة aij ، حيث 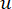 تمثل جينا باستثناء i و j ، وتمثل
تمثل جينا باستثناء i و j ، وتمثل 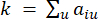 اتصال الرأس. يقوم WGCNA بتجميع ملفات تعريف التعبير لميزات النسخ في وحدات متعددة باستخدام مقياس الاختلاف المستخدم بشكل شائع (
اتصال الرأس. يقوم WGCNA بتجميع ملفات تعريف التعبير لميزات النسخ في وحدات متعددة باستخدام مقياس الاختلاف المستخدم بشكل شائع (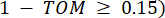 37.
37.
تم تصميم إطار عمل HealthModel في الأصل كبنية تعليمية متعددة المهام26. يستخدم هذا البروتوكول فقط مهمة التدريب المسبق النموذجية لبناء طريقة عرض mqTrans النسخية. يمكن للمستخدم اختيار زيادة تحسين HealthModel المدرب مسبقا ضمن شبكة انتباه الرسم البياني متعدد المهام مع عينات نسخ إضافية خاصة بالمهمة.
التفاصيل الفنية لاختيار الميزة وتصنيفها
ينفذ تجمع تحديد المعالم أحد عشر خوارزمية لتحديد المعالم (FS). من بينها ، ثلاثة خوارزميات FS قائمة على المرشح: اختيار أفضل ميزات K باستخدام معامل المعلومات الأقصى (SK_mic) ، واختيار ميزات K بناء على FPR ل MIC (SK_fpr) ، واختيار ميزات K ذات أعلى معدل اكتشاف خاطئ ل MIC (SK_fdr). بالإضافة إلى ذلك ، تقوم ثلاث خوارزميات FS قائمة على الأشجار بتقييم الميزات الفردية باستخدام شجرة القرار مع مؤشر Gini (DT_gini) ، وأشجار القرار المعززة التكيفية (AdaBoost) ، والغابة العشوائية (RF_fs). يشتمل التجمع أيضا على طريقتين للتغليف: إزالة الميزة العودية باستخدام مصنف متجه الدعم الخطي (RFE_SVC) والتخلص من الميزة العودية باستخدام مصنف الانحدار اللوجستي (RFE_LR). أخيرا ، يتم تضمين خوارزميتين للتضمين: مصنف SVC الخطي مع قيم أهمية ميزة L1 الأعلى مرتبة (lSVC_L1) ومصنف الانحدار اللوجستي مع قيم أهمية ميزة L1 الأعلى مرتبة (LR_L1).
يستخدم تجمع المصنف سبعة مصنفات مختلفة لبناء نماذج تصنيف. تشتمل هذه المصنفات على آلة متجه الدعم الخطي (SVC) ، و Gaussian Naïve Bayes (GNB) ، ومصنف الانحدار اللوجستي (LR) ، وأقرب جار k ، مع تعيين k على 5 افتراضيا (KNN) ، و XGBoost ، والغابة العشوائية (RF) ، وشجرة القرار (DT).
يمكن تعيين التقسيم العشوائي لمجموعة البيانات في القطار: مجموعات فرعية للاختبار في سطر الأوامر. يستخدم المثال الموضح نسبة القطار: اختبار = 8: 2.
Protocol
Representative Results
Discussion
القسم 2 (استخدم HealthModel المدرب مسبقا لإنشاء ميزات mqTrans) من البروتوكول هو الخطوة الأكثر أهمية في هذا البروتوكول. بعد إعداد بيئة العمل الحسابية في القسم 1 ، يقوم القسم 2 بإنشاء عرض mqTrans لمجموعة بيانات نسخية بناء على النموذج المرجعي الكبير المدرب مسبقا. القسم 3 هو مثال توضيحي لاختيار ميزات mqTrans ال?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
تم دعم هذا العمل من قبل فريق الابتكار التكنولوجي الأول والصغير (20210509055RQ) ، ومشاريع العلوم والتكنولوجيا بمقاطعة قويتشو (ZK2023-297) ، ومؤسسة العلوم والتكنولوجيا التابعة للجنة الصحة بمقاطعة قويتشو (gzwkj2023-565) ، ومشروع العلوم والتكنولوجيا التابع لإدارة التعليم في مقاطعة جيلين (JJKH20220245KJ و JJKH20220226SK) ، والمؤسسة الوطنية للعلوم الطبيعية في الصين (U19A2061) ، ومختبر مقاطعة جيلين الرئيسي للحوسبة الذكية للبيانات الضخمة (20180622002JC) ، وصناديق البحوث الأساسية للجامعات المركزية ، JLU. نعرب عن خالص تقديرنا لمحرر المراجعة والمراجعين الثلاثة المجهولين على انتقاداتهم البناءة ، والتي كانت مفيدة في تعزيز صرامة ووضوح هذا البروتوكول بشكل كبير.
Materials
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
References
- Mutz, K. -. O., Heilkenbrinker, A., Lönne, M., Walter, J. -. G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516 (2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684 (2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352 (2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528 (2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. . Analysis of Survival Data. , (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866 (2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307 (2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119 (2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239 (2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334 (2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338 (2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. . BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , (2022).
- Muzio, G., O’Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738 (2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5 (2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883 (2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582 (2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801 (2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138 (2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308 (2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).

