Generering av transkripsjonsreguleringsvisning av transkriptomiske funksjoner for prediksjonsoppgave og mørk biomarkørdeteksjon på små datasett
Summary
Her introduserer vi en protokoll for konvertering av transkriptomiske data til en mqTrans-visning, noe som muliggjør identifisering av mørke biomarkører. Selv om de ikke er differensielt uttrykt i konvensjonelle transkriptomiske analyser, viser disse biomarkørene differensielt uttrykk i mqTrans-visningen. Tilnærmingen fungerer som en komplementær teknikk til tradisjonelle metoder, og avdekker tidligere oversett biomarkører.
Abstract
Transkriptom representerer ekspresjonsnivåene av mange gener i en prøve og har vært mye brukt i biologisk forskning og klinisk praksis. Forskere fokuserte vanligvis på transkriptomiske biomarkører med differensielle representasjoner mellom en fenotypegruppe og en kontrollgruppe av prøver. Denne studien presenterte et læringsrammeverk for multitask graph-attention network (GAT) for å lære de komplekse intergeniske interaksjonene til referanseprøvene. En demonstrativ referansemodell var forhåndstrent på de friske prøvene (HealthModel), som kunne brukes direkte til å generere modellbasert kvantitativ transkripsjonsregulering (mqTrans) visning av de uavhengige testtranskriptomene. Den genererte mqTrans-visningen av transkriptomer ble demonstrert ved prediksjonsoppgaver og mørk biomarkørdeteksjon. Begrepet “mørk biomarkør” stammet fra definisjonen om at en mørk biomarkør viste differensiell representasjon i mqTrans-visningen, men ingen differensialuttrykk i sitt opprinnelige uttrykksnivå. En mørk biomarkør ble alltid oversett i tradisjonelle biomarkørdeteksjonsstudier på grunn av fravær av differensialuttrykk. Kildekoden og manualen for rørledningen HealthModelPipe kan lastes ned fra http://www.healthinformaticslab.org/supp/resources.php.
Introduction
Transkriptom består av uttrykkene av alle genene i en prøve og kan profileres av høykapasitetsteknologier som mikroarray og RNA-seq1. Ekspresjonsnivåene til ett gen i et datasett kalles en transkriptomisk funksjon, og differensialrepresentasjonen av et transkriptomisk trekk mellom fenotype- og kontrollgruppene definerer dette genet som en biomarkør for denne fenotypen 2,3. Transkriptomiske biomarkører har blitt mye brukt i undersøkelsene av sykdomsdiagnose4, biologisk mekanisme5 og overlevelsesanalyse 6,7, etc.
Genaktivitetsmønstre i det friske vevet bærer viktig informasjon om livene 8,9. Disse mønstrene gir uvurderlig innsikt og fungerer som ideelle referanser for å forstå de komplekse utviklingsbanene til godartede lidelser 10,11 og dødelige sykdommer12. Gener interagerer med hverandre, og transkriptomer representerer de endelige uttrykksnivåene etter deres kompliserte interaksjoner. Slike mønstre er formulert som transkripsjonsreguleringsnettverk13 og metabolismenettverk14, etc. Uttrykkene av budbringer-RNA (mRNA) kan transkripsjonelt reguleres av transkripsjonsfaktorer (TF) og lange intergeniske ikke-kodende RNA (lincRNAer)15,16,17. Konvensjonell differensialekspresjonsanalyse ignorerte slike komplekse geninteraksjoner med antagelsen om uavhengighet mellom funksjoner 18,19.
Nylige fremskritt i grafnevrale nettverk (GNN) viser ekstraordinært potensial i å trekke ut viktig informasjon fra OMIC-baserte data for kreftstudier20, for eksempel å identifisere co-uttrykksmoduler21. Den medfødte kapasiteten til GNN gjør dem ideelle for modellering av de intrikate forholdene og avhengighetene mellom gener22,23.
Biomedisinske studier fokuserer ofte på nøyaktig å forutsi en fenotype mot kontrollgruppen. Slike oppgaver er vanligvis formulert som binære klassifikasjoner 24,25,26. Her er de to klasseetikettene vanligvis kodet som 1 og 0, sant og usant, eller til og med positivt og negativt27.
Denne studien hadde som mål å gi en brukervennlig protokoll for generering av transkripsjonsregulering (mqTrans) visning av et transkriptomdatasett basert på den forhåndstrente graf-oppmerksomhetsnettverket (GAT) referansemodell. Multitask GAT-rammeverket fra et tidligere publisert arbeid26 ble brukt til å transformere transkriptomiske funksjoner til mqTrans-funksjonene. Et stort datasett med friske transkriptomer fra University of California, Santa Cruz (UCSC) Xena-plattform28 ble brukt til å forhåndstrene referansemodellen (HealthModel), som kvantitativt målte transkripsjonsforskriftene fra regulatoriske faktorer (TF og lincRNA) til mål-mRNAene. Den genererte mqTrans-visningen kan brukes til å bygge prediksjonsmodeller og oppdage mørke biomarkører. Denne protokollen bruker pasientdatasettet for kolonadenokarsinom (COAD) fra The Cancer Genome Atlas (TCGA) database29 som et illustrerende eksempel. I denne sammenheng kategoriseres pasienter i stadium I eller II som negative prøver, mens de i stadium III eller IV regnes som positive prøver. Fordelingen av mørke og tradisjonelle biomarkører på tvers av de 26 TCGA-krefttypene sammenlignes også.
Beskrivelse av HealthModel-pipelinen
Metodikken som benyttes i denne protokollen er basert på det tidligere publiserte rammeverket26, som skissert i figur 1. For å starte, må brukerne klargjøre inndatasettet, mate det inn i den foreslåtte HealthModel-pipelinen og få mqTrans-funksjoner. Detaljerte instruksjoner for dataforberedelse er gitt i avsnitt 2 i protokollseksjonen. Deretter har brukerne muligheten til å kombinere mqTrans-funksjoner med de originale transkriptomiske funksjonene eller bare fortsette med de genererte mqTrans-funksjonene. Det produserte datasettet blir deretter utsatt for en funksjonsvalgsprosess, der brukerne har fleksibilitet til å velge sin foretrukne verdi for k i k-fold kryssvalidering for klassifisering. Den primære evalueringsmålingen som brukes i denne protokollen, er nøyaktighet.
HealthModel26 kategoriserer de transkriptomiske funksjonene i tre forskjellige grupper: TF (transkripsjonsfaktor), lincRNA (langt intergenisk ikke-kodende RNA) og mRNA (messenger RNA). TF-funksjonene er definert basert på merknadene som er tilgjengelige i Human Protein Atlas30,31. Dette arbeidet benytter merknadene til lincRNA fra GTEx-datasettet32. Gener som tilhører tredjenivåbanene i KEGG-databasen33 regnes som mRNA-egenskaper. Det er verdt å merke seg at hvis en mRNA-funksjon viser regulatoriske roller for et målgen som dokumentert i TRRUST-databasen34, blir den omklassifisert til TF-klassen.
Denne protokollen genererer også manuelt de to eksempelfilene for gen-IDene til regulatoriske faktorer (regulatory_geneIDs.csv) og mål-mRNA (target_geneIDs.csv). Den parvise avstandsmatrisen mellom regulatoriske trekk (TF og lincRNA) beregnes av Pearson-korrelasjonskoeffisientene og grupperes av den populære verktøyvektede genkoekspresjonsnettverksanalysen (WGCNA) 36 (adjacent_matrix.csv). Brukere kan bruke HealthModel-pipelinen direkte sammen med disse eksempelkonfigurasjonsfilene for å generere mqTrans-visningen av et transkriptomisk datasett.
Tekniske detaljer om HealthModel
HealthModel representerer de intrikate forholdene mellom TF og lincRNA som en graf, med inngangsfunksjonene som fungerer som hjørnene betegnet med V og en inter-toppunktkantmatrise betegnet som E. Hver prøve er preget av K-regulatoriske egenskaper, symbolisert som VK×1. Spesifikt omfattet datasettet 425 TF og 375 lincRNA, noe som resulterte i en prøvedimensjonalitet på K = 425 + 375 = 800. For å etablere kantmatrisen E brukte dette arbeidet det populære verktøyet WGCNA35. Den parvise vekten som forbinder to hjørner representert som 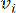 og
og 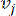 , bestemmes av Pearson-korrelasjonskoeffisienten. Det genregulerende nettverket utviser en skalafri topologi36, preget av tilstedeværelsen av navgener med sentrale funksjonelle roller. Vi beregner korrelasjonen mellom to funksjoner eller toppunkter,
, bestemmes av Pearson-korrelasjonskoeffisienten. Det genregulerende nettverket utviser en skalafri topologi36, preget av tilstedeværelsen av navgener med sentrale funksjonelle roller. Vi beregner korrelasjonen mellom to funksjoner eller toppunkter, 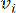 og
og 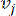 , ved hjelp av det topologiske overlappingsmålet (TOM) som følger:
, ved hjelp av det topologiske overlappingsmålet (TOM) som følger:
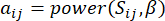 (1)
(1)
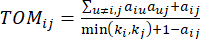 (2)
(2)
Den myke terskelen β beregnes ved hjelp av funksjonen “pickSoft Threshold” fra WGCNA-pakken. Potenseksponentialfunksjonen aij brukes, der 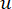 representerer et gen som ekskluderer i og j, og
representerer et gen som ekskluderer i og j, og 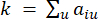 representerer toppunktforbindelsen. WGCNA grupperer uttrykksprofilene til de transkriptomiske trekkene i flere moduler ved hjelp av et vanlig ulikhetsmål (
representerer toppunktforbindelsen. WGCNA grupperer uttrykksprofilene til de transkriptomiske trekkene i flere moduler ved hjelp av et vanlig ulikhetsmål (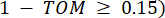 37.
37.
HealthModel-rammeverket ble opprinnelig designet som en multitask læringsarkitektur26. Denne protokollen benytter bare modellens fortreningsoppgave for konstruksjon av den transkriptomiske mqTrans-visningen. Brukeren kan velge å videreutvikle den forhåndstrente HealthModel under multitask graph attention network med flere oppgavespesifikke transkriptomiske prøver.
Tekniske detaljer om funksjonsvalg og klassifisering
Funksjonsutvalget implementerer elleve algoritmer for funksjonsvalg (FS). Blant dem er tre filterbaserte FS-algoritmer: velge K beste funksjoner ved hjelp av maksimal informasjonskoeffisient (SK_mic), velge K-funksjoner basert på FPR for MIC (SK_fpr), og velge K-funksjoner med den høyeste falske oppdagelsesfrekvensen for MIC (SK_fdr). I tillegg vurderer tre trebaserte FS-algoritmer individuelle funksjoner ved hjelp av et beslutningstre med Gini-indeksen (DT_gini), adaptive boosted decision trees (AdaBoost) og random forest (RF_fs). Bassenget inneholder også to wrapper-metoder: Rekursiv funksjonseliminering med lineær støttevektorklassifiserer (RFE_SVC) og eliminering av rekursive funksjoner med logistisk regresjonsklassifiserer (RFE_LR). Til slutt inkluderes to innebyggingsalgoritmer: lineær SVC-klassifiserer med de topprangerte L1-funksjonsviktighetsverdiene (lSVC_L1) og logistisk regresjonsklassifiserer med de topprangerte L1-funksjonsviktighetsverdiene (LR_L1).
Klassifiseringsutvalget bruker syv forskjellige klassifiserere for å bygge klassifiseringsmodeller. Disse klassifisererne omfatter lineær støttevektormaskin (SVC), Gaussian Naïve Bayes (GNB), logistisk regresjonsklassifiserer (LR), k-nærmeste nabo, med k satt til 5 som standard (KNN), XGBoost, tilfeldig skog (RF) og beslutningstre (DT).
Den tilfeldige delingen av datasettet i toget: testundergrupper kan settes i kommandolinjen. Det demonstrerte eksemplet bruker forholdet mellom tog: test = 8: 2.
Protocol
Representative Results
Discussion
Seksjon 2 (Bruk den forhåndstrente HealthModel til å generere mqTrans-funksjonene) i protokollen er det mest kritiske trinnet i denne protokollen. Etter å ha forberedt det beregningsmessige arbeidsmiljøet i seksjon 1, genererer seksjon 2 mqTrans-visningen av et transkriptomisk datasett basert på den forhåndstrente store referansemodellen. Seksjon 3 er et demonstrativt eksempel på å velge de genererte mqTrans-funksjonene for biomarkørdeteksjoner og prediksjonsoppgaver. Brukerne kan utføre andre transkriptomiske …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dette arbeidet ble støttet av Senior and Junior Technological Innovation Team (20210509055RQ), Guizhou Provincial Science and Technology Projects (ZK2023-297), Science and Technology Foundation of Health Commission of Guizhou Province (gzwkj2023-565), Science and Technology Project of Education Department of Jilin Province (JJKH20220245KJ og JJKH20220226SK), National Natural Science Foundation of China (U19A2061), Jilin Provincial Key Laboratory of Big Data Intelligent Computing (20180622002JC), og de grunnleggende forskningsfondene for de sentrale universitetene, JLU. Vi uttrykker vår dypeste takknemlighet til redaktøren og de tre anonyme anmelderne for deres konstruktive kritikk, som har vært medvirkende til å vesentlig forbedre strengheten og klarheten i denne protokollen.
Materials
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
References
- Mutz, K. -. O., Heilkenbrinker, A., Lönne, M., Walter, J. -. G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516 (2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684 (2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352 (2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528 (2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. . Analysis of Survival Data. , (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866 (2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307 (2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119 (2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239 (2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334 (2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338 (2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. . BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , (2022).
- Muzio, G., O’Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738 (2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5 (2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883 (2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582 (2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801 (2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138 (2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308 (2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).

