Küçük Veri Kümelerinde Tahmin Görevi ve Karanlık Biyobelirteç Tespiti için Transkriptomik Özelliklerin Transkripsiyonel Düzenleme Görünümünün Oluşturulması
Summary
Burada, transkriptomik verileri bir mqTrans görünümüne dönüştürmek için karanlık biyobelirteçlerin tanımlanmasını sağlayan bir protokol sunuyoruz. Konvansiyonel transkriptomik analizlerde diferansiyel olarak ifade edilmese de, bu biyobelirteçler mqTrans görünümünde diferansiyel ekspresyon sergiler. Yaklaşım, daha önce gözden kaçan biyobelirteçleri ortaya çıkaran geleneksel yöntemlere tamamlayıcı bir teknik olarak hizmet eder.
Abstract
Transkriptom, bir örnekteki birçok genin ekspresyon seviyelerini temsil eder ve biyolojik araştırmalarda ve klinik uygulamada yaygın olarak kullanılmaktadır. Araştırmacılar genellikle bir fenotip grubu ile bir kontrol grubu örneklem arasında farklı temsillere sahip transkriptomik biyobelirteçlere odaklandılar. Bu çalışma, referans örneklerin karmaşık genler arası etkileşimlerini öğrenmek için çok görevli bir grafik-dikkat ağı (GAT) öğrenme çerçevesi sunmuştur. Bağımsız test transkriptomlarının model tabanlı kantitatif transkripsiyonel düzenleme (mqTrans) görünümünü oluşturmak için doğrudan kullanılabilecek sağlıklı örnekler (HealthModel) üzerinde gösterici bir referans modeli önceden eğitildi. Transkriptomların oluşturulan mqTrans görünümü, tahmin görevleri ve karanlık biyobelirteç tespiti ile gösterildi. Ortaya çıkan “karanlık biyobelirteç” terimi, karanlık bir biyobelirteçin mqTrans görünümünde diferansiyel temsil gösterdiği, ancak orijinal ekspresyon seviyesinde diferansiyel ekspresyon göstermediği tanımından kaynaklanmıştır. Diferansiyel ekspresyonun olmaması nedeniyle geleneksel biyobelirteç tespit çalışmalarında karanlık bir biyobelirteç her zaman göz ardı edilmiştir. HealthModelPipe işlem hattının kaynak kodu ve kılavuzu http://www.healthinformaticslab.org/supp/resources.php’dan indirilebilir.
Introduction
Transkriptom, bir örnekteki tüm genlerin ekspresyonlarından oluşur ve mikrodizi ve RNA-seq1 gibi yüksek verimli teknolojilerle profillenebilir. Bir veri kümesindeki bir genin ekspresyon seviyelerine transkriptomik özellik denir ve fenotip ile kontrol grupları arasındaki bir transkriptomik özelliğin diferansiyel temsili, bu geni bu fenotipin bir biyobelirteci olarak tanımlar 2,3. Transkriptomik biyobelirteçler, hastalık teşhisi4, biyolojik mekanizma5 ve sağkalım analizi 6,7 vb. araştırmalarda yaygın olarak kullanılmaktadır.
Sağlıklı dokulardaki gen aktivite paternleri yaşamlar hakkında önemli bilgiler taşır 8,9. Bu kalıplar paha biçilmez içgörüler sunar ve iyi huylu bozuklukların10,11 ve ölümcül hastalıkların12 karmaşık gelişimsel yörüngelerini anlamak için ideal referanslar olarak hareket eder. Genler birbirleriyle etkileşime girer ve transkriptomlar, karmaşık etkileşimlerinden sonraki son ekspresyon seviyelerini temsil eder. Bu tür kalıplar, transkripsiyonel düzenleme ağı13 ve metabolizma ağı14 vb. olarak formüle edilmiştir. Haberci RNA’ların (mRNA’lar) ekspresyonları, transkripsiyon faktörleri (TF’ler) ve uzun intergenik kodlamayan RNA’lar (lincRNA’lar) tarafından transkripsiyonel olarak düzenlenebilir15,16,17. Konvansiyonel diferansiyel ekspresyon analizi, özellikler arası bağımsızlık varsayımıyla bu tür karmaşık gen etkileşimlerini göz ardı etmiştir18,19.
Grafik sinir ağlarındaki (GNN’ler) son gelişmeler, kanser çalışmaları20 için OMIC tabanlı verilerden önemli bilgilerin çıkarılmasında, örneğin birlikte ekspresyon modüllerinintanımlanmasında 21 olağanüstü bir potansiyel olduğunu göstermektedir. GNN’lerin doğuştan gelen kapasitesi, onları genler arasındaki karmaşık ilişkileri ve bağımlılıkları modellemek için ideal kılar22,23.
Biyomedikal çalışmalar genellikle kontrol grubuna karşı bir fenotipi doğru bir şekilde tahmin etmeye odaklanır. Bu tür görevler genellikle ikili sınıflandırmalar 24,25,26 olarak formüle edilir. Burada, iki sınıf etiketi genellikle 1 ve 0, doğru ve yanlış, hatta pozitif ve negatif27 olarak kodlanır.
Bu çalışma, önceden eğitilmiş grafik-dikkat ağı (GAT) referans modeline dayalı bir transkriptom veri kümesinin transkripsiyonel düzenleme (mqTrans) görünümünü oluşturmak için kullanımı kolay bir protokol sağlamayı amaçlamıştır. Daha önce yayınlanmış bir çalışma26’dan çok görevli GAT çerçevesi, transkriptomik özellikleri mqTrans özelliklerine dönüştürmek için kullanıldı. Düzenleyici faktörlerden (TF’ler ve lincRNA’lar) hedef mRNA’lara transkripsiyon düzenlemelerini kantitatif olarak ölçen referans modeli (HealthModel) önceden eğitmek için Kaliforniya Üniversitesi, Santa Cruz (UCSC) Xena platform28’den sağlıklı transkriptomlardan oluşan geniş bir veri seti kullanıldı. Oluşturulan mqTrans görünümü, tahmin modelleri oluşturmak ve karanlık biyobelirteçleri tespit etmek için kullanılabilir. Bu protokol, açıklayıcı bir örnek olarak Kanser Genom Atlası (TCGA) veritabanı29’dan kolon adenokarsinomu (COAD) hasta veri setini kullanır. Bu bağlamda, evre I veya II’deki hastalar negatif örnekler olarak kategorize edilirken, evre III veya IV’teki hastalar pozitif örnekler olarak kabul edilir. 26 TCGA kanser türü arasında karanlık ve geleneksel biyobelirteçlerin dağılımları da karşılaştırılmıştır.
HealthModel işlem hattının açıklaması
Bu protokolde kullanılan metodoloji, Şekil 1’de belirtildiği gibi daha önce yayınlanmış çerçeve26’ya dayanmaktadır. Başlamak için kullanıcıların giriş veri kümesini hazırlaması, önerilen HealthModel işlem hattına beslemesi ve mqTrans özelliklerini edinmesi gerekir. Ayrıntılı veri hazırlama talimatları protokol bölümünün 2. bölümünde verilmiştir. Daha sonra, kullanıcılar mqTrans özelliklerini orijinal transkriptomik özelliklerle birleştirme veya yalnızca oluşturulan mqTrans özellikleriyle devam etme seçeneğine sahiptir. Üretilen veri kümesi daha sonra bir özellik seçim sürecine tabi tutulur ve kullanıcılar, sınıflandırma için k kat çapraz doğrulamada k için tercih ettikleri değeri seçme esnekliğine sahiptir. Bu protokolde kullanılan birincil değerlendirme ölçütü doğruluktur.
HealthModel26, transkriptomik özellikleri üç farklı gruba ayırır: TF (Transkripsiyon Faktörü), lincRNA (uzun intergenik kodlamayan RNA) ve mRNA (haberci RNA). TF özellikleri, İnsan Protein Atlası30,31’de bulunan ek açıklamalara göre tanımlanır. Bu çalışma, GTEx veri kümesi32’deki lincRNA’ların ek açıklamalarını kullanır. KEGG veri tabanındaki33 üçüncü seviye yollara ait genler, mRNA özellikleri olarak kabul edilir. Bir mRNA özelliği, TRRUST veri tabanında34 belgelendiği gibi bir hedef gen için düzenleyici roller sergiliyorsa, TF sınıfına yeniden sınıflandırıldığını belirtmekte fayda var.
Bu protokol ayrıca düzenleyici faktörlerin (regulatory_geneIDs.csv) ve hedef mRNA’nın (target_geneIDs.csv) gen kimlikleri için iki örnek dosyayı manuel olarak oluşturur. Düzenleyici özellikler (TF’ler ve lincRNA’lar) arasındaki ikili mesafe matrisi, Pearson korelasyon katsayıları ile hesaplanır ve popüler araç ağırlıklı gen ortak ekspresyon ağı analizi (WGCNA)36 (adjacent_matrix.csv) ile kümelenir. Kullanıcılar, bir transkriptomik veri kümesinin mqTrans görünümünü oluşturmak için bu örnek yapılandırma dosyalarıyla birlikte HealthModel işlem hattını doğrudan kullanabilir.
HealthModel’in teknik detayları
HealthModel, TF’ler ve lincRNA’lar arasındaki karmaşık ilişkileri bir grafik olarak temsil eder, girdi özellikleri V ile gösterilen köşeler ve E olarak gösterilen köşeler arası kenar matrisi olarak hizmet eder. Her numune, VK×1 olarak sembolize edilen K düzenleyici özelliklerle karakterize edilir. Spesifik olarak, veri kümesi 425 TF ve 375 lincRNA’yı kapsıyordu ve bu da K = 425 + 375 = 800’lük bir örnek boyutsallığı ile sonuçlandı. Kenar matrisi E’yi oluşturmak için, bu çalışma popüler araç WGCNA35’i kullandı. ve 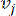 olarak
olarak 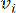 temsil edilen iki köşeyi birbirine bağlayan ikili ağırlık, Pearson korelasyon katsayısı ile belirlenir. Gen düzenleyici ağ, önemli fonksiyonel rollere sahip hub genlerinin varlığı ile karakterize edilen ölçeksiz bir topoloji36 sergiler. İki özellik veya köşe
temsil edilen iki köşeyi birbirine bağlayan ikili ağırlık, Pearson korelasyon katsayısı ile belirlenir. Gen düzenleyici ağ, önemli fonksiyonel rollere sahip hub genlerinin varlığı ile karakterize edilen ölçeksiz bir topoloji36 sergiler. İki özellik veya köşe 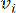 arasındaki korelasyonu ve
arasındaki korelasyonu ve 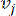 topolojik örtüşme ölçüsünü (TOM) kullanarak aşağıdaki gibi hesaplıyoruz:
topolojik örtüşme ölçüsünü (TOM) kullanarak aşağıdaki gibi hesaplıyoruz:
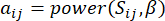 (1)
(1)
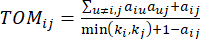 (2)
(2)
Yumuşak eşik β , WGCNA paketinden ‘pickSoft Threshold’ işlevi kullanılarak hesaplanır. Kuvvet üstel fonksiyonu aij uygulanır, burada 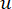 i ve j hariç bir geni temsil eder ve
i ve j hariç bir geni temsil eder ve 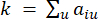 köşe bağlantısını temsil eder. WGCNA, transkriptomik özelliklerin ekspresyon profillerini, yaygın olarak kullanılan bir farklılık ölçüsü kullanarak birden fazla modülde kümeler (
köşe bağlantısını temsil eder. WGCNA, transkriptomik özelliklerin ekspresyon profillerini, yaygın olarak kullanılan bir farklılık ölçüsü kullanarak birden fazla modülde kümeler (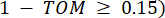 37.
37.
HealthModel çerçevesi başlangıçta çok görevli bir öğrenme mimarisiolarak tasarlanmıştır 26. Bu protokol yalnızca transkriptomik mqTrans görünümünün oluşturulması için model ön eğitim görevini kullanır. Kullanıcı, göreve özgü ek transkriptomik örneklerle çoklu görev grafiği dikkat ağı altında önceden eğitilmiş HealthModel’i daha da iyileştirmeyi seçebilir.
Özellik seçimi ve sınıflandırmasının teknik detayları
Özellik seçim havuzu, on bir özellik seçimi (FS) algoritması uygular. Bunlardan üçü filtre tabanlı FS algoritmalarıdır: Maksimum Bilgi Katsayısını (SK_mic) kullanarak K en iyi özelliklerini seçmek, MIC’nin FPR’sine (SK_fpr) göre K özelliklerini seçmek ve MIC’nin en yüksek yanlış keşif oranına sahip K özelliklerini seçmek (SK_fdr). Ek olarak, üç ağaç tabanlı FS algoritması, Gini indeksi (DT_gini), uyarlanabilir artırılmış karar ağaçları (AdaBoost) ve rastgele orman (RF_fs) içeren bir karar ağacı kullanarak bireysel özellikleri değerlendirir. Havuz ayrıca iki sarmalayıcı yöntemi içerir: Doğrusal destek vektör sınıflandırıcısı (RFE_SVC) ile özyinelemeli özellik eleme ve lojistik regresyon sınıflandırıcısı (RFE_LR) ile özyinelemeli özellik eleme. Son olarak, iki ekleme algoritması dahil edilmiştir: en üst sıradaki L1 özellik önem değerlerine (lSVC_L1) sahip doğrusal SVC sınıflandırıcısı ve en üst sıradaki L1 özellik önem değerlerine (LR_L1) sahip lojistik regresyon sınıflandırıcısı.
Sınıflandırıcı havuzu, sınıflandırma modelleri oluşturmak için yedi farklı sınıflandırıcı kullanır. Bu sınıflandırıcılar, doğrusal destek vektör makinesi (SVC), Gauss Naïve Bayes (GNB), lojistik regresyon sınıflandırıcısı (LR), k-en yakın komşu, k varsayılan olarak 5’e ayarlanmış (KNN), XGBoost, rastgele orman (RF) ve karar ağacından (DT) oluşur.
Veri kümesinin trene rastgele bölünmesi: test alt kümeleri komut satırında ayarlanabilir. Gösterilen örnek, tren: test = 8: 2 oranını kullanır.
Protocol
Representative Results
Discussion
Protokolün 2. bölümü (mqTrans özelliklerini oluşturmak için önceden eğitilmiş HealthModel’i kullanın) bu protokol içindeki en kritik adımdır. Bölüm 1’de hesaplamalı çalışma ortamını hazırladıktan sonra, bölüm 2, önceden eğitilmiş büyük referans modeline dayalı bir transkriptomik veri kümesinin mqTrans görünümünü oluşturur. Bölüm 3, biyobelirteç tespitleri ve tahmin görevleri için oluşturulan mqTrans özelliklerinin seçilmesinin açıklayıcı bir örneğidir. Kullanıcılar, …
Disclosures
The authors have nothing to disclose.
Acknowledgements
Bu çalışma, Kıdemli ve Genç Teknolojik İnovasyon Ekibi (20210509055RQ), Guizhou Eyaleti Bilim ve Teknoloji Projeleri (ZK2023-297), Guizhou Eyaleti Sağlık Komisyonu Bilim ve Teknoloji Vakfı (gzwkj2023-565), Jilin Eyaleti Eğitim Departmanı Bilim ve Teknoloji Projesi (JJKH20220245KJ ve JJKH20220226SK), Çin Ulusal Doğa Bilimleri Vakfı (U19A2061), Jilin Eyaleti Büyük Veri Akıllı Bilgi İşlem Anahtar Laboratuvarı tarafından desteklenmiştir (20180622002JC) ve Merkez Üniversiteler için Temel Araştırma Fonları, JLU. İnceleme editörüne ve üç isimsiz hakeme, bu protokolün titizliğini ve netliğini önemli ölçüde artırmada etkili olan yapıcı eleştirileri için en içten teşekkürlerimizi sunarız.
Materials
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
References
- Mutz, K. -. O., Heilkenbrinker, A., Lönne, M., Walter, J. -. G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516 (2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684 (2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352 (2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528 (2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. . Analysis of Survival Data. , (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866 (2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307 (2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119 (2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239 (2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334 (2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338 (2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. . BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , (2022).
- Muzio, G., O’Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738 (2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5 (2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883 (2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582 (2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801 (2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138 (2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308 (2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).

