Genereren van de transcriptionele regulatieweergave van transcriptomische kenmerken voor voorspellingstaak en detectie van donkere biomarkers op kleine datasets
Summary
Hier introduceren we een protocol voor het converteren van transcriptomische gegevens naar een mqTrans-weergave, waardoor de identificatie van donkere biomarkers mogelijk wordt. Hoewel deze biomarkers niet differentieel tot expressie worden gebracht in conventionele transcriptomische analyses, vertonen ze differentiële expressie in de mqTrans-weergave. De aanpak dient als een aanvullende techniek op traditionele methoden en onthult eerder over het hoofd geziene biomarkers.
Abstract
Transcriptoom vertegenwoordigt de expressieniveaus van veel genen in een monster en wordt veel gebruikt in biologisch onderzoek en de klinische praktijk. Onderzoekers concentreerden zich meestal op transcriptomische biomarkers met differentiële representaties tussen een fenotypegroep en een controlegroep van monsters. Deze studie presenteerde een multitask graph-attention network (GAT) leerkader om de complexe intergene interacties van de referentiemonsters te leren. Een demonstratief referentiemodel werd vooraf getraind op de gezonde monsters (HealthModel), dat direct kon worden gebruikt om de modelgebaseerde kwantitatieve transcriptionele regulatie (mqTrans)-weergave van de onafhankelijke testtranscriptomen te genereren. De gegenereerde mqTrans-weergave van transcriptomen werd gedemonstreerd door voorspellingstaken en detectie van donkere biomarkers. De bedachte term “donkere biomarker” kwam voort uit de definitie dat een donkere biomarker differentiële representatie vertoonde in de mqTrans-weergave, maar geen differentiële expressie in het oorspronkelijke expressieniveau. Een donkere biomarker werd altijd over het hoofd gezien in traditionele biomarkerdetectiestudies vanwege de afwezigheid van differentiële expressie. De broncode en de handleiding van de pipeline HealthModelPipe zijn te downloaden van http://www.healthinformaticslab.org/supp/resources.php.
Introduction
Transcriptoom bestaat uit de expressies van alle genen in een monster en kan worden geprofileerd door high-throughput technologieën zoals microarray en RNA-seq1. De expressieniveaus van één gen in een dataset worden een transcriptomisch kenmerk genoemd, en de differentiële representatie van een transcriptomisch kenmerk tussen het fenotype en de controlegroep definieert dit gen als een biomarker van dit fenotype 2,3. Transcriptomische biomarkers zijn op grote schaal gebruikt bij het onderzoeken van ziektediagnose4, biologisch mechanisme5 en overlevingsanalyse 6,7, enz.
Genactiviteitspatronen in de gezonde weefsels bevatten cruciale informatie over het leven 8,9. Deze patronen bieden inzichten van onschatbare waarde en fungeren als ideale referenties voor het begrijpen van de complexe ontwikkelingstrajecten van goedaardige aandoeningen10,11 en dodelijke ziekten12. Genen interageren met elkaar en transcriptomen vertegenwoordigen de uiteindelijke expressieniveaus na hun gecompliceerde interacties. Dergelijke patronen worden geformuleerd als transcriptioneel regulatienetwerk13 en metabolismenetwerk14, enz. De expressies van boodschapper-RNA’s (mRNA’s) kunnen transcriptioneel worden gereguleerd door transcriptiefactoren (TF’s) en lange intergene niet-coderende RNA’s (lincRNA’s)15,16,17. Conventionele differentiële expressieanalyse negeerde dergelijke complexe geninteracties met de aanname van onafhankelijkheid tussen kenmerken 18,19.
Recente ontwikkelingen op het gebied van grafische neurale netwerken (GNN’s) tonen een buitengewoon potentieel aan bij het extraheren van belangrijke informatie uit OMIC-gebaseerde gegevens voor kankerstudies20, bijvoorbeeld het identificeren van co-expressiemodules21. De aangeboren capaciteit van GNN’s maakt ze ideaal voor het modelleren van de ingewikkelde relaties en afhankelijkheden tussen genen22,23.
Biomedische studies richten zich vaak op het nauwkeurig voorspellen van een fenotype ten opzichte van de controlegroep. Dergelijke taken worden gewoonlijk geformuleerd als binaire classificaties 24,25,26. Hier worden de twee klasselabels meestal gecodeerd als 1 en 0, waar en onwaar, of zelfs positief en negatief27.
Deze studie had tot doel een gebruiksvriendelijk protocol te bieden voor het genereren van de transcriptionele regulatie (mqTrans)-weergave van een transcriptoomdataset op basis van het vooraf getrainde graph-attention network (GAT)-referentiemodel. Het multitask GAT-framework van een eerder gepubliceerd werk26 werd gebruikt om transcriptomische functies te transformeren naar de mqTrans-functies. Een grote dataset van gezonde transcriptomen van het Xena-platform28 van de University of California, Santa Cruz (UCSC) werd gebruikt om het referentiemodel (HealthModel) vooraf te trainen, dat kwantitatief de transcriptievoorschriften van de regulerende factoren (TF’s en lincRNA’s) tot de doel-mRNA’s heeft gemeten. De gegenereerde mqTrans-weergave kan worden gebruikt om voorspellingsmodellen te bouwen en donkere biomarkers te detecteren. Dit protocol maakt gebruik van de patiëntdataset colonadenocarcinoom (COAD) uit de database van The Cancer Genome Atlas (TCGA)29 als illustratief voorbeeld. In deze context worden patiënten in stadium I of II gecategoriseerd als negatieve monsters, terwijl patiënten in stadium III of IV als positieve monsters worden beschouwd. De verdelingen van donkere en traditionele biomarkers over de 26 TCGA-kankertypes worden ook vergeleken.
Beschrijving van de HealthModel-pijplijn
De methodologie die in dit protocol wordt gebruikt, is gebaseerd op het eerder gepubliceerde raamwerk26, zoals geschetst in figuur 1. Om te beginnen moeten gebruikers de invoergegevensset voorbereiden, deze invoeren in de voorgestelde HealthModel-pijplijn en mqTrans-functies verkrijgen. Gedetailleerde instructies voor het opstellen van gegevens zijn te vinden in hoofdstuk 2 van het protocolgedeelte. Vervolgens hebben gebruikers de mogelijkheid om mqTrans-functies te combineren met de originele transcriptomische functies of alleen door te gaan met de gegenereerde mqTrans-functies. De geproduceerde dataset wordt vervolgens onderworpen aan een functieselectieproces, waarbij gebruikers de flexibiliteit hebben om hun voorkeurswaarde voor k te kiezen in k-voudige kruisvalidatie voor classificatie. De primaire evaluatiemaatstaf die in dit protocol wordt gebruikt, is nauwkeurigheid.
HealthModel26 categoriseert de transcriptomische kenmerken in drie verschillende groepen: TF (transcriptiefactor), lincRNA (lang intergeen niet-coderend RNA) en mRNA (boodschapper-RNA). De TF-kenmerken worden gedefinieerd op basis van de annotaties die beschikbaar zijn in de Human Protein Atlas30,31. Dit werk maakt gebruik van de annotaties van lincRNA’s uit de GTEx-dataset32. Genen die behoren tot de routes op het derde niveau in de KEGG-database33 worden beschouwd als mRNA-kenmerken. Het is vermeldenswaard dat als een mRNA-kenmerk regulerende rollen vertoont voor een doelgen, zoals gedocumenteerd in de TRRUST-database34, het opnieuw wordt geclassificeerd in de TF-klasse.
Dit protocol genereert ook handmatig de twee voorbeeldbestanden voor de gen-ID’s van regulerende factoren (regulatory_geneIDs.csv) en doel-mRNA (target_geneIDs.csv). De paarsgewijze afstandsmatrix tussen de regulerende kenmerken (TF’s en lincRNA’s) wordt berekend door de Pearson-correlatiecoëfficiënten en geclusterd door de populaire tool weighted gene co-expression network analysis (WGCNA)36 (adjacent_matrix.csv). Gebruikers kunnen de HealthModel-pijplijn rechtstreeks gebruiken in combinatie met deze voorbeeldconfiguratiebestanden om de mqTrans-weergave van een transcriptomische gegevensset te genereren.
Technische details van HealthModel
HealthModel geeft de ingewikkelde relaties tussen TF’s en lincRNA’s weer als een grafiek, waarbij de invoerfuncties dienen als de hoekpunten die worden aangeduid met V en een intervertex-randmatrix die wordt aangeduid als E. Elk monster wordt gekenmerkt door K-regulerende kenmerken, gesymboliseerd als VK×1. In het bijzonder omvatte de dataset 425 TF’s en 375 lincRNA’s, wat resulteerde in een monsterdimensionaliteit van K = 425 + 375 = 800. Om de randmatrix E vast te stellen, werd voor dit werk gebruik gemaakt van de populaire tool WGCNA35. Het paarsgewijze gewicht dat twee hoekpunten met elkaar verbindt, weergegeven als 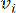 en
en 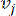 , wordt bepaald door de Pearson-correlatiecoëfficiënt. Het genregulerende netwerk vertoont een schaalvrije topologie36, gekenmerkt door de aanwezigheid van hub-genen met een cruciale functionele rol. We berekenen de correlatie tussen twee objecten of hoekpunten,
, wordt bepaald door de Pearson-correlatiecoëfficiënt. Het genregulerende netwerk vertoont een schaalvrije topologie36, gekenmerkt door de aanwezigheid van hub-genen met een cruciale functionele rol. We berekenen de correlatie tussen twee objecten of hoekpunten, 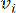 en
en 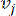 , met behulp van de topologische overlapmaat (TOM) als volgt:
, met behulp van de topologische overlapmaat (TOM) als volgt:
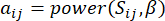 (1)
(1)
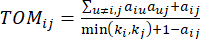 (2)
(2)
De zachte drempel β wordt berekend met behulp van de functie ‘pickSoft Threshold’ uit het WGCNA-pakket. De machtexponentiële functie aij wordt toegepast, waarbij 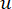 een gen exclusief i en j wordt weergegeven, en
een gen exclusief i en j wordt weergegeven, en 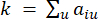 de hoekpuntconnectiviteit wordt weergegeven. WGCNA clustert de expressieprofielen van de transcriptomische kenmerken in meerdere modules met behulp van een veelgebruikte ongelijkheidsmaat (
de hoekpuntconnectiviteit wordt weergegeven. WGCNA clustert de expressieprofielen van de transcriptomische kenmerken in meerdere modules met behulp van een veelgebruikte ongelijkheidsmaat (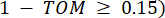 37.
37.
Het HealthModel-raamwerk is oorspronkelijk ontworpen als een multitask-leerarchitectuur26. Dit protocol maakt alleen gebruik van de pre-trainingstaak van het model voor de constructie van de transcriptomische mqTrans-weergave. De gebruiker kan ervoor kiezen om het vooraf getrainde HealthModel verder te verfijnen onder het multitask graph attention network met aanvullende taakspecifieke transcriptomische voorbeelden.
Technische details van de selectie en classificatie van functies
De functieselectiepool implementeert elf algoritmen voor functieselectie (FS). Onder hen zijn er drie op filters gebaseerde FS-algoritmen: het selecteren van de beste kenmerken van K met behulp van de maximale informatiecoëfficiënt (SK_mic), het selecteren van K-kenmerken op basis van de FPR van MIC (SK_fpr) en het selecteren van K-kenmerken met het hoogste percentage valse detectie van MIC (SK_fdr). Daarnaast beoordelen drie op bomen gebaseerde FS-algoritmen individuele kenmerken met behulp van een beslissingsboom met de Gini-index (DT_gini), adaptieve versterkte beslissingsbomen (AdaBoost) en willekeurig bos (RF_fs). De pool bevat ook twee wrapper-methoden: recursieve functie-eliminatie met de lineaire ondersteuningsvectorclassificatie (RFE_SVC) en recursieve functie-eliminatie met de logistische regressieclassificatie (RFE_LR). Ten slotte zijn er twee inbeddingsalgoritmen opgenomen: lineaire SVC-classificatie met de hoogst gerangschikte L1-functiebelangrijkheidswaarden (lSVC_L1) en logistische regressieclassificatie met de hoogst gerangschikte L1-functiebelangrijkheidswaarden (LR_L1).
De classificatiepool maakt gebruik van zeven verschillende classificaties om classificatiemodellen te bouwen. Deze classificaties bestaan uit lineaire ondersteuningsvectormachine (SVC), Gaussiaanse naïeve Bayes (GNB), logistische regressieclassificatie (LR), k-dichtstbijzijnde buur, waarbij k standaard is ingesteld op 5 (KNN), XGBoost, willekeurig bos (RF) en beslissingsboom (DT).
De willekeurige splitsing van de dataset in de trein: testsubsets kunnen worden ingesteld in de opdrachtregel. In het gedemonstreerde voorbeeld wordt de verhouding trein: test = 8: 2 gebruikt.
Protocol
Representative Results
Discussion
Sectie 2 (Gebruik het vooraf getrainde HealthModel om de mqTrans-functies te genereren) van het protocol is de meest kritieke stap binnen dit protocol. Na het voorbereiden van de computationele werkomgeving in sectie 1, genereert sectie 2 de mqTrans-weergave van een transcriptomische dataset op basis van het vooraf getrainde grote referentiemodel. Sectie 3 is een demonstratief voorbeeld van het selecteren van de gegenereerde mqTrans-functies voor biomarkerdetecties en voorspellingstaken. De gebruikers kunnen andere trans…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dit werk werd ondersteund door het Senior en Junior Technological Innovation Team (20210509055RQ), Guizhou Provincial Science and Technology Projects (ZK2023-297), de Science and Technology Foundation of Health Commission van de provincie Guizhou (gzwkj2023-565), Science and Technology Project van het Education Department van de provincie Jilin (JJKH20220245KJ en JJKH20220226SK), de National Natural Science Foundation of China (U19A2061), het Jilin Provincial Key Laboratory of Big Data Intelligent Computing (20180622002JC), en de Fondsen voor Fundamenteel Onderzoek voor de Centrale Universiteiten, JLU. We betuigen onze oprechte waardering aan de review-editor en de drie anonieme reviewers voor hun constructieve kritiek, die een belangrijke rol heeft gespeeld bij het aanzienlijk verbeteren van de nauwkeurigheid en duidelijkheid van dit protocol.
Materials
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
References
- Mutz, K. -. O., Heilkenbrinker, A., Lönne, M., Walter, J. -. G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516 (2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684 (2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352 (2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528 (2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. . Analysis of Survival Data. , (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866 (2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307 (2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119 (2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239 (2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334 (2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338 (2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. . BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , (2022).
- Muzio, G., O’Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738 (2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5 (2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883 (2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582 (2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801 (2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138 (2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308 (2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).

