Génération de la vue de régulation transcriptionnelle des caractéristiques transcriptomiques pour la tâche de prédiction et la détection de biomarqueurs sombres sur de petits ensembles de données
Summary
Ici, nous introduisons un protocole de conversion des données transcriptomiques en une vue mqTrans, permettant l’identification de biomarqueurs sombres. Bien qu’ils ne soient pas exprimés de manière différentielle dans les analyses transcriptomiques conventionnelles, ces biomarqueurs présentent une expression différentielle dans la vue mqTrans. L’approche sert de technique complémentaire aux méthodes traditionnelles, dévoilant des biomarqueurs jusque-là négligés.
Abstract
Le transcriptome représente les niveaux d’expression de nombreux gènes dans un échantillon et a été largement utilisé dans la recherche biologique et la pratique clinique. Les chercheurs se sont généralement concentrés sur les biomarqueurs transcriptomiques avec des représentations différentielles entre un groupe phénotypique et un groupe témoin d’échantillons. Cette étude a présenté un cadre d’apprentissage multitâche graph-attention network (GAT) pour apprendre les interactions intergéniques complexes des échantillons de référence. Un modèle de référence démonstratif a été pré-entraîné sur les échantillons sains (HealthModel), qui a pu être directement utilisé pour générer la vue de régulation transcriptionnelle quantitative basée sur le modèle (mqTrans) des transcriptomes de test indépendants. La vue mqTrans générée des transcriptomes a été démontrée par des tâches de prédiction et la détection de biomarqueurs sombres. Le terme inventé « biomarqueur sombre » découle de sa définition selon laquelle un biomarqueur sombre présentait une représentation différentielle dans la vue mqTrans mais pas d’expression différentielle dans son niveau d’expression d’origine. Un biomarqueur sombre a toujours été négligé dans les études traditionnelles de détection de biomarqueurs en raison de l’absence d’expression différentielle. Le code source et le manuel du pipeline HealthModelPipe peuvent être téléchargés à partir de http://www.healthinformaticslab.org/supp/resources.php.
Introduction
Le transcriptome est constitué de l’expression de tous les gènes d’un échantillon et peut être profilé par des technologies à haut débit telles que les puces à ADN et le séquençage de l’ARN1. Les niveaux d’expression d’un gène dans un ensemble de données sont appelés une caractéristique transcriptomique, et la représentation différentielle d’une caractéristique transcriptomique entre le phénotype et le groupe témoin définit ce gène comme un biomarqueur de ce phénotype 2,3. Les biomarqueurs transcriptomiques ont été largement utilisés dans les enquêtes sur le diagnostic de la maladie4, le mécanisme biologique5 et l’analyse de survie6, 7, etc.
Les modèles d’activité des gènes dans les tissus sains transportent des informations cruciales sur la vie 8,9. Ces modèles offrent des informations précieuses et constituent des références idéales pour comprendre les trajectoires développementales complexes des troubles bénins10,11 et des maladies mortelles12. Les gènes interagissent les uns avec les autres, et les transcriptomes représentent les niveaux d’expression finaux après leurs interactions compliquées. De tels modèles sont formulés comme le réseau de régulation transcriptionnelle13 et le réseau métabolique14, etc. L’expression des ARN messagers (ARNm) peut être régulée transcriptionnellement par des facteurs de transcription (TF) et de longs ARN intergéniques non codants (ARNlinc)15,16,17. L’analyse conventionnelle de l’expression différentielle a ignoré ces interactions complexes entre les gènes avec l’hypothèse de l’indépendance inter-caractéristiques18,19.
Les progrès récents dans les réseaux neuronaux à graphes (GNN) démontrent un potentiel extraordinaire dans l’extraction d’informations importantes à partir de données OMIC pour les études sur le cancer20, par exemple, l’identification de modules de co-expression21. La capacité innée des GNN les rend idéaux pour modéliser les relations et les dépendances complexes entre les gènes22,23.
Les études biomédicales se concentrent souvent sur la prédiction précise d’un phénotype par rapport au groupe témoin. De telles tâches sont généralement formulées sous forme de classifications binaires 24,25,26. Ici, les deux étiquettes de classe sont généralement codées comme 1 et 0, vrai et faux, ou même positif et négatif27.
Cette étude visait à fournir un protocole facile à utiliser pour générer la vue de régulation transcriptionnelle (mqTrans) d’un ensemble de données de transcriptomes basé sur le modèle de référence du réseau d’attention graphique pré-entraîné (GAT). Le cadre GAT multitâche d’un travail précédemment publié26 a été utilisé pour transformer les caractéristiques transcriptomiques en caractéristiques mqTrans. Un grand ensemble de données de transcriptomes sains provenant de la plate-forme Xena28 de l’Université de Californie à Santa Cruz (UCSC) a été utilisé pour pré-entraîner le modèle de référence (HealthModel), qui a mesuré quantitativement les régulations de transcription des facteurs régulateurs (TF et ARNlinc) aux ARNm cibles. La vue mqTrans générée pourrait être utilisée pour construire des modèles de prédiction et détecter des biomarqueurs sombres. Ce protocole utilise l’ensemble de données de patients atteints d’adénocarcinome du côlon (COAD) de la base de données29 de l’Atlas du génome du cancer (TCGA) à titre d’exemple. Dans ce contexte, les patients des stades I ou II sont classés comme des échantillons négatifs, tandis que ceux des stades III ou IV sont considérés comme des échantillons positifs. Les distributions des biomarqueurs sombres et traditionnels dans les 26 types de cancer TCGA sont également comparées.
Description du pipeline HealthModel
La méthodologie employée dans ce protocole est basée sur le cadre26 publié précédemment, comme indiqué à la figure 1. Pour commencer, les utilisateurs doivent préparer le jeu de données d’entrée, l’introduire dans le pipeline HealthModel proposé et obtenir des fonctionnalités mqTrans. Des instructions détaillées pour la préparation des données sont fournies à la section 2 de la section sur le protocole. Par la suite, les utilisateurs ont la possibilité de combiner les caractéristiques mqTrans avec les caractéristiques transcriptomiques d’origine ou de procéder uniquement avec les caractéristiques mqTrans générées. Le jeu de données produit est ensuite soumis à un processus de sélection de caractéristiques, les utilisateurs ayant la possibilité de choisir leur valeur préférée pour k dans la validation croisée k fois pour la classification. La principale mesure d’évaluation utilisée dans ce protocole est la précision.
HealthModel26 classe les caractéristiques transcriptomiques en trois groupes distincts : TF (facteur de transcription), lincRNA (long ARN intergénique non codant) et ARNm (ARN messager). Les caractéristiques TF sont définies sur la base des annotations disponibles dans l’Atlas des protéines humaines30,31. Ce travail utilise les annotations des ARNlinc du jeu de données GTEx32. Les gènes appartenant aux voies de troisième niveau de la base de données KEGG33 sont considérés comme des caractéristiques de l’ARNm. Il convient de noter que si une caractéristique de l’ARNm présente des rôles régulateurs pour un gène cible, comme documenté dans la base de données TRRUST34, elle est reclassée dans la classe TF.
Ce protocole génère également manuellement les deux fichiers d’exemple pour les identifiants de gènes des facteurs de régulation (regulatory_geneIDs.csv) et de l’ARNm cible (target_geneIDs.csv). La matrice de distance par paires entre les caractéristiques régulatrices (TF et ARNlinc) est calculée par les coefficients de corrélation de Pearson et regroupée par l’outil populaire WGCNA (Weighted Geme Co-Expression Network Analysis)36 (adjacent_matrix.csv). Les utilisateurs peuvent utiliser directement le pipeline HealthModel avec ces exemples de fichiers de configuration pour générer la vue mqTrans d’un jeu de données transcriptomique.
Détails techniques de HealthModel
HealthModel représente les relations complexes entre les TF et les lincRNAs sous la forme d’un graphique, les entités en entrée servant de sommets notés V et une matrice d’arêtes inter-sommets désignée par E. Chaque échantillon est caractérisé par des caractéristiques régulatrices K , symbolisées par VK×1. Plus précisément, l’ensemble de données comprenait 425 TF et 375 ARNlinc, ce qui donne une dimensionnalité de l’échantillon de K = 425 + 375 = 800. Pour établir la matrice d’arête E, ce travail a utilisé l’outil populaire WGCNA35. Le poids par paire reliant deux sommets représentés par 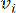 et
et 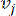 , est déterminé par le coefficient de corrélation de Pearson. Le réseau de régulation des gènes présente une topologie sans échelle36, caractérisée par la présence de gènes pivots ayant des rôles fonctionnels pivots. Nous calculons la corrélation entre deux entités ou sommets,
, est déterminé par le coefficient de corrélation de Pearson. Le réseau de régulation des gènes présente une topologie sans échelle36, caractérisée par la présence de gènes pivots ayant des rôles fonctionnels pivots. Nous calculons la corrélation entre deux entités ou sommets, 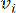 et
et 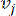 , à l’aide de la mesure de chevauchement topologique (TOM) comme suit :
, à l’aide de la mesure de chevauchement topologique (TOM) comme suit :
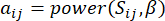 (1)
(1)
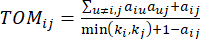 (2)
(2)
Le β de seuil souple est calculé à l’aide de la fonction « pickSoft Threshold » du package WGCNA. La fonction exponentielle de puissance aij est appliquée, où 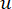 représente un gène excluant i et j, et
représente un gène excluant i et j, et 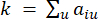 représente la connectivité du sommet. WGCNA regroupe les profils d’expression des caractéristiques transcriptomiques en plusieurs modules à l’aide d’une mesure de dissimilarité couramment utilisée (
représente la connectivité du sommet. WGCNA regroupe les profils d’expression des caractéristiques transcriptomiques en plusieurs modules à l’aide d’une mesure de dissimilarité couramment utilisée (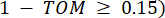 37.
37.
Le cadre HealthModel a été conçu à l’origine comme une architecture d’apprentissage multitâche26. Ce protocole utilise uniquement la tâche de pré-entraînement du modèle pour la construction de la vue transcriptomique mqTrans. L’utilisateur peut choisir d’affiner davantage le HealthModel pré-entraîné dans le cadre du réseau d’attention graphique multitâche avec des échantillons transcriptomiques supplémentaires spécifiques à la tâche.
Détails techniques de la sélection et de la classification des caractéristiques
Le pool de sélection de caractéristiques implémente onze algorithmes de sélection de caractéristiques (FS). Parmi eux, trois sont des algorithmes FS basés sur des filtres : la sélection de K meilleures caractéristiques à l’aide du coefficient d’information maximal (SK_mic), la sélection de K caractéristiques en fonction du FPR de MIC (SK_fpr) et la sélection de K entités avec le taux de fausses découvertes le plus élevé de MIC (SK_fdr). De plus, trois algorithmes FS basés sur des arbres évaluent les caractéristiques individuelles à l’aide d’un arbre de décision avec l’indice de Gini (DT_gini), d’arbres de décision adaptatifs boostés (AdaBoost) et d’une forêt aléatoire (RF_fs). Le pool intègre également deux méthodes d’encapsulation : l’élimination de caractéristiques récursives avec le classificateur de vecteurs de support linéaire (RFE_SVC) et l’élimination de caractéristiques récursives avec le classificateur de régression logistique (RFE_LR). Enfin, deux algorithmes d’incorporation sont inclus : le classificateur SVC linéaire avec les valeurs d’importance des caractéristiques L1 les mieux classées (lSVC_L1) et le classificateur de régression logistique avec les valeurs d’importance des caractéristiques L1 les mieux classées (LR_L1).
Le pool de classificateurs utilise sept classificateurs différents pour créer des modèles de classification. Ces classificateurs comprennent la machine à vecteurs de support linéaire (SVC), le bayésien naïf gaussien (GNB), le classificateur de régression logistique (LR), le k plus proche voisin, avec k défini sur 5 par défaut (KNN), XGBoost, la forêt aléatoire (RF) et l’arbre de décision (DT).
La division aléatoire du jeu de données en sous-ensembles train : test peut être définie dans la ligne de commande. L’exemple illustré utilise le rapport de train : test = 8 : 2.
Protocol
Representative Results
Discussion
La section 2 (Utiliser le HealthModel pré-entraîné pour générer les fonctionnalités mqTrans) du protocole est l’étape la plus critique de ce protocole. Après avoir préparé l’environnement de travail de calcul de la section 1, la section 2 génère la vue mqTrans d’un jeu de données transcriptomiques basé sur le modèle de référence de grande taille pré-entraîné. La section 3 est un exemple démonstratif de sélection des caractéristiques mqTrans générées pour les tâches de détection et de pr?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ce travail a été soutenu par l’équipe d’innovation technologique senior et junior (20210509055RQ), les projets scientifiques et technologiques de la province du Guizhou (ZK2023-297), la Fondation pour la science et la technologie de la Commission de la santé de la province du Guizhou (gzwkj2023-565), le projet scientifique et technologique du Département de l’éducation de la province du Jilin (JJKH20220245KJ et JJKH20220226SK), la Fondation nationale des sciences naturelles de Chine (U19A2061), le Laboratoire clé de la province du Jilin pour l’informatique intelligente des mégadonnées (20180622002JC) et le Fonds de recherche fondamentale pour les universités centrales, JLU. Nous adressons notre plus sincère gratitude à l’éditeur de la revue et aux trois examinateurs anonymes pour leurs critiques constructives, qui ont contribué à améliorer considérablement la rigueur et la clarté de ce protocole.
Materials
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
References
- Mutz, K. -. O., Heilkenbrinker, A., Lönne, M., Walter, J. -. G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516 (2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684 (2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352 (2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528 (2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. . Analysis of Survival Data. , (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866 (2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307 (2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119 (2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239 (2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334 (2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338 (2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. . BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , (2022).
- Muzio, G., O’Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738 (2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5 (2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883 (2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582 (2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801 (2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138 (2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308 (2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).

