Summary
Här beskriver vi en enkel och tillgänglig strategi för visualisering, kvantifiering och kartläggning av immunceller i formalin-fast paraffin-inbäddade tumör vävnad sektioner. Denna metod kombinerar befintliga bild- och digitalanalystekniker i syfte att utöka multiplexeringskapaciteten och multiparameteranalysen av bildanalyser.
Abstract
Immunlandskapet i tumörmikromiljön (TME) är en avgörande faktor för cancerprogression och svar på terapi. Specifikt har densiteten och placeringen av immunceller i TME viktiga diagnostiska och prognostiska värden. Multiomic profilering av TME har exponentiellt ökat vår förståelse av de många cellulära och molekylära nätverk som reglerar tumör initiering och progression. Dessa tekniker ger dock inte information om den rumsliga organisationen av celler eller cellcellinteraktioner. Prisvärd, tillgänglig och lätt att utföra multiplexeringstekniker som tillåter rumslig upplösning av immunceller i vävnadssektioner behövs för att komplettera encellbaserade teknik med hög genomströmning. Här beskriver vi en strategi som integrerar seriell avbildning, sekventiell märkning och bildjustering för att generera virtuella multiparameterbilder av hela vävnadssektioner. Virtuella bilder analyseras därefter på ett automatiserat sätt med hjälp av användardefinierade protokoll som möjliggör identifiering, kvantifiering och mappning av cellpopulationer av intresse. Bildanalysen görs, i detta fall med hjälp av analysmodulerna Tissuealign, Author och HISTOmap. Vi presenterar ett exempel där vi tillämpat denna strategi framgångsrikt på ett kliniskt prov, maximera den information som kan erhållas från begränsade vävnadsprover och ge en opartisk bild av TME i hela vävnaden avsnitt.
Introduction
Cancer utveckling är resultatet av en multistep process som innebär ömsesidiga interaktioner mellan maligna celler och TME. Andra än tumörceller, TME består av nonmalignant celler, stromal celler, immun cell populationer, och extracellulär matris (ECM)1. Den rumsliga organisationen av de olika cellulära och strukturella komponenterna i tumörvävnaden och det dynamiska utbytet mellan cancer och angränsande icke-cancerceller modulerar i slutändan tumörprogression och svar på terapi2,3,4. Det har visat sig att immunsvaret i cancer är spatiotemporally regleras5,6. Olika immuncellpopulationer som infiltrerar neoplastiska lesionen och den intilliggande vävnaden uppvisar distinkta rumsliga fördelningsmönster och varierande aktiverings- och differentieringstillstånd som är associerade med olika funktioner (t.ex. pro- kontra antitumor). Dessa olika immun populationer och deras parametrar coevolve övertid med tumör och stromal fack.
Framväxten av teknik som tillåter single cell multiomics profilering har exponentiellt ökat vår förståelse av de många cellulära och molekylära nätverk som reglerar carcinogenes och tumör progression. De flesta enskilda cellbaserade analysverktyg med hög genomströmning kräver dock vävnadsstörningar och isolering av en cell, vilket resulterar i förlust av information om den rumsliga organisationen av celler och cellcellinteraktioner7. Eftersom placeringen och arrangemanget av specifika immunceller i TME har diagnostiska och prognostiska värde, teknik som möjliggör rumslig upplösning är ett viktigt komplement till encelliga immun profilering tekniker.
Traditionellt har bildframställningstekniker som immunohistochemistry (IHC) och multipleximmunfluorescens (mIF) begränsats till ett litet antal biomarkörer som kan visualiseras samtidigt. Denna begränsning har hämmat studien av spatiotemporal dynamiken i tumör-infiltrera immunceller, som vanligtvis definieras av flera fenotypiska markörer. De senaste framstegen inom bildbehandling och analysverktyg har utökat möjligheterna till multiplexering. Nya antikroppsbaserade märkningstekniker som histo-cytometri och avbildningsmassacytometri har använts för att rumsligt separera upp till 12 respektive 32 biomarkörer,respektive 8,9. Masspektrometri imaging, en teknik som inte kräver märkning, har potential att avbilda tusentals biomarkörer samtidigt i en enda vävnad avsnitt10,11. Även om dessa tekniker har redan visat stor potential för att dissekera vävnaden immun landskapet i cancer, de använder mycket sofistikerad och dyr utrustning och programvara och är inte lätt tillgängliga för de flesta forskare.
Alternativt har multiplexeringskapaciteten hos traditionella IHC och mIF utökats med hjälp av seriell avbildning, sekventiella avikappningsrundor och spektralavbildning7,,12,13,14,15,16. Dessa tekniker genererar flera bilder från samma eller från seriella vävnadssektioner som kan konsolideras i virtuella multiparameterbilder med hjälp av bildanalysprogramvara. Därför ökar antalet markörer som kan visualiseras och analyseras samtidigt.
Här föreslår vi en strategi för rationell design av vävnad multiplex analyser med kommersiellt tillgängliga reagenser, prisvärd mikroskopi utrustning, och användarvänlig programvara(figur 1). Denna metod integrerar seriell avbildning, sekventiell multiplex märkning, hela vävnadsavbildning och vävnadsjustering för att generera virtuella multiparameter diabilder som kan användas för automatiserad kvantifiering och kartläggning av immunceller i vävnadssektioner. Med hjälp av denna strategi skapade vi en virtuell bild bestående av 11 biomarkörer plus två ofta använda histologiska fläckar: hematoxylin och eosin (H&E) och picrosirius red (PSR). Flera immun cell populationer identifierades, ligger och kvantifieras i olika vävnadsutrymmen och deras rumsliga fördelningen lösas med hjälp av vävnad heatmaps. Denna strategi maximerar den information som kan erhållas från begränsade kliniska prover och är tillämplig på formalin-fast paraffin-inbäddade (FFPE) arkiverade vävnadsprover, inklusive hela vävnad, core nål tarmbiopsier och vävnad mikroarrayer. Vi föreslår denna metod som en användbar guide för att utforma anpassade analyser för identifiering, kvantifiering och kartläggning av immuncellpopulationer i TME.
Protocol
Tre seriella FFPE sektioner från resected hepatit B-virus (HBV)-associerade mänskliga hepatocellular carcinom erhölls från Centre hospitalier de l'Université de Montréal (CHUM) Hepatopancreatobiliary Cancer Clinical Database och Biological Specimen Databas (HBP Biobank). Patienter som deltog i denna vävnadsbank gav informerat samtycke. Denna studie godkändes av den institutionella etikkommittén (protokoll nummer 09.237) och genomfördes i enlighet med Helsingforsdeklarationen.
1. Infärgningsprotokoll för Hematoxylin och eosin (H&E)
OBS: H & E färgning utfördes av molekylära patologi kärnan anläggningen i Centre de Recherches du Centre hospitalier de l'Université de Montréal (CRCHUM) med hjälp av Shandon multiprogram robotic slide stainer med hjälp av följande program.
- För deparaffinering, sänk ned diabilder 3x för 2,5 min vardera i xylenersättning.
VARNING: Xylensubstitut är brandfarliga, hudr irriterande och skadliga vid inandning. - För rehydrering, sänk ned diabilder i 100% etanol 3x i 2,5 min vardera. Tvätta i 1 min i dubbeldestillerat vatten (ddH2O) för att rehydrera.
- Inkubera i 1 min i hematoxylin. Tvätta 3x i 1 min vardera i ddH2O.
- Inkubera i 5 s med eosin. Tvätta 30 s med 95% etanol. Tvätta 2x i 1 min med 100% etanol.
VARNING: Etanol är brandfarligt och ett ögonr irriterande. Eosin är ett ögonirriterande. - För uttorkning, sänk 3x i 1,5 min vardera i xylensubstitut. Montera bilderna manuellt.
Den beräknade tiden för att köra den här delen av protokollet är 30 min.
2. Multiplex immunofluorescens färgning protokoll för FFPE sektioner
OBS: Detta protokoll har anpassats från Robertson et al.17.
- Deparaffinering och rehydrering
OBS: Före antikroppsmedied märkning av FFPE-sektioner med IHC eller mIF ska paraffinet tas bort. Underlåtenhet att effektivt ta bort paraffin resulterar i suboptimal färgning.- Placera 4 μm FFPE vävnadssektionen glider in i glasglashållare. Sänk ned rutschkanan under rökhuven i en Coplinburk som innehåller 37°C förvärmt xylen i 10 min.
VARNING: Xylen är brandfarligt, irriterande i huden och skadlig vid inandning. - Omröra bilderna manuellt i 10 s varannan minut. Upprepa 1x i färsk xylen i ytterligare 5 min.
- Sänk ned rutschbanorna sekventiellt i 5 min i var och en av följande lösningar: 1) xylen: etanol (1:1 v/v); 2) 100% etanol; 3) 70% etanol; 4) 50% etanol; 5) 30% etanol; 6) fosfatbuffrad koksaltlösning (PBS).
Håll bilderna i PBS tills de är redo att utföra antigenhämtningen. Håll devaxerade sektionerna hydrerade hela tiden. Uttorkning kommer att orsaka ospecifik antikroppsbindning och därmed hög bakgrundsfärgning.
- Placera 4 μm FFPE vävnadssektionen glider in i glasglashållare. Sänk ned rutschkanan under rökhuven i en Coplinburk som innehåller 37°C förvärmt xylen i 10 min.
- Värmeinducerad antigenhämtning
OBS: Antigener kan maskeras vid formalin-fixering, förhindra antikroppsbindning och därmed visualisering. Användningen av antigen avslöja buffertar och förfaranden delvis återupprätta den inhemska konformation av epitopes och därmed återställer antikroppar erkännande. Typen av antigenhämtningsbuffert och varaktighet bör optimeras för de specifika analysförhållandena (t.ex. mål, antikropp, vävnad osv.).- Sänk ned dewaxed diabilder i en Coplin burk som innehåller antigen hämtningslösningen (recept i Tabell av material).
- Placera den stängda Coplinburken i en elektrisk tryckkokare med kranvatten. Vattennivån bör inte överstiga halva burkens höjd så att vattnet inte blandas med antigenhämtningslösningen.
- Stäng locket och tryckventilen på spisen. Välj högt tryck i 10 min och börja. När du är klar, koppla ur spisen, släpp trycket, öppna locket och håll burken inne i spisen i 30 min, så att bilderna svalna.
- Blockering av ospecifik bindning
- Överför racket med rutschbanorna till en Coplin burk fylld med PBS. Skölj bort antigenhämtningsbufferten med PBS 2x i 5 min vardera.
- Omringa vävnadssektionerna med en PAP-penna för att skapa en hydrofoba barriär. Sänk ned rutschbanorna i en Coplin burk som innehåller 0,1 M glycin i PBS. Inkubera i 15 min vid rumstemperatur (RT).
OBS: Glycin mättar aldehyd grupper som genereras under antigen hämtning. Dessa grupper kan binda primära och sekundära antikroppar unspecifically. - Skölj av glycinlösningen genom att tvätta 2x med PBS i 5 min. Placera bilderna i en fuktighetskammare och tillsätt tillräckligt med blockeringslösning för att täcka alla vävnadssektioner. Undvik att svämma över den hydrofoba barriären. Inkubera i 30 min på RT.
OBS: Receptet för blockeringslösningen finns i tabellen över material. Blockeringslösningen bör innehålla ett protein (t.ex. Det kan också innehålla rengöringsmedel som Triton X-100 eller Tween 20 som minskar hydrofoba interaktioner mellan antikroppar och vävnadsmål, vilket gör antigen erkännande mer selektiv. Tillsatsen av 10% totalt serum från de arter där vävnaden kommer ifrån skulle blockera Fc-receptorer, och därmed minska ospecificerad antikroppsbindning. Slutligen, tillägg av 10% av serum från arten sekundära antikroppar höjdes i skulle minimera direkt ospecifik fastsättning av sekundära antikroppar till vävnaden avsnitt.
- Märkning av immunofluorescens
- Skölj med PBS-Tween (0,1% v/v) 2x i 5 min styck och placera tillbaka rutschbanorna i fuktighetskammaren.
- Tillsätt cocktail av primära antikroppar återuppstagd i blockerande lösning. Inkubera över natten vid 4 °C.Inkubera overnight at 4 °C. Primära och sekundära antikroppar som används för denna studie anges i Tabell över material.
OBS: Cocktailen av primära antikroppar bör innehålla antingen antikroppar som föds upp hos olika arter eller från samma art men av olika isotyper. För en förteckning över de primär-sekundära antikroppspar som används i denna studie, se tabell 2. Uppgifter om alla antikroppar som används finns i tabellen över material och tabell 2. - Skölj med PBS-Tween (0,1% v/v) 3x i 5 min och placera tillbaka bilderna i fuktighetskammaren. I mörkret, tillsätt cocktail av sekundära antikroppar och inkubera i 1 h vid RT.
OBS: När de primära antikropparna kommer från olika arter bör de sekundära antikropparna väljas så att var och en av dem endast binder till en av de primära antikropparna och inte till varandra. Detta uppnås vanligen genom att använda sekundära antikroppar som alla tas upp i samma art så länge denna art skiljer sig från de arter där de primära antikropparna genererades. I de fall där de primära antikropparna har höjts i samma art men har olika isotyper bör isotypspecifika sekundära antikroppar användas. - Skölj med PBS-Tween (0,1% v/v) 3x i 5 min styck. Skölj med ddH2O. Avlägsna överflödig vätska och montera i monteringsmedia med DAPI. Vilken volym som används beror på avsnittets storlek. Vanligtvis räcker 40 μL för att täcka ytan på en vanlig mikroskopibild.
- Placera täcket på sektionen och tryck försiktigt ut överflödigt monteringsmedlande för att undvika bubbelbildning. Låt rutschbanorna torka i 20 min vid RT i mörker och förvara i 4 °C tills de är klara för förvärv.
- Hämta bilder för alla kanaler med hela bildskannern (se Tabell över material).
OBS: Antikropparna validerades med hjälp av mänskliga hepatocellular carcinom vävnad som en positiv kontroll. För varje primär antikropp färgades tre seriella sektioner med antingen primär antikropp, isotypkontroll eller endast blockerande lösning utan någon variation i resten av färgningsprotokollet. De förvärvade bilderna jämfördes för att fastställa specificiteten hos färgningen. Färgning ansågs specifik när signalen i avsnittet ruvade med primära antikroppar hade det förväntade mönstret och var lätt att skilja från bakgrunden. Primära antikroppar som ger en hög bakgrundssignal eller märkning vävnadskomponenter i isotyp och inga primära antikroppar avsnitt ansågs ospecificerad. Den beräknade tiden för att slutföra den här delen av protokollet är 2 dagar. De kontroller som krävs omfattar: (1) Isotype-kontroll för att fastställa bidraget från ospecificerad bindning av den primära antikroppen till bakgrundssignalen. Den ena delen färgas på samma sätt som de andra provvävnaderna, förutom att den inkuberas med en antikropp med samma isotyp och ursprung för den primära antikroppen men som är specifik för ett mål som saknas i vävnadssektionen. Om lämplig antikropp för isotypkontroll inte är tillgänglig kan den ersättas med total IgG från samma art där den primära antikroppen har upphöjts. (2) Ingen primär antikroppskontroll (dvs. negativ kontroll) för att fastställa färgningens specificitet och för att uppskatta bidraget från ospecificerad bindning av sekundära antikroppar till bakgrundssignalen. I detta fall färgas kontrollsektionen på samma sätt som de andra sektionerna förutom att ingen primär antikropp tillsätts. (3) Positiv kontroll för att fastställa att färgning fungerar. I detta fall utförs färgningen på en vävnadssektion som är känd för att uttrycka den markör som känns igen av den primära antikroppen.
3. Picro-sirius röd (PSR) / snabb grön färgning protokoll
OBS: Målet med denna färgning är att visualisera fibrillar kollagen I och III i FFPE vävnad sektioner. Detta protokoll har anpassats från Segnani et al.18. Alla steg utförs i en kemisk huva.
- Utför deparaffinering och rehydrering av vävnadssektioner som liknar multipleximmunfluorescensfärgningsprotokollet för FFPE-sektionerna (avsnitt 2.1).
OBS: Om det avsnitt som ska färgas tidigare har använts för immunofluorescensmärkning och paraffinet redan har tagits bort, är deparaffineringsrehydreringsstegen användbara för att ta bort monteringsmediet. DAPI tas inte bort med denna procedur, men det inte märkbart störa PSR färgning. - Sänk ned rutschbanorna i en burk som innehåller picro-sirius röd/snabb grön lösning (recept i bord av material)och inkubera i 30 min vid RT (mer än 30 min resulterar i ospecifik färgning av kärnorna av hepatocyter).
- Tvätta bilderna snabbt i ddH2O (5 dips). Tvätta sedan snabbt i etanol 100% (5 dips). Tvätta i 30 s i xylen-100% etanol (1:1 v/v). Tvätta i 30 s i xylen. Montera med monteringsmedia (se Tabell över material) innan xylen har helt avdunstat (detta hjälper till med monteringen).
Obs: Den beräknade tiden för att utföra denna del av protokollet är 1 h.
4. Elution av antikroppar från vävnadssektioner
OBS: För att återanvända vävnadssektioner i sekventiella märkningsanalyser krävs fullständigt avlägsnande av primära och sekundära antikroppar. Bundna antikroppar togs bort enligt tidigare beskrivna13.
Värm ett vattenbad till 56 °C. Placera sektionerna inuti en burk som innehåller strippning buffert (recept i Tabell of Materials),stäng locket, och försegla den med paraffin film tejp för att förhindra läckage under skakningar.
- Lägg burken i vattenbadet och inkubera i 30 min med omrörning.
- Tvätta 4x i 15 min vardera i DDH2O vid RT. Skölj med PBS-Tween (0,1% v/v).
- Håll sektionerna hydrerade i PBS-Tween eller vatten tills de är redo att ombeställa sektionen med den andra omgången av primära antikroppar.
Obs: Den beräknade tiden för att utföra denna del av protokollet är 2 h. - Kontrollera effektiviteten i antikroppsutellitförfarandet.
OBS: Innan protokollet för antikroppsutellit används i en sekventiell märkningsanalys bör effektiviteten i avlägsnandet av primära och sekundära antikroppar kontrolleras.- Utför färgning och bildinsamling av ett avsnitt med ett givet primär-sekundärt antikroppspar av intresse enligt vad som anges i multipleximmunfluoresfärgningsprotokollet för FFPE-sektioner (avsnitten 2.1–2.4.6).
- Vid bildinsamling utför elueringen av vävnadsbundna primär-sekundära antikroppskomplex enligt avsnitten 4.1–4.3.
- Inkubera sektionen med samma sekundära antikropp och samma tillstånd som används i steg 2.4.3.
- Utför tvätt-, monterings- och bildförvärvsteg enligt 2.4.4–2.4.6.
- Jämför sida vid sida bilder som förvärvats före och efter strippningen för att fastställa om den specifika signalen har försvunnit.
OBS: Jämförelse av bilder före och efter borttagning av antikroppar kommer att validera elueringsprocedurens effektivitet. Det är dock normalt att se en ökning av bakgrundssignalen i alla kanaler, samt spridning av DAPI. Detta begränsar antalet avrundor av strippning som kan utföras på samma vävnadssektion. Tre omgångar strippning verkar vara det maximala.
5. Bild förvärv
- Generera bilder med hjälp av en hel bildskanner.
- Använd en 20x 0.75NA objektiv och en upplösning på 0,3225 μm /pixel.
6. Bildanalys
Den metod som beskrivs här refererar till det aktuella exemplet. Se tabell 1 och texten för att anpassa sig till andra specifika prover.
- Utför vävnadsjustering med hjälp av Tissualign-modulen i bildanalysprogramvaran (VIS i det här protokollet, se Tabell över material).
- Öppna bildanalysprogramvaran och klicka på fliken Tissuealign.
- Importera bilderna som ska justeras till bildfältet genom att gå till Arkiv | databas och markera den första bilden som ska justeras. Gå tillbaka till fliken Tissuealign och ladda bilden genom att klicka på knappen Ladda i bildfältet. Bilden visas i bildfältet och på arbetsytan.
Endast den räntestapel som ska laddas i skjutfältet. - Upprepa steg 6.1.2 för alla bilder i den ordning som ska justeras, ladda dem en efter en. När alla bilder av intresse har lästs in på bildfältet fortsätter du att länka bilderna genom att trycka på Nästa i arbetsflödesstegen i menyfliksområdet.
- Dra sedan och släpp den andra bilden ovanpå den första bilden. Den första och andra bilderna är nu länkade. Upprepa det här steget för att de andra bilderna ska justeras, en efter en, på ett ordnat sätt. Namnet på den första bilden ändras, vilket indikerar att den har länkats till de andra bilderna. Samtidigt visas de länkade bilderna på arbetsytan till höger om bildfältet.
- Justera bilderna antingen med automatisk justering, halvautomatisk justering eller manuell justering. Det är alltid att föredra att prova automatisk justering först. Om du vill ha automatisk justering trycker du på knappen Nästa i arbetsflödesstegen (steg 3) i menyfliksområdet.
- Granska den automatiska justeringen genom att navigera på olika platser i vävnaden och visuellt kontrollera att motsvarande strukturer i olika bilder är ordnade på samma sätt i bildens två dimensioner.
- Om resultatet av den automatiska justeringen inte är tillfredsställande, förbättra den med hjälp av stift (använd minst tre stift per bild) som anger homologa vävnadsfunktioner i de länkade bilderna. När stiften är placerade på homologa platser i de länkade bilderna, har användaren två val: halvautomatisk justering eller manuell justering. För halvautomatisk justering klicka på knappen Autojustera baserat på de aktuella preciserna i menyfliksområdet. Om du vill justera manuellt klickar du på knappen Använd pins i menyfliksområdet.
- När du är nöjd med justeringen klickar du på knappen Nästa i arbetsflödesstegen och sparar den sammansatta bilden i databasen.
OBS: Justera sex bilder som spänner över 11 markörer plus H & E och PSR bilder tog 15 min i analysen presenteras.
- Utför vävnadsdetektering med det användardefinierade protokollanalysprotokollet paket 1 (APP 1, tabell 1).
- Öppna modulen Bildanalys för programvaran genom att klicka på fliken Bildanalys i menyfliksområdet.
- Importera den sammansatta (justerade) bilden genom att gå till Arkiv | Databas och välja bilden av intresse och klicka tillbaka fliken Bildanalys.
- Öppna appvalsdialogrutan genom att klicka på ikonen Öppna APP och välj vilket PROGRAM (Analysis Protocol Package) som ska användas. I detta fall väljer DU APP 1 för vävnadsdetektering.
- När APP 1 har öppnats, bekräfta att APP1 fungerar korrekt genom att gå till en vald vävnadsplats och klicka på förhandsgranskningsknappen. Om resultaten är tillfredsställande går du vidare till nästa steg.
- Klicka här om du vill köra APP 1 och bearbeta avbildningen med den markerade APPEN.
- Exportera data (t.ex. bilder, mått etc.) när analysen görs genom att klicka på Arkiv/Export.
OBS: APP 1 skapar en region av intresse (ROI) som beskriver vävnaden (ROI Tissue) och beräknar vävnadsområdet. - Spara den ändrade bilden med den nyskapade avkastningen genom att gå till Arkiv | Spara.
OBS: Upptäcka vävnaden och skapa en ROI med APP 1 i det medföljande exemplet tog 5 min i bilden analysstationen beskrivs. Området av vävnaden bearbetas var 3,2 cm2.
- Utför vävnadssegmentering i Stroma och Parenchyma med APP 2 (tabell 1).
OBS: APP 2 fungerar på den fördefinierade ROI-vävnaden. APP 2 segmenterar vävnaden i ROIs Stroma och Parenchyma.- Öppna modulen Bildanalys genom att klicka på fliken Bildanalys i menyfliksområdet.
- Importera bilden som innehåller ROI-vävnaden genom att gå till Arkiv | Databas och välja den bild som sparats i steg 6.2.7. Gå tillbaka till fliken Bildanalys och ladda bilden genom att klicka på knappen Ladda i bildfältet. Bilden visas i bildfältet och på arbetsytan.
- Öppna APP 2 med appvalsdialogrutan som i 6.2.3.
- Förhandsgranska APP 2 genom att bearbeta i ett markerat synfält. Om resultaten är tillfredsställande kör du APP 2 på hela bilden genom att klicka på knappen Kör. Som produktionen av APP 2 segmenteras ROI-vävnaden i ROIs Stroma och Parenchyma och deras respektive områden bestäms. Exportresultat som i 6.2.6. Spara den ändrade bilden som i 6.2.7.
OBS: Segmentering vävnaden i Stroma och Parenchyma med HJÄLP AV APP 2 tog 4 h i analysen stationen presenteras. Området av vävnaden bearbetas var 3,2 cm2.
- Identifiera och kvantifiera FoxP3hiCD4+-celler med hjälp av det användardefinierade protokollet APP 3 (Tabell 1).
OBS: APP 3 fungerar på fördefinierade ROIs Stroma och Parenchyma.- Öppna modulen Bildanalys och importera bilden som innehåller ROIs Stroma och Parenchyma som i 6.3.1 och 6.3.2. Öppna APP 3 med appvalsdialogrutan som i 6.2.3.
- Förhandsgranska APP 3-bearbetning i ett valt synfält berikat i FoxP3hiCD4+-celler. Om resultaten är tillfredsställande kör du APP 3 på hela bilden. Som utdata från APP 3, alla enskilda FoxP3hiCD4 + objekt kommer att märkas och deras vävnad koordinater lagras. Tätheter av FoxP3hiCD4 + objekt i ROIs Stroma och Parenchyma kommer att fastställas. Exportera resultaten som i 6.2.6.
- Utför vävnadsvärmemappning av FoxP3hiCD4+ märkta objekt.
- Öppna det användardefinierade protokollet FoxP3hiCD4+ MAP med appvalsdialogrutan som i 6.2.3.
OBS: FoxP3hiCD4 + MAP använder koordinaterna för FoxP3hiCD4 + märkta objekt för att generera densitet heatmaps. Identifiera och räkna FoxP3hejCD4 + märkta objekt med HJÄLP AV APP 3 tog 25 min i bilden analysstationen beskrivs. Området av vävnaden bearbetas var 3,2 cm2. - Kör FoxP3hiCD4+ MAP genom att trycka på körknappen. Exportera vävnadsvärmekartan genom att klicka på Arkiv | Export | arbetsområde.
OBS: Kartläggning FoxP3hejCD4 + märkta objekt med FoxP3hiCD4 + MAP tog 5 min i bilden analysstationen beskrivs.
- Öppna det användardefinierade protokollet FoxP3hiCD4+ MAP med appvalsdialogrutan som i 6.2.3.
- Identifiera och kvantifiera CD8+, CD68+, MPO+, αSMA och CD34 + objekt med hjälp av de användardefinierade protokollen APP 4, APP5, APP6, APP7 respektive APP 8 (Tabell 1) enligt avsnitt 6.4 till 6.4.3.2 och läsa in appen av intresse i varje enskilt fall.
OBS: APPs 4 till 8 arbete på fördefinierade ROIs Stroma och Parenchyma.
Representative Results
Översikt över strategin för visualisering, kvantifiering och kartläggning av cellpopulationer av intresse för TME
För att kvantifiera cellpopulationer av intresse (COI) i olika vävnadsfack (TCs) och för att karakterisera deras rumsliga organisation, utformade vi ett arbetsflöde som integrerar prisvärda och lättanvända tekniker och maximerar den positionsinformation som kan erhållas från dyrbara FFPE kliniska exemplar (figur 1). För det första färgades seriella hela vävnads-FFPE-sektioner för visualisering av COI (t.ex. immunceller) och TC (t.ex. stroma kontra parenkym) (figur 1, steg 1). Antalet på varandra följande sektioner som ska färgas bör hållas till det minimum som gör det möjligt visualisering av celler av intresse eller vävnadsfunktioner som behövs för att ta itu med forskningsfrågan. Ju mindre antal seriesektioner, desto högre vävnadsarkitektur likhet och konkordans över sammanhängande sektioner. Dessutom kan multiplexering kapacitet utökas genom återanvändning av fluorescerande färgade sektioner genom strippning och avbruten teknik19.
När färgning stegen gjordes, en hel bild scanner användes för att digitalisera bilderna. Bilder som förvärvats från seriella avsnitt justerades och konsoliderades till en virtuell multiplexbild på ett automatiserat sätt(figur 1, avsnitt 2). Därefter avgränsades en roi för vävnaden med ett användardefinierat protokoll som identifierade vävnadsassocierade pixlar (TAPs) (figur 1, steg 3). Därefter segmenterades ROI-vävnaden i TV-skivor som definieras som ytterligare avkastning på sysselsatt kapital. (Figur 1, steg 4). Därefter har användardefinierade protokoll upptäckts och kvantifierats cois i olika TC(figur 1, steg 5). Slutligen genererades vävnadsvärmekartor av COI baserat på deras densitet och vävnadskoordinater (figur 1, steg 6).
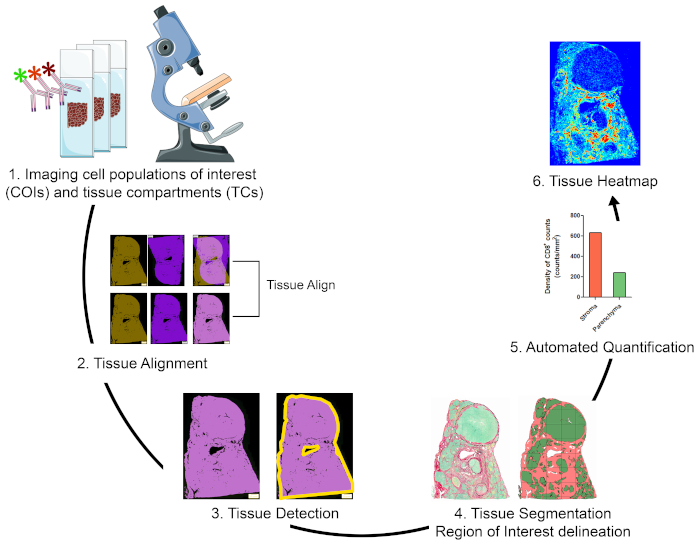
Figur 1: Schematisk representation av strategin för att visualisera, kvantifiera och kartlägga immunceller i TME. (1)Seriell hela vävnadssektioner var färgade för märkning av COI och TCs. Färgade hela vävnadssektioner digitaliserades med hjälp av en hel bildskanner. (2)Bilder som förvärvats från serieavsnitt kopplades, anpassades och avregistrerades på ett automatiserat sätt med hjälp av en Tissuealign-analysmodul. En sammansatt bild genererades från hög precision justering av enskilda bilder. (3)Ett användardefinierat protokoll användes för automatisk detektion av vävnadsassocierade pixlar (TAPs) i den sammansatta bilden. (4)Vävnaden segmenterades i TCs (t.ex. stroma och parenkym) definieras som ROIs. (5)Användardefinierade protokoll användes för automatisk detektion och kvantifiering av COI i olika TC.(6)Vävnadsvärmekartor för COI genererades. Klicka här för att se en större version av denna siffra.
Cois och TCs för avbildning
Tre seriella FFPE hela vävnad delar av resected tumör från ett ämne med HBV-associerade levercancer carcinom var färgas i en eller flera rundor av färgning som i figur 2A. Avsnitt I var färgas med H & E för att visa vävnadsarkitektur, cellmorfologi, och för att bestämma kliniskt relevanta parametrar såsom typ av malignitet, tumör grad, och övergripande bedömning av immun infiltration (Figur 2C). I sammanhängande avsnitt II användes två omgångar mIF för märkning av leverparenkymala och icke-parenkymala celler (figur 2A). I den första omgången, normala och tumör fartyg visualiserades med CD34 färgning av endotel celler. Dessutom identifierades epitelceller (hepatocyter och cholangiocyter) med cytokeratin 8/18, och fibrogenic aktiverade lever stellate celler identifierades som alfa glatt muskulatur aktin positiva (αSMA +) celler (figur 2C). Efter bild förvärv, vävnad sektioner var strippad och reprobed med antikroppar mot makrofager (CD68), och myofibroblasts (desmin). För att bättre karakterisera tumör immun infiltrate, intilliggande seriell avsnitt III var färgas med hjälp av två rundor av mIF för cellulära markörer CD3, CD4, CD8, gaffelhuvudet rutan P3 (FoxP3) och myeloperoxidase (MPO). I samtliga fall användes DAPI som en nukleär mothåll. Slutligen var avsnitt III färgas med PSR fläck och mothållna med snabb grön för att visualisera fibrillar kollagen och segment vävnaden i stroma och parenkym (figur 2C).
En hel bild scanner utrustad med en 20X objektiv användes för att digitalisera färgade sektioner och för att skapa virtuella bilder. Sex bilder förvärvades från de tre serieavsnitten (figur 2B) och de virtuella bilderna analyserades därefter med vis-programvaran enligt den schematiska representationen i figur 1.
Bildanalys
Bildanalysen omfattade fem steg: 1) vävnadsjustering; 2) vävnad upptäckt; 3) vävnad segmentering; 4) Automatiserad kvantifiering av coi. och 5) vävnad värme kartläggning. Alla protokoll för bildanalys har utvecklats med hjälp av author-modulen i bildanalysprogramvaran och kallas i texten för APP.
Vävnadsjustering
Sex virtuella bilder från tre seriella sektioner, som spänner över 11 markörer plus H & E och PSR fläckar, laddades in i Tissualign modulen av bildanalys programvara. Därefter var bilderna länkade, anpassade och avregistrerade på ett automatiserat sätt, vilket genererade en 11-plex plus H & E och PSR virtuell sammansatt bild, som innehåller alla lager av de enskilda bilderna(Figur 2A-C). Justeringen var korrekt när det gäller bilder från intilliggande serieavsnitt, som visar motsvarande vävnadsstrukturer placerade och ordnade på ett homologt sätt vid justeringen (figur 2C och figur S1A). Dessutom var justeringen exakt på den individuella cellnivån för bilder från samma avsnitt (figur S1B). Tiden för automatisk justering beror på antalet, storleken, komplexiteten och likheten hos de bilder som ska justeras. Anpassningen av de ovan nämnda sex virtuella bilderna tog 15 min i vår VIS station.

Figur 2: Färgning av seriella vävnadssektioner och bildjustering. (A)Sammanfattning av färgningar som gjorts på tre seriella avsnitt för visualisering av COI och TCs. Nummer inom parentes anger bildbeteckning. För avsnitten II och III, var vävnader strippad och reprobed med en andra cocktail av antikroppar. (B)Översikt över sex enskilda hela vävnadsbilder före och efter vävnadsjustering (vänster respektive höger). Skalbar = 3 500 μm(C) Zoomad vy av justerade bilder. Skala bar = 80 μm. Klicka här för att se en större version av denna siffra.
Upptäckt av vävnad
När bilderna var länkade och anpassade, försökte vi identifiera TAPs(figur 3A). För att utforma en APP för automatisk upptäckt av TAPs (APP 1, Tabell 1),drog vi nytta av två egenskaper som skiljer TAPs från pixlar som inte är associerade med vävnad. Först är DAPI-signalen (blått band) begränsad till kärnorna, som är belägna uteslutande i vävnaden, vilket innebär att alla DAPI + pixlar är en delmängd av TAPs. För det andra har TAPs högre autofluorescenssignal i de gröna och gula banden jämfört med pixlar som inte är associerade med vävnaden. Därför utvecklade vi APP 1 för vävnadsdetektering (tabell 1), som detekterar TAPs baserat på baslinjesignal i dessa kanaler med hjälp av enkla tröskelvärden. Tröskelvärden för de blå, gröna och gula banden sattes så att TAPs hade bakgrundsintensitetsvärden över tröskelvärdena, medan pixlar som inte är associerade med vävnaden hade värden nedan. APP 1 för vävnadsdetektering tillämpades på bild-IIA, som innehåller lager i de blå, gröna och gula kanalerna (figur 3A). Som utgångar av APP 1, en ljusgrön mask fastställdes ovanpå TAPs, och en ROI kallas "Tissue" var avgränsade (utgång, figur 3A). Dessutom fastställdes vävnadsområdet som en kvantitativ produktionsvariabel. Eftersom APP 1 inte innehåller pixlarna som inte är associerade med vävnaden i ROI-vävnaden uteslöts de från efterföljande analys baserad på denna roi (figur 3A). Precisionen i APP 1 vid identifiering av TAPs visas i figur 3A.
Vävnadssegmentering och avgränsning av ROIs för TCs
Därefter fortsatte vi att definiera olika fack inuti ROI vävnad genom segmentering vävnaden i stroma kontra parenkym. Vi använde PSR färgade bilden (IIIC, figur 2C),där stroma kan definieras som det område som förknippas med nedfall av fibrillar kollagen (rött band), parenkymet som det område där fibrillar kollagen är frånvarande, och den snabba gröna mothäftning färgämne råder (grönt band) (figur 3B). Vi skapade APP 2(Tabell 1)för att digitalt avgränsa TCs Stroma och Parenchyma. Denna APP fungerar på den fördefinierade ROI-vävnaden (utgång, figur 3A)och använder representativa stroma- och parenkymområden för att träna klassifierarverktyget som är integrerat i modulen Bildanalys. Den tränade klassificeraren tilldelar pixlarna till antingen en stroma eller en parenkymetikett (lax respektive grön, figur 3B). Vid klassificering av pixlar utförde APP 2 morfologiska operationer som syftar till att definiera ROIs Stroma och Parenchyma (figur 3B och tabell 1). Prestandan för APP 2 vid klassificering av pixlar och generera respektive ROIs visas i figur 3B. Dessutom kvantifierar APP 2 området av stroma och parenkym. Slutligen, även om segmenteringen görs med hjälp av PSR färgade avsnitt, kan de skisserade stroma och parenkym regioner överföras till någon bild som är anpassad till PSR-bilden.

Figur 3: Automatiserad vävnadsdetektering/segmentering och generering av respektive rois. (A) Bild IIA användes för att identifiera TAPs (vänster bild, skalstång = 6000 μm). En ljusgrön mask tilldelades TAPs med HJÄLP AV APP 1(Tabell 1)som genererar en avkastning som kallas Vävnad (utgång 1). Höger, infälld visar zoomad vy som visar precisionen i APP 1 på att upptäcka TAPs. Skalstång = 350 μm. (B) ROI-vävnaden (utgång 1) segmenteras i stroma och parenkym med APP 2. Bilden till vänster visar en vy av ROI Tissue segmenteras i ROI stroma (lax) och ROI parenkym (grön). Skala bar = 4.500 μm. Till höger, zoomade vyer av infälld för ROI Tissue, den ursprungliga PSR färgning (bild IIIC), och ROIs stroma och parenkym. Skalstång = 250 μm. Klicka här för att se en större version av denna siffra.
Automatiserad kvantifiering av coi
Därefter fortsatte vi att identifiera, lokalisera och kvantifiera COI i ROIs Stroma och Parenchyma. APPs 3 till 8(Tabell 1)skapades för att lokalisera och räkna följande COI: CD4 +FoxP3+, CD8+, CD68+, MPO+, αSMA+, respektive CD34+. APP 3 har utformats för att lokalisera och räkna CD4+FoxP3+-celler (bild IIIA, figur 2C) som surrogatmarkörer för regulatoriska T-celler (Tregs). Detta protokoll upptäcker kolisering av signalen från den nukleära transkriptionsfaktorn FoxP3 (rött band) och DNA-märkningsfärgen DAPI (blått band). Med tanke på att nyligen aktiverade T-celler upregulate FoxP3, för att berika för Tregs vi sätta trösklar för preselecting endast ljusa FoxP3 + celler (FoxP3hi). Därefter, av alla förvalda DAPI + FoxP3hi celler, endast de som var omgivna av ljusa ringformade CD4-signaler (grönt band) var märkta och räknas som FoxP3hiCD4 + celler (rosa etikett, Figur 4A). Densiteten hos FoxP3hiCD4+ celler i ROIs Stroma och Parenchyma fastställdes som kvantitativa utdatavariabler av APP 3 (figur 4A).
På samma sätt har APPs 4 till 6 utformats för detektion av CD8+, CD68+ och MPO+-celler. Dessa API:er har samma baslinjedesign för att identifiera och kvantifiera COI. Specifikt identifieras COI baserat på signalintensitet från den specifika cellpopulationens biomarkör, och sedan utförs flera morfologiska steg efterbearbetning för att avgränsa enskilda celler (tabell 1). De enskilda cellerna eller COI är märkta, räknade och deras vävnadskoordinater registreras. ApPs 4 till 6 bestämmer också densiteten hos COI i ROIs Stroma och Parenchyma (figur 4B-D).
Kvaliteten på vår DAPI färgning var inte tillräckligt bra för att integrera kärnor segmentering i APPs 3 till 6, så vi kan inte se till att alla individuellt märkta objekt är enskilda celler. Av denna anledning uttryckte vi celltätheten i antal märkta föremål/mm2 (figur 4). Cellaggregaten har dock delats upp i enskilda celler i stegen efter bearbetning som inbyggts i APPs 3 till 6, och omfattande visuell inspektion visade att de flesta märkta objekt motsvarade enskilda celler.
För att upptäcka αSMA+ och CD34+ utvecklade vi APPs 7 respektive 8(tabell 1). Båda ap:erna känner av den specifika signalen baserat på tröskelvärden och bestämmer procentandelen positivt område i rois Stroma och Parenchyma (figur 4E–F).
En av de mest intressanta möjligheterna att generera virtuella multiplex diabilder är analysen av colocalization uttryck. Vi genererade APP 10 för att upptäcka kolacalisering mellan αSMA och desmin, två markörer som uttrycks av myofibroblasts i levern. APP 10 använder tröskelvärden för att hitta pixlar som är positiva för αSMA, desmin och αSMA plus desmin (Tabell 1). Som kvantitativa utdatavariabler bestämmer APP 10 αSMA+-området, desmin+-området och området för colocalized uttryck för dessa två markörer (figur S3).

Figur 4: Identifiering och kvantifiering av cois i TCs stroma och parenkym. (A–F) Automatisk identifiering och kvantifiering av CD4+FoxP3+, CD8+, CD68+, MPO+, αSMA+, och CD34+ COI:er i ROIs Stroma och Parenchyma med protokollen 3, 4, 5, 6, 7 respektive 8 (tabell 1). Visas till vänster är de ursprungliga bilderna, i mitten bearbetade bilder, och till höger kvantifieringar. För figur 4A–D, skala bar = 40 μm. För figurerna 4E och F, skala bar = 350 μm. Klicka här för att se en större version av denna siffra.
Som ett alternativ till att kvantifiera COI i TCs Stroma och Parenchyma, bestämde vi tätheten av immunceller i de olika maligna knölar som heter 1 till 4 (Figur 5A, Hoch I). Avkastningen på sysselsatt kapital för varje nodule har avgränsats manuellt enligt figur 5A. Särskiljande vävnad immun signaturer kännetecknas varje nodule, ytterligare avslöjar inneboende heterogenitet TME.
Värmekartor för vävnad
Som nämnts ovan, APPs 3 till 8 lagra vävnadkoordinater för varje individuellt märkt objekt. Denna funktion gör det möjligt för automatiserad generering av vävnadskartor där regioner med hög densitet hos en viss cellpopulation visas som hotspots (röd) och regioner med relativt låg densitet som kalla fläckar (mörkblå). Mellandensitetsvärden tilldelas färger enligt den färgskala som visas i figur 5. Vävnad heatmaps genererades av APPs som delade upp bilderna i cirklar med 50 μm diameter och tilldelas en färg enligt den relativa densiteten hos en viss COI inuti cirkeln. Som visas i figur 5B-Gvar placeringsmönstren och intensitetsfördelningen för de olika coi i TME ganska varierande. På individuell härdningsnivå var dessutom placeringen av olika populationer i vävnadsområdet unik (figur S2A–C). För att ge ett exempel på kraften i denna teknik och för att visualisera den rumsliga organisationen av hotspots från olika populationer i samma knöl extraherades de aktiva fläckarna från enskilda celltyper manuellt och kartlades tillsammans på konturerna av nodule 2 (figur S2, Figur Doch figur E).

Figur 5: Vävnadsvärmekartor för COI i TME. (A)Picrosirius Röd färgning som visar var knölarna 1, 2, 3 och 4 finns. (B–G) Mjukpappersvärmekartor för CD4+FoxP3+, CD8+, CD68+, MPO+, CD34+ respektive αSMA+ COI. Mörkblå anger relativ låg densitet, och rött indikerar relativ hög densitet. Mellanliggande densitetsvärden tilldelas färger enligt den visade färgskalan. (H och I) Kvantifiering av COI i knölar 1, 2 och 3 + 4 organiserade per celltyp och per nodule, respektive. Klicka här för att se en större version av denna siffra.

Kompletterande figur S1: Validering av vävnadsjustering. (A) CD34 färgning (i rött) görs på avsnitt II (ingång 1) används för att generera en CD34 mask i grönt (utgång 1). Den gröna masken (utgång 1) läggs över på H&E-bilden från det justerade serieavsnittet I (ingång 2). Den sammanfogade bilden visar perfekt korrespondens av vaskulära strukturer. Skalstång = 50 μm. (B)Bild IIIA som visar sammanslagningen av DAPI, CD4 och FoxP3 (ingång 1) användes för att generera en etikett för CD4 + FoxP3 + celler (utgång 1 i magenta). Output 1 etikett överfördes till justerad bild IIIB (ingång 2) och visar perfekt korrespondens mellan paren FoxP3/DAPI och CD4/CD3 i sammanfogningen bilden. Skalstång = 15 μm. Klicka här för att se en större version av denna siffra.

Kompletterande figur S2: Zoomad bild av vävnadsvärmekartor. (A–C) Vävnadsvärmekartor för CD4+FoxP3+, CD8+, CD68+, och MPO+-celler i knölar 1–4. Skalstänger i knölar 1, 2 och 3 + 4 representerar 1 500 μm, 700 μm respektive 500 μm. (D)Kontur av nodule 2 med svart heldragen linje. (E)Aktiva punkter för CD4+FoxP3+, CD8+, CD68+, och MPO+-celler i nodule 2 extraherades och mappades samman på den 2-kontur som definieras i D. Klicka här för att se en större version av denna siffra.

Kompletterande figur S3: Kolacaliseringsanalys. (A) Till vänster och mitten finns bilder av αSMA-etikett i grönt respektive desmin-etikett. Till höger finns ett αSMA/desmin dubbelt positivt område i gult. (B)Kvantifiering av αSMA+-området, desmin + område och αSMA/desmin dubbelt positivt område. Skalstång = 150 μm. Klicka här för att se en större version av denna siffra.
| App | Syfte | Klassificering | Klassificering | Steg efter bearbetning | Utdatavariabler |
| Metod | Funktioner | ||||
| (pixelvärde) | |||||
| 1 | Upptäckt av vävnad | Tröskel | Kanal DAPI (150) | o Etikettobjekt med colocalized över tröskelvärden för de 3 kanalerna | o ROI-vävnad |
| Kanal FITC/A488 (120) | o Stäng positivt objekt 5 pixlar | o Vävnadsområde | |||
| Kanal TRITC/A568 (40) | o Skapa ROI-vävnad | ||||
| 2 | Vävnadssegmentering | Beslut Skog | RGB-R median | o Fyll hål | o ROI Stroma |
| RGB-G median | o Skapa ROI Stroma | o Stroma-området | |||
| RGB-B median | o Skapa ROI Parenchyma | o ROI Parenchyma | |||
| IHS-S median | o Parenchyma-området | ||||
| Medianvärde för H&E Eosin | |||||
| 3 | Så här hittar och kvantifierar du CD4+ FoxP3+-celler | Tröskel | Kanal DAPI (>600) | o Etikettobjekt med colocalization av DAPI och Cy5/A647, omgiven av FITC/A488-signal | o Antal och densitet av CD4 + FoxP3 + celler i ROIs Stroma och Parenchyma |
| KANAL FITC/A488 poly utjämning (>850) | o Klara föremål mindre än 7 μm2 | o Koordinater för enskilda CD4+FoxP3+-celler | |||
| Kanal Cy5/A647(>800) | |||||
| 4 | Så här hittar och kvantifierar du CD8+-celler | Tröskel | Kanal DAPI (<1200) | o Rensa positiva objekt mindre än 15 μm2 | o Antal och densitet av CD8 + celler i ROIs Stroma och Parenchyma |
| Kanal Cy5/A647 median (>80) | o Stäng positiva objekt 2 pixlar | o Koordinater för enskilda celler | |||
| o Separata objekt | |||||
| 5 | Så här hittar och kvantifierar du CD68+-celler | Tröskel | Kanal FITC/A488 (>200) | o Rensa positiva objekt mindre än 20 μm2 | o Antal och densitet av CD68 + celler i ROIs Stroma och Parenchyma |
| o Vidga positiva objekt 3 pixlar | o Koordinater för enskilda CD68+-celler | ||||
| o Separata objekt | |||||
| 6 | Så här lokaliserar och kvantifierar du MPO+-celler | Tröskel | Kanal DAPI (>400) | o Klara föremål mindre än 5 μm2 | o Antal och densitet av MPO + celler i ROIs Stroma och Parenchyma. |
| Kanal TRITC/A568 (900-4000) | o Vidga 3 pixlar positiva objekt | o Koordinater för enskilda MPO+-celler. | |||
| o Separata objekt | |||||
| 7 | Så här lokaliserar och kvantifierar du αSMA+-området | Tröskel | Kanal TRITC/CF568 (>1050) | o Rensa positiva objekt mindre än 25 μm2 | o Antal och densitet av αSMA+ område i ROIs Stroma och Parenchyma |
| o Vidga 3 pixlar positiva objekt | o Koordinater för αSMA+-pixlar | ||||
| 8 | Så här hittar och kvantifierar du CD34+-området | Tröskel | Kanal DAPI (<5000) | o Rensa positiva objekt mindre än 25 μm2 | o Antal och densitet av CD34 + område i ROIs Stroma och Parenchyma |
| Kanal Cy5/A647 median (>120) | o Vidga 3 pixlar positiva objekt | o Koordinater för CD34+ pixlar | |||
| 9 | Skapa vävnadsvärmekartor för en viss cellpopulation | Värmekarta för objekt | Värmekarta för objekt | o Heatmap (uppräknat) | |
| Ritningsradie 50 μm | --- | ||||
| 10 | Kvantifiera kolisering mellan αSMA och Desmin | Tröskel | Kanal TRITC (CF568) (>1050) | o Etikettobjekt med över tröskelvärden för TRITC (CF568) | o Kvantifiera colokaliserat uttryck för αSMA och Desmin |
| Kanal Cy5 (A647) (>1000) | o Etikettobjekt med över tröskelvärden för Cy5 (A647) | ||||
| o Etikettobjekt med colocalization av över tröskelvärden för TRITC (CF568) och Cy5 (A647) | |||||
| o Rensa positiva objekt mindre än 25 μm2 |
Tabell 1: Allmänna parametrar som används för utformning av apps som används för bildanalys. De parametrar som anges i den här tabellen är anpassade till de unika egenskaperna hos de bilder som används i denna analys (t.ex. bakgrund, artefakter osv.) och kanske inte är tillämpliga på andra bilder. Eftersom de nämnda stegen efter bearbetning definierades för de specifika bilder som analyserades i den här studien, är de avsiktligt inte detaljerade. Användaren bör anpassa APPs till de bilder som ska analyseras.
| Avsnitt/Färgning | Primär antikropp | Sekundär antikropp |
| Avsnitt II/1st Färgning | Mus IgG2a anti-mänskliga αSMA Mus IgG1 anti-människa CD34 Kanin anti-mänskliga Cytokeratin 8/18 |
Get anti-mus IgG2a CF568 Råtta anti-mus IgG1 A647 Åsna anti-kanin A488 |
| Avsnitt II/2nd Färgning | Kanin anti-mänskliga Desmin Mus anti-mänskliga CD68 |
Åsna anti-kanin A647 Åsna anti-mus DyLight 755 |
| Avsnitt III/1st Färgning | Mus anti-mänskliga CD4 Kanin anti-mänskliga FoxP3 Get anti-mänskliga MPO |
Åsna anti-mus A488 Åsna anti-kanin A647 Åsna anti-get A568 |
| Avsnitt III/2nd Färgning | Kanin anti-mänskliga CD3 Mus anti-mänskliga CD8 |
Åsna anti-mus DyLight 755 Åsna anti-kanin A647 |
Tabell 2: Primär-Sekundära antikroppspar för mIF.
Discussion
Enkla, tillgängliga och lätta att utföra multiplextekniker som tillåter rumslig upplösning av immunceller i vävnadssektioner behövs för att kartlägga immunförsvaret i cancer och andra immunologiska sjukdomar. Här beskriver vi en strategi som integrerar allmänt tillgängliga märkning och digital analys tekniker för att utöka multiplexering kapacitet och flerdimensionell bedömning av bildframställning analyser12,13,17,19. Färgning av tre seriella sektioner för olika markörer, och återanvändning av sektioner genom strippning och reprobing tekniker, gjorde det möjligt för oss att visualisera 11 parametrar utöver H & E och PSR fläckar. Sex bilder från dessa avsnitt var i linje på ett automatiserat sätt med hjälp av vävnad justering modulen. Justeringen var exakt på den enskilda cellnivån för bilder som kommer från samma avsnitt och mycket samstämmiga för bilder som kommer från angränsande sektioner. Virtuell multiplexering gjorde det möjligt för oss att avgöra hur markörer som visualiseras i ett avsnitt relaterar rumsligt till markörer som visualiseras i ett annat sammanhängande avsnitt. Medan vissa av de färgningar märkta COI, andra märkt TCs, så att vi kan kvantifiera COI i de olika TCs. Användningen av programvaruverktyg för automatiserad kvantifiering av COI förenklas och påskyndas avsevärt bearbetningen av bilder. Dessutom tillämpades digital analys på hela vävnadssektioner i stället för utvalda synfält, vilket resulterade i en opartisk representation av TME. Eftersom vävnadskoordinaterna för COI registrerades var det dessutom möjligt att generera värmekartor för vävnad.
Det finns flera områden i det här protokollet där felsökning kan behövas. För det första kan dålig antigen hämtning påverka kvaliteten på mIF, därför bör typen av antigen hämtningsbuffert och varaktighet optimeras för de specifika tillstånd för analys/biomarkör som används. För det andra bör den typ av blockerande lösning som används anpassas till vävnader/antigen/arter av primära och sekundära antikroppar. I våra händer, tillägg av 10% totalt serum från de arter där vävnaden kommer från blockerade Fc receptorer, och därmed minskat ospecificerad antikropp bindande. Tillsats av 10% av serum från arten de sekundära antikropparna höjdes i skulle minimera direkt ospecifik fastsättning av sekundära antikroppar till vävnadssektionen. För det tredje är validering av specificiteten hos de primära och sekundära antikropparna med rätt positiva och negativa kontroller nödvändig. För det fjärde, ökad autofluorescens i vissa kanaler och spridning av DAPI på primära antikroppar strippning är också vanliga. För att ta itu med den förbättrade autofluorescensen använde vi primära/sekundära antikroppspar där den specifika signalen hade intensitetsvärden minst 5x i bakgrunden. Slutligen kan vissa antikroppar med hög affinitet inte elueras med regelbundna strippningsprocedurer. I detta fall rekommenderar vi att du använder sådana antikroppar i den sista rundan av märkning. Användaren kan behöva prova olika färgningssekvenser för att hitta den optimala konfigurationen för de antikroppar av intresse. Strippnings effektivitet bör bekräftas innan en andra eller tredje omgång märkning.
Den största begränsningen och utmaningen med denna strategi är att hitta rätt kombinationer av primära och sekundära fluorescerande antikroppar för markörer av intresse. Att hitta primära antikroppar som föds upp hos olika arter eller med olika isotyper som kan användas samtidigt begränsas av vad som är kommersiellt tillgängligt. De flesta hela diaskannrar är utrustade med lampor och filter som tillåter avbildning högst fem kanaler, och sekundära antikroppar i rätt art och rätt fluorofore är inte alltid tillgängliga. Vi övervann delvis dessa begränsningar med hjälp av seriella färgningar och sekventiell märkning. Flera antikroppskombinationer kan behöva testas för att komma fram till den bästa kombinationen för markörer av intresse. En annan begränsning är kvaliteten på DAPI färgning, eftersom strippning och reprobing kanske inte alltid tillåter utför kärnor segmentering.
Vävnadsmodulen kräver minimal utbildning och inga programmeringsfärdigheter från användarna. Programvaran tillåter teoretiskt anpassning av ett obegränsat antal bilder. Exakt anpassning beror dock på relateradhet i sektioner, där närmare sektioner som är mer histologiskt samstämmiga är mer exakt anpassade. Vi använde author-modulen i VIS för att generera APPs. Grundläggande kunskaper om bildanalys behövs för att skapa APPs, men detta är lika fallet när du använder någon annan bildanalys programvara. De unika fördelarna med VIS jämfört med andra bildanalysprogram inkluderar automatiserad anpassning av bilder från sektioner som utarbetats med olika metoder (t.ex. Detta möjliggör colocalization studier av flera markörer av intresse med hjälp av virtuella multiplexering. Dessutom möjliggör den flexibla och användarvänliga utformningen av AP:er användarspecifik anpassning. Automatiserad kvantifiering och kartläggning, och möjligheten att bearbeta hela vävnadssektioner, sparar tid och minskar bias jämfört med manuell räkning genom visuell inspektion.
Denna strategi är ett mycket användbart forskningsverktyg för vävnadsimmunologi i samband med cancer och autoimmunitet men är fortfarande inte förbrukat för klinisk användning. Med ytterligare standardisering och validering kan den användas i framtiden för flera tillämpningar (t.ex. för att kartlägga immunlandskapet i cancer för att förutsäga och övervaka svaret på immunoterapeutiska medel). Det kan också anpassas till olika inflammatoriska tillstånd (t.ex. inflammatorisk tarmsjukdom) för att kombinera patologisk utvärdering med prognostiska biomarkörer.
De viktigaste kritiska stegen i detta protokoll är effektiviteten/specificiteten hos märkningen och robustheten hos de utformade API:erna för avsedd användning eller biomarkör. Därför är regelbunden validering genom visuell inspektion, särskilt vid utformningen av en ny APP, viktigt. Effektiv användning av flera omgångar av strippning och reprobing eller olika typer av fläckar på samma avsnitt är kritiska komponenter och kan vara vävnad eller avsnitt specifika. Det är viktigt att verifiera effektiviteten i sådana processer innan du fortsätter med stor batchanalys.
Sammanfattningsvis tillhandahåller vi en strategi som maximerar den kvantitativa och rumsliga information som kan erhållas från värdefulla kliniska vävnadsprover. De resurser, den utrustning och den kunskap som krävs för att genomföra denna metod är allmänt tillgängliga. Vi föreslår denna metod som en användbar guide för planering analyser som syftar till att identifiera, kvantifiera och kartlägga immuncellpopulationer i TME.
Disclosures
Författarna förklarar inga intressekonflikter.
Acknowledgments
Vi tackar studiedeltagaren. Vi tackar Louise Rousseau, samordnare för HBP biobank för återvinning av vävnadsprover och alla tillhörande klinisk information. Vi erkänner de molekylära patologi och cell imaging kärnanläggningar på CRCHUM och Michael Persch från Visiopharm för utmärkt tekniskt stöd. Finansiering: Denna studie stöddes av bidrag från Canadian Liver Foundation, Fonds de recherche du Québec–Santé (FRQS) AIDS and Infectious Disease Network (Réseau SIDA-MI) och Canadian Network on Hepatit C (CanHepC). CanHepC finansieras genom ett gemensamt initiativ från Canadian Institutes of Health Research (CIHR) (NHC-142832) och Public Health Agency of Canada. M.F.M. fick stipendier från Université de Montréal, Bourse Gabriel Marquis och FRQS. T.F. fick doktorandstipendium från CIHR och CanHepC. S.T. innehar Roger-Des-Groseillers ordförande i hepatobiliary och bukspottskörteln onkologiska kirurgi, Université de Montréal.
Författare bidrag: M.F.M. utformade, utförde experiment och analyserade data. T.F. designade experiment. A.C-B. teknisk vägledning. G.S. utförde all patologisk bedömning av försökspersonen och gav input på alla patologiska aspekter. L.M. utförde H&E-färgning, optimerade och utförde bildförvärv. M.N.A. utförde PSR-fläcken och gav värdefull teknisk ingång. N.B. bidrog till bildanalysen. S.T. är huvudforskare för HBP biobank och ansvarar för att övervaka den övergripande driften av biobanken. Han gav också ovärderlig input på alla aspekter av projektet och dess kliniska konsekvenser. M.F.M, T.F., och N.H.S. konceptualiserade och utformade studien. N.H.S. övervakade arbetet och erhöll finansiering. M.F.M., T.F., A.C-B och N.H.S. skrev manuskriptet. Alla författare granskade och godkände manuskriptet.
Materials
| Name | Company | Catalog Number | Comments |
| Antigen Retrieval Solution: Sodium Citrate Buffer (10 mM Sodium Citrate, 0.05% v/v Tween 20, pH 6.0) | |||
| Blocking Solution: 1 % BSA, 10 % filtered human serum, 10 % filtered donkey serum, 0.1 % Tween 20, and 0.3% Triton in PBS | |||
| Bovine serum albumin (BSA) | Multicell | 800-095-EG | |
| Coplin jars (EASYDIP SLIDE STAINING SYSTEM) | Newcomersupply | 5300KIT | |
| Cover slides | Fisherbrand | 12-545E 22*50 | |
| Direct Red 80 | Sigma Aldrich | 365548 | |
| Donkey Serum | Sigma Aldrich | D9663 | |
| Ethanol 100% | |||
| Electric pressure cooker | Salton | ||
| Eosin | Leica Biosystems | 3801600 | CAUTION, eye irritation |
| Fast Green FCF | Sigma Aldrich | F7252 | CAUTION, harmful by inhalation, ingestion and skin absortion |
| FFPE section (4μm) slides | |||
| Glycine 0,1 M in PBS | |||
| Hematoxylin Stain Solution, Gil 1. Formulation, Regular Strength | Ricca Chemical Company | 3535-32 | |
| Holder (EasyDip Staining Jar Holder) | Newcomersupply | 5300RK | |
| Human Serum | Gemini | 22210 | |
| Humidity chamber | Millipore Sigma | Z670138-1EA | |
| Pap pen | abcam | ab2601 | |
| PBS | |||
| PBS-Tween 20 (0.1% v/v) | |||
| Permount Mounting Media | Fisher Chemical | SP15-500 | |
| Picric Acid 1.3 % | Sigma Aldrich | P6744 | CAUTION, skin and eye irritation |
| Picro-Sirius Red/Fast Green solution: Fast Green 0.1 % w/v + Sirius Red 0.2 % w/v in 1,3 % picric acid solution | |||
| Primary Antibody Anti-αSMA | Mouse IgG2a 1A4 | Sigma A2547 | Dilution 1/100 |
| Primary Antibody Anti-CD34 | Mouse IgG1 HPCA1/763 | Novus Biologicals NBP2-44568 | Dilution 1/250 |
| Primary Antibody Anti-Cytokeratin 8/18 | Rabbit EP17/EP30 | Agilent IR09461-2 | Ready to use |
| Primary Antibody Anti-CD68 | Mouse KP1 | Abcam ab955 | Dilution 1/200 |
| Primary Antibody Anti-Desmin | Rabbit Polyclonal | Invitrogen PA5-16705 | Dilution 1/200 |
| Primary Antibody Anti-CD4 | Mouse N1UG0 | Affymetrix 14-2444 | Dilution 1/250 |
| Primary Antibody Anti-FoxP3 | Rabbit 1054C | R & D MAB8214 | Dilution 1/100 |
| Primary Antibody Anti-MPO | Goat Polyclonal | R & D Systems AF3667 | Dilution 1/250 |
| Primary Antibody Anti-CD3 | Rabbit SP7 | Abcam ab16669 | Dilution 1/200 |
| Primary Antibody Anti-CD8 | Mouse C8/144B | Invitrogen 14-0085-80 | Dilution 1/200 |
| Secondary Antibody Donkey anti-mouse A488 | Polyclonal | Invitrogen A-21202 | Dilution 1/500 |
| Secondary Antibody Donkey anti-Rabbit A488 | Polyclonal | Invitrogen A-21206 | Dilution 1/500 |
| Secondary Antibody Donkey anti-goat A568 | Polyclonal | Invitrogen A-11057 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit A647 | Polyclonal | Invitrogen A-31573 | Dilution 1/500 |
| Secondary Antibody Rat anti-mouse IgG1 A647 | RMG1-1 | Biolegend 406618 | Dilution 1/500 |
| Secondary Antibody Goat anti-mouse IgG2a CF568 | Polyclonal | Sigma Aldrich SAB4600315 | Dilution 1/500 |
| Secondary Antibody Donkey anti-mouse DyLight 755 | Polyclonal | Invitrogen SA5-10171 | Dilution 1/500 |
| Secondary Antibody Donkey anti-rabbit DyLight 755 | Polyclonal | Invitrogen SA5-10043 | Dilution 1/500 |
| SDS | BioShop | SDS001,500 | CAUTION, oral skin and eye toxicity |
| Shandon multi-program robotic slide stainer | LabX | 11384903 | |
| Shandon Xylene Substitute, | Thermo Fisher Scientific | CA89413-336 | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Shaking water bath | |||
| SlowFade Gold antifade reagent with DAPI | Invitrogen | S36938 | |
| Sodium Citrate Dihydrate | Millipore Sigma | 1545801 | CAUTION, eye irritation |
| Stripping Buffer: mix 20 ml 10% w/v SDS with 12.5 ml 0.5 M Tris-HCl (pH 6.8), and 67.5 ml ultra-pure water. Under a fume hood, add 800 uL of 2-mercapto ethanol (114,4 mM final concentration) | |||
| Triton X-100 | Sigma Aldrich | T8787-50ML | |
| Tris-HCl | BioShop | 77-86-1 | |
| Tween 20 | Fisher Scientific | BP337-500 | |
| VIS Software | Visiopharm | ||
| Whole slide scanner Olympus BX61VS | Olympus | Microscope: Olympus Slide Scanner BX61VS, 5 slides scanner, motorized stage, autofocus. Camera: Lightsource: Xcite-120. Filters: BrightLine® Sedat filter set (# LED-DA/FI/TR/Cy5-4X4M-B-000, Semrock) | |
| Xylene | Sigma Aldrich | 214736-4L | CAUTION, Flammable, skin and eye irritation, Harmful when inhaled |
| Xylene : Ethanol solution (1:1 v/v) | |||
| 2-mercaptoethanol | Sigma | M6250 | CAUTION, harmful by ingestion, inhalation, fatal if sking absortion. Eye irritation. Use fume hood |
References
- Greten, F. R., Grivennikov, S. I. Inflammation and Cancer:Triggers, Mechanisms, and Consequences. Immunity. 51 (1), 27-41 (2019).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Binnewies, M., et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nature Medicine. 24 (5), 541-550 (2018).
- Taube, J. M., et al. Implications of the tumor immune microenvironment for staging and therapeutics. Modern Pathology: an official journal of the United States and Canadian Academy of Pathology, Inc. 31 (2), 214-234 (2018).
- Bindea, G., et al. Spatiotemporal dynamics of intratumoral immune cells reveal the immune landscape in human cancer. Immunity. 39 (4), 782-795 (2013).
- Galon, J., et al. Towards the introduction of the 'Immunoscore' in the classification of malignant tumours. The Journal of Pathology. 232 (2), 199-209 (2014).
- Finotello, F., Eduati, F. Multi-Omics Profiling of the Tumor Microenvironment: Paving the Way to Precision Immuno-Oncology. Frontiers in Oncology. 8, 430 (2018).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: a method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Giesen, C., et al. Highly multiplexed imaging of tumor tissues with subcellular resolution by mass cytometry. Nature Methods. 11 (4), 417-422 (2014).
- Porta Siegel, T., et al. Mass Spectrometry Imaging and Integration with Other Imaging Modalities for Greater Molecular Understanding of Biological Tissues. Molecular Imaging and Biology : MIB: the official publication of the Academy of Molecular Imaging. 20 (6), 888-901 (2018).
- Buchberger, A. R., DeLaney, K., Johnson, J., Li, L. Mass Spectrometry Imaging: A Review of Emerging Advancements and Future Insights. Analytical Chemistry. 90 (1), 240-265 (2018).
- Pirici, D., et al. Antibody elution method for multiple immunohistochemistry on primary antibodies raised in the same species and of the same subtype. Journal of Histochemistry and Cytochemistry. 57 (6), 567-575 (2009).
- Gendusa, R., Scalia, C. R., Buscone, S., Cattoretti, G. Elution of High-affinity (>10-9 KD) Antibodies from Tissue Sections: Clues to the Molecular Mechanism and Use in Sequential Immunostaining. Journal of Histochemistry and Cytochemistry. 62 (7), 519-531 (2014).
- van der Loos, C. M. Multiple immunoenzyme staining: methods and visualizations for the observation with spectral imaging. Journal of Histochemistry and Cytochemistry. 56 (4), 313-328 (2008).
- Stack, E. C., Wang, C., Roman, K. A., Hoyt, C. C. Multiplexed immunohistochemistry, imaging, and quantitation: a review, with an assessment of Tyramide signal amplification, multispectral imaging and multiplex analysis. Methods. 70 (1), 46-58 (2014).
- Toth, Z. E., Mezey, E. Simultaneous visualization of multiple antigens with tyramide signal amplification using antibodies from the same species. Journal of Histochemistry and Cytochemistry. 55 (6), 545-554 (2007).
- Robertson, D., Savage, K., Reis-Filho, J. S., Isacke, C. M. Multiple immunofluorescence labeling of formalin-fixed paraffin-embedded (FFPE) tissue. BMC Cell Biology. 9, 13 (2008).
- Segnani, C., et al. Histochemical Detection of Collagen Fibers by Sirius Red/Fast Green Is More Sensitive than van Gieson or Sirius Red Alone in Normal and Inflamed Rat Colon. PloS One. 10 (12), 0144630 (2015).
- Bolognesi, M. M., et al. Multiplex Staining by Sequential Immunostaining and Antibody Removal on Routine Tissue Sections. Journal of Histochemistry and Cytochemistry. 65 (8), 431-444 (2017).



