ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Este método descreve a purificação por citometria de fluxo de MEP e MKp de fêmures, tíbias e ossos pélvicos.
Abstract
Megacariócitos de medula óssea são grandes células poliploides que garantem a produção de plaquetas sanguíneas. Elas surgem de células-tronco hematopoiéticas através de megacaripoiesis. As etapas finais deste processo são complexas e classicamente envolvem os progenitores megacariócitos bipotentes -Eritrócitos (MEP) e os progenitores megacayócitos unipotentes (MKp). Essas populações precedem a formação de megacaiócitos de boa fé e, como tal, seu isolamento e caracterização poderiam permitir a análise robusta e imparcial da formação de megacaiócitos. Este protocolo apresenta em detalhes o procedimento para coletar células hematopoiéticas da medula óssea do rato, o enriquecimento de progenitores hematopoiéticos através do esgotamento magnético e, finalmente, uma estratégia de classificação celular que produz populações altamente purificadas de MEP e MKp. Primeiro, as células de medula óssea são coletadas do fêmur, da tíbia, e também da crista ilíaca, um osso que contém um alto número de progenitores hematopoiéticos. O uso de ossos de crista ilíaca aumenta drasticamente o número total de células obtidas por camundongo e, portanto, contribui para um uso mais ético dos animais. Um esgotamento da linhagem magnética foi otimizado usando contas magnéticas de 450 nm permitindo uma classificação celular muito eficiente por citometria de fluxo. Por fim, o protocolo apresenta a estratégia de rotulagem e gating para a classificação das duas populações progenitoras megacarióficas altamente purificadas: MEP (Lin-Sca-1-c-Kit+CD16/32-CD150+CD9dim) e MKp (Lin- Sca-1-c-Kit+CD16/32-CD150+CD9bright ). Esta técnica é fácil de implementar e fornece material celular suficiente para realizar i) caracterização molecular para um conhecimento mais profundo de sua identidade e biologia, ii) ensaios de diferenciação in vitro, que proporcionarão uma melhor compreensão dos mecanismos de maturação de megacaiócitos, ou iii) modelos in vitro de interação com seu microambiente.
Introduction
Plaquetas de sangue são produzidas por megacariócitos. Estas grandes células poliploides estão localizadas na medula óssea e, como em todas as células sanguíneas, elas são derivadas de Células-Tronco Hematopoiéticas (HSC)1. O caminho clássico de produção de megacaiócitos na medula óssea tem origem no HSC e envolve a geração de diferentes progenitores que restringem progressivamente seu potencial de diferenciação2. O primeiro progenitor a assinar o compromisso com a linhagem megacariocítica é o Progenitor megacarito-eritrócito (MEP), um progenitor bipotente capaz de produzir células eritrógradas e megacariócitos3,4,5. O MEP então produz um progenitor/precursor unipotente (MKp) que se diferenciará em um megacaiócito maduro capaz de produzir plaquetas. Os mecanismos envolvidos na geração desses progenitores, bem como sua diferenciação e maturação em megacaitos são complexos e apenas parcialmente compreendidos. Além disso, ainda não está clara a heterogeneidade da população do MEP em termos de potencial de diferenciação e de comprometimento intrínseco dessas células. Para decifrar esses processos, é essencial obter (ou ter acesso) a populações purificadas de MEP e MKp para análises moleculares e celulares únicas.
Vários estudos demonstraram combinações particulares de marcadores de superfície celular para a identificação de progenitores comprometidos com a linhagem megacariocítica no mouse6,7,8. A partir destes, foi elaborado um método que permite a purificação de MEP e MKp de camundongos. Este método foi otimizado para a obtenção de células em número e qualidade adequados para um grande número de ensaios. Com considerações éticas em mente, e a fim de minimizar o número de animais envolvidos nos experimentos, nós provocamos para colher a medula óssea do fêmur e tíbia, e também da crista ilíaca. Este osso contém uma alta frequência e número de progenitores hematopoiéticos e é a maior parte do tempo danificado durante a longa colheita óssea. Apresentado aqui é um método detalhado para a coleta confiável deste osso.
O segundo critério de otimização é produzir populações celulares altamente purificadas. A Classificação celular ativada fluorescente (FACS) é um método de escolha para obter populações purificadas de células de interesse. No entanto, os baixos rendimentos são alcançados quando a população celular de interesse é muito rara. Assim, são necessários procedimentos de enriquecimento. Neste protocolo, optou-se por um procedimento de seleção negativa usando contas magnéticas.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Os protocolos envolvendo animais foram realizados de acordo com o Comitê de Ética em Experimentos Animais da Universidade de Estrasburgo (Comité Régional d'Ethique en Matière d'Expérimentation Animale Strasbourg. Número de licença: E67-482-10).
1. Coleção de ossos de rato
- Sacrifique o animal em conformidade com as diretrizes institucionais.
NOTA: Os dados apresentados neste manuscrito foram obtidos de camundongos C57Bl/6 de 8 a 12 semanas de idade. O número de células obtidas, e a frequência das populações citadas podem variar de acordo com a idade e a cepa do rato. - Pulverize o corpo com 70% de etanol.
- Com a tesoura, faça uma incisão de 0,5-1 cm da pele perpendicular à coluna vertebral e rasgue a pele ao redor de todo o corpo. Puxe a pele da parte inferior do corpo e remova a pele.
- Coloque o animal na almofada de dissecção, de frente para baixo. Localize os ossos pélvicos deslizando os dedos ao longo da coluna exposta de cima para baixo. Para localizar a crista ilíaca, identifique a pequena colisão na região lombar próxima às barras traseiras (região anterosuperiora do osso pélvico). Figura 1A, B apresenta uma representação esquemática da anatomia do rato.

Figura 1: Anatomia do rato. (A) Raio-X do rato mostrando os ossos de cetameira. Observe o espaço entre o osso pélvico e a coluna vertebral (seta amarela), onde a tesoura deve ser inserida para separar adequadamente os espinhos traseiros do corpo do mouse (linha pontilhada amarela). (B) Representação esquemática dos ossos de interesse ricos em medula óssea. Os ossos pélvicos são retratados em vermelho, os fêmures em roxo, e as tíbias em verde. (C) Representação esquemática do osso pélvico do camundongo. O ísio corresponde à parte rica em medula do osso pélvico e é destacado em vermelho. Clique aqui para ver uma versão maior desta figura.
- Coloque a tesoura paralela à coluna vertebral contra as vértebras e perto da colisão da crista ilíaca. Vá cortar os músculos ao longo da lateral da coluna acima do osso pélvico deslizando a tesoura ao longo das vértebras até a cauda.
NOTA: Esta primeira seção de músculos também pode ser realizada usando uma lâmina de bisturi. - Coloque a tesoura paralela à coluna vertebral e proceda ao corte entre as vértebras e a crista ilíaca, conforme indicado pela linha pontilhada amarela na Figura 1A. Certifique-se de permanecer o mais perto possível das vértebras. Corte os músculos restantes para separar o membro do corpo.
NOTA: Deve haver pouca ou nenhuma resistência. - Repita do outro lado para desprender o segundo membro.
- Transfira os membros em uma superfície limpa e descarte o resto do corpo em conformidade com as diretrizes institucionais.
- Exponha os ossos pélvicos, femorais e tibiais removendo o máximo possível de tecido circundante com os fórceps e os bisturis.
- Desloque cuidadosamente a cabeça femoral do osso pélvico segurando a extremidade distal do fêmur com os fórceps enquanto corta suavemente os músculos ao redor da articulação com os bisturis. Mexa os ossos para facilitar a luxação.
- Raspe o músculo restante do osso pélvico e corte com um bisturi no meio da cavidade que segurou a cabeça do fêmur. O ípio é mantido como rico em progenitores hematopoiéticos, enquanto o lado triangular muito fino do osso é descartado, como mostrado na Figura 1C.
- Remova os tecidos residuais ao redor do ípio com o bisturi e coloque o osso limpo em PBS estéril complementado com 2% de soro de bezerro recém-nascido (PBS-2%NBCS).
- Usando uma tesoura, corte o pé da perna no tornozelo.
- Segure a parte inferior da tíbia com os fórceps e raspe o músculo em direção ao joelho. Descarte a fíbula e corte pelo planalto tibial com o bisturi. Coloque a tíbia em PBS-2%NBCS estérei.
- Remova os tecidos residuais ao redor do fêmur com bisturis.
- Segure o lado superior do fêmur com fórceps; coloque a lâmina do bisturi na base da rótula. Aplique uma força em direção à rótula paralela ao fêmur até o descolamento da rótula. Coloque o fêmur em PBS-2%NBCS estéril. A remoção da rótula proporciona um acesso limpo para a inserção da agulha para a lavagem da medula.
2. Esgotamento magnético das células positivas da linhagem
- Em um armário de fluxo laminar, transfira os ossos em uma placa de Petri estéril cheia de PBS-2% NBCS estéril.
- Com um bisturi cortado na cabeça dos fêmures.
- Encha uma seringa de 1 mL com PBS-2%NBCS e anexe uma agulha de 21 G à tomada.
- Encha um tubo de polipropileno de 5 mL com 2 mL de PBS-2% NBCS estéreis.
- Segure o fêmur com os fórceps; insira suavemente a agulha na ranhura esquerda após a remoção da rótula. Aplique rotação na agulha ao inserir para evitar a conexão da agulha. Certifique-se de que a agulha está completamente inserida no osso até o bisel.
- Transfira o osso com a agulha para o tubo contendo 2 mL de PBS-2%NBCS. Dispense e aspire o PBS-2% NBCS da seringa até que o osso esteja limpo.
- Retire a agulha do fêmur e insira-a no orifício do lado oposto onde estava a cabeça do fêmur. Dispense e aspire novamente o tampão e descarte o osso.
- Para a crista ilíaca e tíbia, segure o osso com os fórceps; insira suavemente a agulha no lado aberto. Aplique rotação na agulha ao inserir para evitar a conexão da agulha. Certifique-se de que a agulha está completamente inserida no osso até o bisel. Transfira o osso com a agulha para o tubo contendo 2 mL de PBS-2%NBCS. Dispense e aspire o PBS-2% NBCS da seringa até que o osso esteja limpo. Descarte os ossos.
NOTA: Ossos de até três ratos podem ser colocados no mesmo tubo. Acumule as suspensões de células. - Passe a suspensão celular agrupada através de uma tampa de filtro de célula de 40 μm colocada em um tubo de poliestireno estéril de 5 mL.
- Vá para a contagem das células.
NOTA: A contagem de células pode ser realizada com qualquer hemótmetro, usando o Trypan Blue para avaliação de viabilidade ou com qualquer contador automatizado de células. Um rato normalmente rende 105 ± 7 x 106 células. - Tire 100 μL da suspensão celular como Medula Óssea Total, adicione 500 μL de PBS-2% NBCS e guarde-o no gelo para o procedimento de coloração.
- Pelota a suspensão filtrada por centrifugação a 400 x g por 5 min a 4 °C e descarte o supernatante.
NOTA: Os glóbulos vermelhos podem ser reutilizados ressumando a pelota em solução de lise recém-preparada (1/10 em dH2O). Incubar por 5 minutos até que a suspensão fique clara e vermelha brilhante e adicione 10 volumes de PBS estéreis. Prossiga para lavar as células em PBS-2%NBCS por centrifugação a 400 x g para 5 minutos a 4 °C. Tenha cuidado ao remover o supernatante, pois a pelota celular está muito solta. Realize uma segunda lavagem com PBS-2%NBCS por centrifugação a 400 x g por 5 min a 4 °C e prossiga para a etapa 2.13. - Resuspend a pelota celular em coquetel de anticorpos primários recém-preparado com uma razão de 100 μL por 1 x 107 células. Incubar no gelo por 30-45 min.
| Anticorpo | Diluição |
| Gr-1-biotina | 1:500 |
| B220-biotina | 1:500 |
| Mac-1-biotina | 1:500 |
| CD3-biotina | 1:500 |
| CD4-biotina | 1:500 |
| CD5-biotina | 1:500 |
| CD8-biotina | 1:500 |
| TER119-biotina | 1:1000 |
| CD127-biotina | 1:500 |
Mesa 1.
- Despocalá-lo 10 μL da suspensão celular em um tubo de poliestireno estéril de 5 mL rotulado fração lin-pos. Adicione 90 μL de PBS-2% NBCS e guarde-o no gelo para o procedimento de coloração.
- Prossiga para lavar as células duas vezes com PBS-2%NBCS estéreis por centrifugação a 400 x g por 5 min a 4 °C. Certifique-se de fazer a última lavagem em um tubo de polipropileno de 5 mL estéril.
- Durante as etapas de lavagem, prepare as contas para o esgotamento magnético.
- Resuspenda as contas no frasco por um vórtice completamente para 30 s.
- Transfira um volume de contas correspondente a duas contas por célula alvo em um tubo de polipropileno de 5 mL.
- Lave as contas duas vezes com PBS-2%NBCS colocando o tubo no ímã e removendo o tampão de lavagem usando uma pipeta pasteur de vidro estéril.
- Resuspenda as contas em 500 μL de PBS-2NBCS estéreis.
- Resuspenque a pelota de células rotuladas em 250 μL de contas e misture suavemente por 5 minutos no gelo. Adicione 2 mL de PBS-2%NBCS e misture suavemente. Não agite o tubo.
- Coloque o tubo sobre o ímã por 2 minutos.
- Prosssiga para coletar a fração não magnética com uma pipeta pasteur de vidro estéril e adicioná-la aos 250 μL restantes de contas magnéticas. Sele o tubo com parafilme.
- Coloque o tubo em um rolo de tubo por 20 minutos a 4 °C.
- Adicione 2 mL de PBS-2%NBCS e misture suavemente. Não agite o tubo.
- Coloque o tubo no ímã por 2 minutos.
- Prossiga para coletar a fração não magnética em um tubo de polipropileno de 5 mL estéril rotulado Fração Lin-Neg com uma pipeta pasteur de vidro estéril.
- Pelotar as células por centrifugação a 400 x g por 5 min a 4 °C e remover o sobrenatante.
- Resuspende as células não magnéticas em 500 μL de PBS-2% NBCS estéreis.
- Vá para a contagem das células.
NOTA: Um rato normalmente rende 3,9 ± 1,1 x 106 células. A coloração típica de linhagem pré e pós-esgotamento são apresentadas na Figura 2B.
3. Classificação celular de progenitores de megacaiócitos por citometria de fluxo
- Pegue os tubos rotulados de Medula Óssea Total, Fração de Lin-Pos e Fração Lin-Neg.
- Prossiga para dividir o conteúdo do tubo Total Medula Óssea igualmente em seis tubos estéreis de 5 mL de poliestireno. Rotule os tubos com os números 1-6.
- Proceda para rotular o tubo Fração Lin-Pos com o número 7.
- Prossiga para dividir o conteúdo do tubo Fração Lin-Neg da seguinte forma.
- Transfira 50 μL para um tubo de poliestireno estéril de 5 mL contendo 250 μL de PBS-2%NBCS estéreis. Em seguida, divida seu conteúdo igualmente em tubos de poliestireno de 5 mL estéreis. Rotule esses tubos com os números 8-10.
- Os 450 μL restantes da suspensão celular da Fração Lin-Neg correspondem ao tubo com o número 11.
- Adicione os anticorpos aos tubos conforme descrito na Tabela 2.
| Tubo | Etiqueta | Coquetel de anticorpos |
| Medula Óssea Total | ||
| 1 | Controle não manchado | |
| 2 | Controle único manchado | CD45-FITC (1/200) |
| 3 | Controle único manchado | CD45-PE (1/200) |
| 4 | Controle único manchado | TER119-APC (1/200) |
| 5 | Controle único manchado | CD45-PECy7 (1/200) |
| 6 | Controle único manchado | Biotina CD45-APC-Cy7 (1/200) |
| Fração de Lin-Pos | ||
| 7 | Controle único manchado | Controle único manchado. Streptavidin-APC-Cy7 (1/500) |
| Fração Lin-Neg | ||
| 8 | Controle FMO FITC | c-kit-APC (1/200) + Sca-1-PE (1/200) + CD16/32-PE (1/200) + CD150-PECy7 (1/200) + Streptavidin-APC-Cy7 (1/500) |
| 9 | Controle do FMO PE | CD9-FITC (1/200) + c-kit-APC (1/200) + CD150-PECy7 (1/200) + Streptavidin-APC-Cy7 (1/500) |
| 10 | Controle FMO PECy7 | CD9-FITC (1/200) + c-kit-APC (1/200) + Sca-1-PE (1/200) + CD16/32-PE (1/200) + Streptavidin-APC-Cy7 (1/500) |
| 11 | Tubo positivo para classificação | CD9-FITC (1/200) + c-kit-APC (1/200) + Sca-1-PE (1/200) + CD16/32-PE (1/200) + CD150-PECy7 (1/200) + Streptavidin-APC-Cy7 (1/500) |
Mesa 2.
- Incubar no gelo por 30-45 min no escuro.
- Lave as células com PBS-2%NBCS estéreis por centrifugação a 400 x g por 5 min a 4 °C.
- Resuspende as pelotas de células da seguinte forma.
- Para os tubos de 1 a 10, resuspenque a pelota em 300 μL de PBS-2%NBCS estéril complementado com 7AAD (2,5 μg/mL final) (PBS-7AAD).
ATENÇÃO: 7AAD é um intercalante de DNA e, portanto, deve ser manuseado com EPI (luvas) apropriados. - Para tubo 11, resuspenque a pelota em PBS-7AAD estéril a uma concentração máxima de 5 x 106 células por mL e um volume mínimo de 1 mL.
- Para os tubos de 1 a 10, resuspenque a pelota em 300 μL de PBS-2%NBCS estéril complementado com 7AAD (2,5 μg/mL final) (PBS-7AAD).
- Prepare dois tubos de coleta de polipropileno rotulados MEP e MKp contendo 2 mL de PBS-2%NBCS.
NOTA: Alternativamente, as células podem ser coletadas em cultura média ou tampão de lise celular, dependendo da aplicação subsequente para as células classificadas. O uso de tubos de poliestireno não é recomendado devido a possível interferência com as gotículas carregadas que contêm as células de interesse. - Mantenha todos os tubos no gelo no escuro.
- Vá para a configuração do classificador de células.
- Use os tubos 1-7 para configurar tensão e compensação, tubos 7-10 para determinar os portões de classificação para as populações celulares de interesse e tubo 11 para triagem celular.
- Os primeiros passos da estratégia de gating visam excluir doublets e células mortas da análise, conforme descrito na Figura 3. Identifique células viáveis únicas e exiba o gráfico de pontos SSC-vs Lin-APC-Cy7 para confirmar a eficiência do esgotamento da linhagem. A partir do Lin- células um portão é definido para selecionar células positivas para c-kit e negativa ou fraca para Sca-1 e CD16/32. Um gráfico de ponto de expressão CD9 vs CD150 para as células selecionadas permite a identificação de quatro populações.
NOTA: As células MEP e MKp são positivas para CD150. Três níveis de expressão para CD9 podem ser definidos (neg, dim e alto). MKp expressam alto nível de CD9 e MEP express CD9 em um nível intermediário de intensidade de fluorescência. A população de MEP corresponde a Lin- c-Kit+ Sca-1-CD16/32-/dim CD150+ POPULAÇÃO CD9dim e MKp corresponde a Lin- c-Kit+ Sca-1-CD16/32-/dim CD150+ CD9dim eCD9 brilhante . A discriminação entre as populações de cd9 alta e CD9 dim para as células positivas CD150 é definida com base no nível máximo de expressão CD9 na população negativa cd150. Um mouse normalmente rende 5,3 ± 0,6 x 103 MKp e 27,2 ± 2,4 x 103 MEP.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
A análise fenotípica das células identificadas como MEP e MKp foram realizadas por citometria de fluxo. As células foram rotuladas com anticorpos conjugados de fluorescência para CD41a e CD42c, marcadores clássicos das linhagens megacariocíticas e plaquetas. Ambos os marcadores foram expressos pelas células da população de MKp, enquanto esses marcadores ainda não foram detectados na superfície das células da população do MEP(Figura 4Ai,4Aii). Poliploidia é uma marca registrada de megacaiócitos. O conteúdo de DNA das populações classificadas também foi analisado e demonstrado que as células são em sua maioria 2N para a população do MEP e uma pequena proporção das células MKp são 4N, mas ploidy maior não são significativamente detectadas nessas populações (Figura 4Aiii).
Para confirmar a identidade das populações celulares classificadas, foram realizados diversos ensaios de diferenciação para avaliar sua capacidade de diferenciação em direção às linhagens megacariocíticas e eritrócrias. Primeiro, foram realizados ensaios clonogênicos semissólidos para quantificar progenitor megacariocítico (CFU-MK) e progenitores eritródicos (BFU-E). O CFU-MK foi detectado tanto nas populações de PME quanto em MKp, mas não na outra população testada(Figura 4B). BFU-E não foram detectados na população de MKp, mas foram detectados na população de PME e na população decélulas dim CD150-CD9 dim(Figura 4C).
A diferenciação das células classificadas também foi seguida na cultura líquida na presença de uma baixa concentração de citocinas hematopoiéticas. Imagens representativas da observação microscópica no3º dia de diferenciação mostram que o MPE e o MKp produziram principalmente megacaiócitos identificados como células grandes (Figura 5Aiii,5Aiv). Os megacariócitos foram identificados utilizando-se a expressão CD41 e CD42c e representam 53,9 ± 10,4% e 82,0 ± 2,0% das células produzidas a partir de populações de células MEP e MKp, respectivamente (Figura 5B). Notavelmente, a ploidia dos megacaitos produzidos analisados pelo marcador de DNA Hoescht 33242, foi maior para o megacaito derivado da população de MKp em comparação com a população do MPE sugerindo um estado mais maduro(Figura 5C). Finalmente, as células produzidas a partir de cada população no3º dia foram submetidas a um ensaio de formação de proplato9. Observou-se que apenas as células derivadas da população de MKp eram capazes de emissão de proplatelet nesta condição(Figura 5D). Isso sugere um estágio de maturação mais avançado para a população de MKp. Além disso, quando a duração da cultura for estendida por até 4-5 dias, os megacaitos gerados pelo MEP também estenderão os proplatelets.

Figura 2: Esgotamento magnético das células de linhagem comprometidas (Lin). Primeiro, as células de medula óssea não variadas são rotuladas com o coquetel de anticorpos anti-rato de rato biotina- conjugado. As células são então incubadas com contas magnéticas revestidas de Ig anti-rato e, posteriormente, submetidas ao esgotamento magnético usando um ímã forte. O ímã manterá a fração lin+ magnética rotulada contra as paredes do tubo, enquanto a fração lin-negativa não magnética não rotulada será coletada em um novo tubo. (B) As células comprometidas com linhagem podem ser identificadas usando streptavidina conjugada fluorescente. Análise típica da expressão de linhagem em células antes do esgotamento magnético (medula óssea total) e após esgotamento magnético (Fração de Lin) N = 21. Clique aqui para ver uma versão maior desta figura.

Figura 3: Estratégia de gating de classificação celular. (A) Seleção dos eventos correspondentes a células únicas viáveis. (B) Seleção populacional de PMe e MKp. (i) A população de Lin Neg é selecionada a partir dos eventos de célula única viável. (ii) Progenitores que expressam c-kit e com baixa ou nenhuma expressão de antígeno Sca-1 ou CD16/32 são então selecionados. (iii) Os níveis de expressão CD9 e CD150 definem quatro populações celulares. MKp são definidos como CD9brilhanteCD150+ células, MEP são definidos como CD9dimCD150+. O limite mais alto para a expressão CD9 para a população CD9dimCD150+ baseia-se no nível máximo de expressão CD9 para as células CD150- Para efeito de análise, cd9dimCD150- células (Progenitors) e CD9-CD150- (Double Negative: DN) também foram classificados. (C) Os portões de triagem de células são baseados em controles de Fluorescência Menos Um (FMO). (i) Controle de FMO para portões CD9(ii) controle FMO para portões CD150. Clique aqui para ver uma versão maior desta figura.

Figura 4: Caracterização das populações de células MEP e MKp. (A) Análise de citometria de fluxo da expressão(i) CD41, (ii) conteúdo de CD42c e(iii)conteúdo de DNA (Hoechst33342) nas populações celulares CD9+CD150dim (MEP) e CD9+CD150bright (MKp). (B) Quantificação de CFU-MK das populações celulares classificadas. CD9-CD150- (DN), CD9+CD150- (Prog), CD9+CD150dim (MEP) e CD9+CD150brilhantes (MKp) populações de células foram classificadas e emplacaradas em gel de colágeno de acordo com as instruções do fabricante. (C) Quantificação do BFU-E das populações celulares classificadas. CD9-CD150- (DN), CD9+CD150- (Prog), CD9+CD150dim (MEP) e CD9+CD150brilhantes (MKp) populações de células foram classificadas e emplacaradas em gel de celulose de metila de acordo com as instruções do fabricante. Clique aqui para ver uma versão maior desta figura.

Figura 5: Potencial de diferenciação de MEP e MKp. CD9-CD150-(DN), CD9+CD150-(Prog), CD9+CD150dim(MEP) e CD9+CD150bright(MKp) populações celulares foram cultivadas por três dias em Stem Médio suplementar com SCF (7,5 ng/mL), Flt-3 (5 ng/mL), IL-6 (1 ng/mL) e TPO (10 ng/mL). (A) As imagens representativas foram tiradas por microscopia de contraste de fase. (B) A porcentagem de CD41+CD42c+ megacariócitos foi então avaliada por citometria de fluxo. N = 3. (C) O nível ploidy do CD41+CD42c+ megacariócitos foi então avaliado com Hoechst por flutometria de fluxo. N = 3. (D) Células produzidas no dia 3 a partir de CD9-CD150-(DN), CD9+CD150-(Prog), CD9+CD150dim(MEP) e populações celulares CD9+CD150brilhantes(MKp) foram colhidas e cultivadas em DMEM média suplementada com 50 ng/mL TPO, 10 % Soro de Bezerro Fetal, e 100 U/mL hirudin. (i) A proporção de megacaiócitos formadores de proplatelet na cultura foi determinada pela observação microscópica. Megacariócitos foram identificados com base no seu tamanho e/ou na presença de proplatelets. N = 2. (ii) Fotografia representativa de um proplatelet com megacaiócito por microscopia de contraste de fase. Clique aqui para ver uma versão maior desta figura.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
O método descrito neste artigo permite a extração e purificação do mouse MEP e MKp. Um parâmetro importante na otimização do protocolo foi a obtenção de número suficiente de células compatíveis com a maioria dos ensaios moleculares e celulares. A prática geral da coleta de ossos de camundongos para extração de células hematopoiéticas geralmente consiste na colheita dos fêmures e tíbias de cada rato. O osso pélvico, outra fonte de material hematopoiético, é, portanto, muitas vezes negligenciado. As razões para não coletar a crista ilíaca é o pouco conhecimento da anatomia interna do esqueleto do rato e o fato de que os usuários estão coletando clássicos de subidas traseiras cortando ou logo acima da cabeça do fêmur. Além disso, muitas vezes se supõe que as células de medula não seriam liberadas do osso ilílico da crista eficientemente devido à presença de trabeculas, que estão ausentes na parte central da tíbia e do fêmur. Neste protocolo, essas duas preocupações são abordadas e um método padronizado, confiável e eficaz é apresentado que permite a descarga adequada de cada osso hindlimb, incluindo o osso pélvico. Em particular, o uso do osso ilílico produz 105 ± 7 x 106 células por rato, enquanto o método clássico geralmente rende 42 ± 5 x 106 células. Um grande benefício desse método é a redução do número de animais necessários para a obtenção de um determinado número de células-alvo, proporcionando assim condições experimentais mais éticas e econômicas. Este procedimento também é aplicável para qualquer estudo que exija suspensão de células de medula óssea, como o isolamento de células-tronco hematopoiéticas10 ou a análise do comportamento progenitor hematopoiético em condições semissólidas11.
A classificação celular usando citometria de fluxo é uma técnica poderosa com uma grande vantagem em termos de pureza quando comparada às técnicas de enriquecimento magnético, mas o rendimento da classificação celular para populações raras pode ser menor do que para populações mais abundantes. O esgotamento magnético de células indesejadas de antemão é, portanto, um método útil para aumentar a frequência das células de interesse. Aqui, o procedimento de esgotamento magnético difere da recomendação do fabricante e leva em consideração a heterogeneidade na expressão dos marcadores de superfície utilizados para remover as células positivas de linhagem indesejadas. Com os protocolos típicos de uma etapa, as células positivas de linhagem com a maior expressão de marcadores de superfície irão saturar rapidamente as contas magnéticas. Eles impedirão a captura subsequente das células rotuladas remanescentes por competição e obstáculos estéricos, reduzindo significativamente a eficácia do esgotamento. Para resolver esse problema, foi projetado um esgotamento magnético de duas etapas que permite a remoção sequencial de todas as células positivas da linhagem, permitindo, portanto, esgotamentos rigorosos adequados para a classificação celular.
Outro parâmetro crítico para alcançar um esgotamento eficiente são as condições adequadas de rotulagem das células positivas de linhagem indesejadas. A titulação de anticorpos foi, portanto, especificamente otimizada. O uso de concentrações mais elevadas de anticorpos resultará em aumento excessivo das contas magnéticas e o esgotamento não específico de células de interesse linhagen-negativa. O uso de populações de células células MKp e MKp altamente purificadas é uma ferramenta importante no estudo da megacariopoiese. Para elucidar os mecanismos que controlam esse processo, o estudo investigou o papel do microambiente celular e demonstrou que uma população de células estrommal de células hepáticas fetais apoiaria a diferenciação do MKp10. A população classificada também poderia ser usada para análises moleculares ou unicelulares. Isso será particularmente relevante considerando a noção emergente de HSC12,13,14. A produção de megacaiócito diretamente da população do HSC sem a geração de um progenitor bipotente seria um caminho de emergência em resposta ao estresse13.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Os autores não declaram interesses financeiros concorrentes.
Acknowledgments
Os autores agradecem Monique Freund, Catherine Ziessel e Ketty pela assistência técnica. Este trabalho foi apoiado pela ARMESA (Association de Recherche et Développement en Médecine et Santé Publique), e por Grant ANR-17-CE14-0001-01 a Henri.de la. Salle.
Materials
| Name | Company | Catalog Number | Comments |
| 21-gauge needles | BD Microlance | 301155 | |
| 7AAD | Sigma-Aldrich | A9400 | |
| Antibody Gr-1-biotin | eBioscience | 13-5931-85 | Magnetic depletion |
| Antibody B220-biotin | eBioscience | 13-0452-85 | Magnetic depletion |
| Antibody Mac-1-biotin | eBioscience | 13-0112-85 | Magnetic depletion |
| Antibody CD3e-biotin | eBioscience | 13-0031-85 | Magnetic depletion |
| Antibody CD4-biotin | eBioscience | 13-9766-82 | Magnetic depletion |
| Antibody CD5-biotin | eBioscience | 13-0051-85 | Magnetic depletion |
| Antibody CD8a-biotin | eBioscience | 13-0081-85 | Magnetic depletion |
| Antibody TER119-biotin | eBioscience | 13-5921-85 | Magnetic depletion |
| Antibody CD127-biotin | eBioscience | 13-1271-85 | Magnetic depletion |
| Antibody CD45-FITC | eBioscience | 11-0451-85 | Cell sorting |
| Antibody CD45-PE | eBioscience | 12-0451-83 | Cell sorting |
| Antibody TER119-APC | eBioscience | 17-5921-83 | Cell sorting |
| Antibody CD45-PECy7 | eBioscience | 25-0451-82 | Cell sorting |
| Antibody CD45-biotin | eBioscience | 13-0451-85 | Cell sorting |
| Antibody CD9-FITC | eBioscience | 11-0091-82 | Cell sorting |
| Antibody c-kit-APC | eBioscience | 17-1171-83 | Cell sorting |
| Antibody Sca-1-PE | eBioscience | 12-5981-83 | Cell sorting |
| Antibody CD16/32-PE | eBioscience | 12-0161-83 | Cell sorting |
| Antibody CD150-PECy7 | eBioscience | 25-1502-82 | Cell sorting |
| Culture medium StemSpan-SFEM | Stemcell technologies | #09650 | |
| Dissection pad | Fisher Scientific | 10452395 | |
| DPBS | Life Technologies | 14190-094 | |
| Ethanol | vWR Chemicals | 83813.360 | |
| Forceps | Euronexia | P-120-AS | |
| Glass pasteur pipette | Dutscher | 42011 | |
| Magnet : DynaMag-5 | Thermo Fisher Scientific | 12303D | |
| Magnetic beads: Dynabeads Sheep Anti-Rat IgG | Thermo Fisher Scientific | 11035 | |
| Megacult | Stemcell technologies | #04970 | |
| MethoCult SF M3436 | Stemcell technologies | #03436 | |
| Newborn Calf Serum | Dutscher | 50750-500 | |
| Red Cell Lysis solution | BD Bioscience | 555899 | |
| Scalpels | Fisher Scientific | 12308009 | |
| Scissors | Euronexia | C-165-ASB | |
| Sterile 1 mL syringes | BD Bioscience | 303172 | |
| Sterile 15mL tubes | Sarstedt | 62.554.502 | |
| Sterile 5mL polypropylene tubes | Falcon | 352063 | |
| Sterile 5mL polystyrene tubes | Falcon | 352054 | |
| Sterile tubes with 70µm cell strainer cap | Falcon | 352235 | |
| Sterile petri dish | Falcon | 353003 | |
| Streptavidin-APC-Cy7 | BD Biosciences | 554063 | Cell sorting |
| Tube roller | Benchmark Scientific | R3005 |
References
- Kaushansky, K. Historical review: megakaryopoiesis and thrombopoiesis. Blood. 111 (3), 981-986 (2008).
- Akashi, K., Traver, D., Miyamoto, T., Weissman, I. L. A clonogenic common myeloid progenitor that gives rise to all myeloid lineages. Nature. 404 (6774), 193-197 (2000).
- Debili, N., et al. Characterization of a bipotent erythro-megakaryocytic progenitor in human bone marrow. Blood. 88 (4), 1284-1296 (1996).
- Forsberg, E. C., Serwold, T., Kogan, S., Weissman, I. L., Passegué, E. New evidence supporting megakaryocyte-erythrocyte potential of flk2/flt3+ multipotent hematopoietic progenitors. Cell. 126 (2), 415-426 (2006).
- Vannucchi, A. M., et al. Identification and characterization of a bipotent (erythroid and megakaryocytic) cell precursor from the spleen of phenylhydrazine-treated mice. Blood. 95 (8), 2559-2568 (2000).
- Pronk, C. J., et al. Elucidation of the phenotypic, functional, and molecular topography of a myeloerythroid progenitor cell hierarchy. Cell Stem Cell. 1 (4), 428-442 (2007).
- Nakorn, T. N., Miyamoto, T., Weissman, I. L. Characterization of mouse clonogenic megakaryocyte progenitors. Proceedings of the National Academy of Sciences of the United States of America. 100 (1), 205-210 (2003).
- Ng, A. P., et al. Characterization of thrombopoietin (TPO)-responsive progenitor cells in adult mouse bone marrow with in vivo megakaryocyte and erythroid potential. Proceedings of the National Academy of Sciences of the United States of America. 109 (7), 2364-2369 (2012).
- Strassel, C., et al. Hirudin and heparin enable efficient megakaryocyte differentiation of mouse bone marrow progenitors. Experimental Cell Research. 318 (1), 25-32 (2012).
- Brouard, N., et al. A unique microenvironment in the developing liver supports the expansion of megakaryocyte progenitors. Blood Advances. 1 (21), 1854-1866 (2017).
- Boscher, J., Gachet, C., Lanza, F., Léon, C. Megakaryocyte culture in 3D methylcellulose-based hydrogel to improve cell maturation and study the impact of stiffness and confinement. Journal of Visualized Experiments:JOVE. , (2021).
- Sanjuan-Pla, A., et al. Platelet-biased stem cells reside at the apex of the haematopoietic stem-cell hierarchy. Nature. 502 (7470), 232-236 (2013).
- Haas, S., et al. Inflammation-driven fast-track differentiation of HSCs into the megakaryocytic lineage. Experimental Hematology. 42 (8), 14 (2014).
- Shin, J. Y., Hu, W., Naramura, M., Park, C. Y. High c-Kit expression identifies hematopoietic stem cells with impaired self-renewal and megakaryocytic bias. The Journal of Experimental Medicine. 211 (2), 217-231 (2014).
Tags
Biologia Edição 171Erratum
Formal Correction: Erratum: Isolation of Mouse Megakaryocyte Progenitors
Posted by JoVE Editors on 07/28/2021.
Citeable Link.
An erratum was issued for: Isolation of Mouse Megakaryocyte Progenitors. A figure was updated.
Figure 2 was updated from:
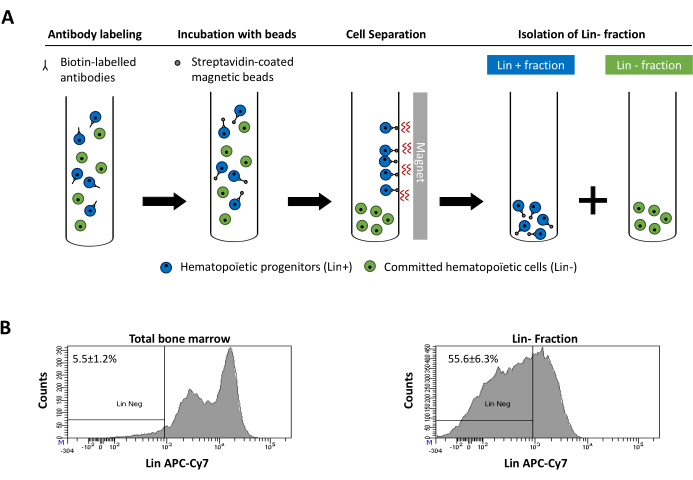
Figure 2: Magnetic depletion of lineage committed (Lin) cells. (A) Schematic representation of the magnetic depletion protocol. First, unsorted bone marrow cells are labeled with the biotin-conjugated rat anti-mouse antibody cocktail. Cells are then incubated with anti-rat Ig coated magnetic beads and subsequently subjected to the magnetic depletion using a strong magnet. The magnet will retain the labeled magnetic Lin+ fraction against the tube walls, while the unlabeled non-magnetic Lin- negative fraction will be collected in a new tube. (B) Lineage committed cells can be identified using fluorescent conjugated streptavidin. Typical analysis of the lineage expression in cells prior to magnetic depletion (total bone marrow) and after magnetic depletion (Lin- Fraction) N = 21. Please click here to view a larger version of this figure.
to:
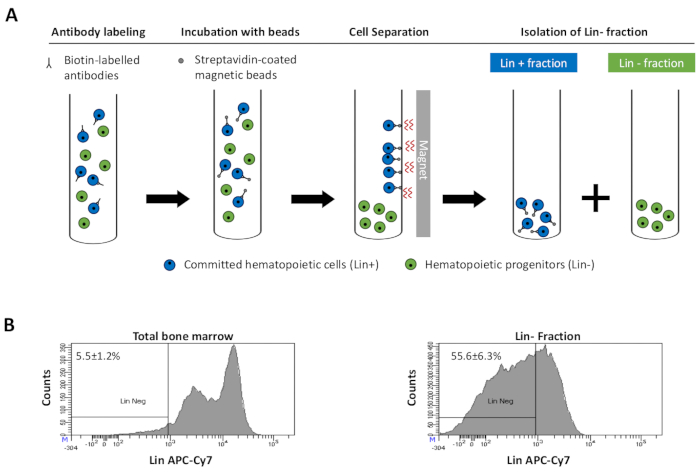
Figure 2: Magnetic depletion of lineage committed (Lin) cells. (A) Schematic representation of the magnetic depletion protocol. First, unsorted bone marrow cells are labeled with the biotin-conjugated rat anti-mouse antibody cocktail. Cells are then incubated with anti-rat Ig coated magnetic beads and subsequently subjected to the magnetic depletion using a strong magnet. The magnet will retain the labeled magnetic Lin+ fraction against the tube walls, while the unlabeled non-magnetic Lin- negative fraction will be collected in a new tube. (B) Lineage committed cells can be identified using fluorescent conjugated streptavidin. Typical analysis of the lineage expression in cells prior to magnetic depletion (total bone marrow) and after magnetic depletion (Lin- Fraction) N = 21. Please click here to view a larger version of this figure.



