ERRATUM NOTICE
Important: There has been an erratum issued for this article. Read more …
Summary
Cette méthode décrit la purification par cytométrie en flux de MEP et de MKp à partir de fémurs, de tibias et d’os pelviens de souris.
Abstract
Les mégacaryocytes de la moelle osseuse sont de grandes cellules polyploïdes qui assurent la production de plaquettes sanguines. Ils proviennent de cellules souches hématopoïétiques par le biais de la mégacaryopoïèse. Les dernières étapes de ce processus sont complexes et impliquent classiquement les progéniteurs bipotents mégacaryocytes-érythrocytaires (MEP) et les progéniteurs mégacaryocytaires unipotents (MKp). Ces populations précèdent la formation de mégacaryocytes de bonne foi et, à ce titre, leur isolement et leur caractérisation pourraient permettre une analyse robuste et impartiale de la formation de mégacaryocytes. Ce protocole présente en détail la procédure de collecte des cellules hématopoïétiques de la moelle osseuse de souris, l’enrichissement des progéniteurs hématopoïétiques par épuisement magnétique et enfin une stratégie de tri cellulaire qui donne des populations de MEP et de MKp hautement purifiées. Tout d’abord, les cellules de la moelle osseuse sont prélevées sur le fémur, le tibia et la crête iliaque, un os qui contient un grand nombre de progéniteurs hématopoïétiques. L’utilisation d’os de la crête iliaque augmente considérablement le nombre total de cellules obtenues par souris et contribue ainsi à une utilisation plus éthique des animaux. Une déplétion de la lignée magnétique a été optimisée à l’aide de billes magnétiques de 450 nm permettant un tri cellulaire très efficace par cytométrie en flux. Enfin, le protocole présente la stratégie d’étiquetage et de contrôle pour le tri des deux populations de progéniteurs mégacarytaires hautement purifiés : MEP (Lin-Sca-1-c-Kit+CD16/32-CD150+CD9dim) et MKp (Lin- Sca-1-c-Kit+CD16/32-CD150+CD9bright ). Cette technique est facile à mettre en œuvre et fournit suffisamment de matériel cellulaire pour effectuer i) la caractérisation moléculaire pour une connaissance plus approfondie de leur identité et de leur biologie, ii) les tests de différenciation in vitro, qui permettront de mieux comprendre les mécanismes de maturation des mégacaryocytes, ou iii) les modèles in vitro d’interaction avec leur microenvironnement.
Introduction
Les plaquettes sanguines sont produites par les mégacaryocytes. Ces grandes cellules polyploïdes sont situées dans la moelle osseuse et comme pour toutes les cellules sanguines, elles sont dérivées de cellules souches hématopoïétiques (CSH)1. La voie classique de production de mégacaryocytes dans la moelle osseuse provient du HSC et implique la génération de différents progéniteurs qui restreignent progressivement leur potentiel de différenciation2. Le premier progéniteur signant l’engagement envers la lignée mégacaryocytaire est le Megakaryocyte-Erythrocyte Progenitor (MEP), un progéniteur bipotent capable de produire à la fois des cellules érythroïdes et des mégacaryocytes3,4,5. Le MEP produit alors un progéniteur/précurseur unipotent (MKp) qui va se différencier en un mégacaryocyte mature capable de produire des plaquettes. Les mécanismes impliqués dans la génération de ces progéniteurs, ainsi que leur différenciation et leur maturation en mégacaryocytes sont complexes et seulement partiellement compris. En outre, l’hétérogénéité de la population de MEP en termes de potentiel de différenciation et le niveau d’engagement intrinsèque de ces cellules ne sont pas encore clairs. Pour déchiffrer ces processus, il est essentiel d’obtenir (ou d’avoir accès à) des populations purifiées de MEP et de MKp pour des analyses moléculaires fines et unicellulaires.
Plusieurs études ont démontré des combinaisons particulières de marqueurs de surface cellulaire pour l’identification des progéniteurs engagés dans la lignée mégacaryocytaire chez la souris6,7,8. À partir de ceux-ci, une méthode a été conçue permettant la purification du MEP et du MKp à partir de souris. Cette méthode a été optimisée pour obtenir des cellules en nombre et en qualité adéquats pour un grand nombre d’essais. Avec des considérations éthiques à l’esprit, et afin de minimiser le nombre d’animaux impliqués dans les expériences, nous avons obtenu de prélever la moelle osseuse du fémur et du tibia, ainsi que de la crête iliaque. Cet os contient une fréquence et un nombre élevés de progéniteurs hématopoïétiques et est la plupart du temps endommagé lors de la récolte d’os longs. Présenté ici est une méthode détaillée pour la collecte fiable de cet os.
Le deuxième critère d’optimisation est de produire des populations cellulaires hautement purifiées. Le tri cellulaire activé par fluorescence (FACS) est une méthode de choix afin d’obtenir des populations purifiées de cellules d’intérêt. Cependant, de faibles rendements sont atteints lorsque la population cellulaire d’intérêt est très rare. Des procédures d’enrichissement sont donc nécessaires. Dans ce protocole, une procédure de sélection négative a été choisie à l’aide de billes magnétiques.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
Les protocoles impliquant des animaux ont été réalisés conformément au Comité CREMEAS d’éthique de l’expérimentation animale de l’Université de Strasbourg (Comité Régional d’Ethique en Matière d’Expérimentation Animale Strasbourg. Numéro de permis : E67-482-10).
1. Collecte d’os de souris
- Sacrifier l’animal conformément aux directives institutionnelles.
NOTE: Les données présentées dans ce manuscrit ont été obtenues à partir de souris C57Bl/6 âgées de 8 à 12 semaines. Le nombre de cellules obtenues et la fréquence des populations citées peuvent varier en fonction de l’âge et de la souche de souris. - Vaporisez le corps avec 70% d’éthanol.
- À l’aide de ciseaux, faites une incision de 0,5 à 1 cm de la peau perpendiculairement à la colonne vertébrale et déchirez la peau autour de tout le corps. Tirez la peau du bas du corps et retirez la peau.
- Placez l’animal sur le tampon de dissection, face vers le bas. Localisez les os pelviens en faisant glisser vos doigts le long de la colonne vertébrale exposée de haut en bas. Pour localiser la crête iliaque, identifiez la petite bosse dans la région lombaire près des membres postérieurs (la région antérosupérieur de l’os pelvien). La figure 1A,B présente une représentation schématique de l’anatomie de la souris.

Figure 1: Anatomie de la souris. (A) Radiographie de la souris montrant les os des membres postérieurs. Notez l’espace entre l’os pelvien et la colonne vertébrale (flèche jaune), où les ciseaux doivent être insérés pour séparer correctement les membres postérieurs du corps de la souris (ligne pointillée jaune). (B) Représentation schématique des os d’intérêt riches en moelle osseuse. Les os pelviens sont représentés en rouge, les fémurs en violet et les tibias en vert. (C) Représentation schématique de l’os pelvien de la souris. L’ilium correspond à la partie riche en moelle de l’os pelvien et est surligné en rouge. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Placez les ciseaux parallèlement à la colonne vertébrale contre les vertèbres et près de la bosse de la crête iliaque. Procédez à couper les muscles le long du côté de la colonne vertébrale au-dessus de l’os pelvien en faisant glisser les ciseaux le long des vertèbres jusqu’à la queue.
REMARQUE: Cette première section de muscles peut également être effectuée à l’aide d’une lame de scalpel. - Placez les ciseaux parallèlement à la colonne vertébrale et procédez à la coupe entre les vertèbres et la crête iliaque, comme l’indique la ligne pointillée jaune sur la figure 1A. Assurez-vous de rester le plus près possible des vertèbres. Coupez les muscles restants pour détacher le membre du corps.
REMARQUE: Il devrait y avoir peu ou pas de résistance. - Répétez de l’autre côté pour détacher le deuxième membre.
- Transférez les membres sur une surface propre et jetez le reste du corps conformément aux directives institutionnelles.
- Exposez les os pelviens, fémoraux et tibias en enlevant autant de tissu environnant que possible avec la pince et les scalpels.
- Procédez à la luxation soigneuse de la tête fémorale de l’os pelvien en tenant l’extrémité distale du fémur avec la pince tout en tranchant doucement les muscles autour de l’articulation avec les scalpels. Remuez les os pour faciliter la luxation.
- Grattez le muscle restant de l’os pelvien et coupez avec un scalpel au milieu de la cavité qui maintenait la tête du fémur. L’ilium est conservé car il est riche en progéniteurs hématopoïétiques tandis que le côté triangulaire très mince de l’os est jeté, comme le montre la figure 1C.
- Enlevez les tissus résiduels autour de l’ilium avec le scalpel et placez l’os nettoyé dans du PBS stérile complété par du sérum de veau nouveau-né à 2% (PBS-2% NBCS).
- À l’aide de ciseaux, coupez le pied de la jambe à la cheville.
- Tenez la partie inférieure du tibia avec la pince et grattez le muscle vers le genou. Jetez les fibules et coupez le plateau tibial avec le scalpel. Placez le tibia dans du PBS-2% NBCS stérile.
- Enlevez les tissus résiduels autour du fémur avec des scalpels.
- Tenez la face supérieure du fémur avec une pince; placez la lame du scalpel à la base de la rotule. Appliquez une force vers la rotule parallèlement au fémur jusqu’au détachement de la rotule. Placez le fémur dans du PBS-2% NBCS stérile. Le retrait de la rotule fournit un accès propre pour l’insertion de l’aiguille pour le rinçage de la moelle.
2. Épuisement magnétique des cellules positives de la lignée
- Dans une armoire à flux laminaire, transférer les os dans une boîte de Petri stérile remplie de PBS-2% NBCS stérile.
- Avec un scalpel coupé la tête des fémurs.
- Remplissez une seringue de 1 mL avec du PBS-2% NBCS stérile et fixez une aiguille de 21 G à la sortie.
- Remplissez un tube en polypropylène de 5 mL avec 2 mL de PBS-2% NBCS stérile.
- Tenez le fémur avec la pince; insérez doucement l’aiguille dans la rainure laissée après le retrait de la rotule. Appliquez une rotation sur l’aiguille lors de l’insertion pour éviter le colmatage de l’aiguille. Assurez-vous que l’aiguille est complètement insérée dans l’os jusqu’au biseau.
- Transférer l’os avec l’aiguille dans le tube contenant 2 mL de PBS-2% NBCS. Distribuer et aspirer le PBS-2%NBCS de la seringue jusqu’à ce que l’os soit clair.
- Retirez l’aiguille du fémur et insérez-la dans le trou du côté opposé où se trouvait la tête du fémur. Distribuer et aspirer à nouveau le tampon et jeter l’os.
- Pour la crête iliaque et le tibia, tenez l’os avec la pince; insérez doucement l’aiguille dans le côté ouvert. Appliquez une rotation sur l’aiguille lors de l’insertion pour éviter le colmatage de l’aiguille. Assurez-vous que l’aiguille est complètement insérée dans l’os jusqu’au biseau. Transférer l’os avec l’aiguille dans le tube contenant 2 mL de PBS-2% NBCS. Distribuer et aspirer le PBS-2%NBCS de la seringue jusqu’à ce que l’os soit clair. Jetez les os.
REMARQUE: Les os de jusqu’à trois souris peuvent être rincés dans le même tube. Regrouper les suspensions cellulaires. - Passer la suspension cellulaire regroupée à travers un bouchon de crépine cellulaire de 40 μm placé sur un tube stérile en polystyrène de 5 mL.
- Procédez au comptage des cellules.
REMARQUE: Le comptage cellulaire peut être effectué avec n’importe quel hémocytomètre, en utilisant Trypan Blue pour l’évaluation de la viabilité, ou avec n’importe quel compteur cellulaire automatisé. Une souris donne généralement 105 ± 7 x 106 cellules. - Essuyez 100 μL de la suspension cellulaire sous forme de moelle osseuse totale, ajoutez 500 μL de PBS-2% NBCS et conservez-la sur de la glace pour la procédure de coloration.
- Pastillez la suspension filtrée par centrifugation à 400 x g pendant 5 min à 4 °C et jetez le surnageant.
REMARQUE: Les globules rouges peuvent être lysés en remettant la pastille dans une solution de lyse fraîchement préparée (1/10ème en dH2O). Incuber pendant 5 min jusqu’à ce que la suspension devienne claire et rouge vif et ajouter 10 volumes de PBS stérile. Procéder au lavage des cellules en PBS-2% NBCS par centrifugation à 400 x g pendant 5 minutes à 4 °C. Soyez prudent lorsque vous retirez le surnageant car la pastille de cellule est très lâche. Effectuez un deuxième lavage avec PBS-2% NBCS par centrifugation à 400 x g pendant 5 min à 4 °C et passez à l’étape 2.13. - Resuspendez la pastille cellulaire dans un cocktail d’anticorps primaires fraîchement préparé avec un rapport de 100 μL pour 1 x 107 cellules. Incuber sur de la glace pendant 30-45 min.
| Anticorps | Dilution |
| Gr-1-biotine | 1:500 |
| B220-biotine | 1:500 |
| Mac-1-biotine | 1:500 |
| CD3-biotine | 1:500 |
| CD4-biotine | 1:500 |
| CD5-biotine | 1:500 |
| CD8-biotine | 1:500 |
| TER119-biotine | 1:1000 |
| CD127-biotine | 1:500 |
Tableau 1.
- Prendre 10 μL de la suspension cellulaire dans un tube stérile en polystyrène de 5 mL marqué Lin-Pos Fraction. Ajouter 90 μL de PBS-2%NBCS et l’économiser sur de la glace pour la procédure de coloration.
- Procéder au lavage des cellules deux fois avec du PBS-2% NBCS stérile par centrifugation à 400 x g pendant 5 min à 4 °C. Assurez-vous de faire le dernier lavage dans un tube stérile en polypropylène de 5 mL.
- Pendant les étapes de lavage, préparez les perles pour l’épuisement magnétique.
- Resussaisissez les perles dans le flacon en les faisant soigneusement tourbillon pendant 30 s.
- Transférer un volume de billes correspondant à deux billes par cellule cible dans un tube en polypropylène de 5 mL.
- Lavez les billes deux fois avec PBS-2%NBCS en plaçant le tube sur l’aimant et en retirant le tampon de lavage à l’aide d’une pipette Pasteur en verre stérile.
- Resuspendez les billes dans 500 μL de PBS-2NBCS stérile.
- Remettez enseillement la pastille de cellules étiquetées dans 250 μL de billes et mélangez doucement pendant 5 min sur de la glace. Ajouter 2 mL de PBS-2% NBCS stérile et mélanger doucement. Ne secouez pas le tube.
- Placez le tube sur l’aimant pendant 2 min.
- Procéder à la collecte de la fraction non magnétique avec une pipette Pasteur en verre stérile et l’ajouter sur les 250 μL restants de billes magnétiques. Scellez le tube avec un parafilm.
- Placez le tube sur un rouleau à tube pendant 20 min à 4 °C.
- Ajouter 2 mL de PBS-2% NBCS stérile et mélanger doucement. Ne secouez pas le tube.
- Placez le tube dans l’aimant pendant 2 min.
- Procéder à la collecte de la fraction non magnétique dans un tube stérile en polypropylène de 5 mL étiqueté Lin-Neg Fraction avec une pipette Pasteur en verre stérile.
- Abreuler les cellules par centrifugation à 400 x g pendant 5 min à 4 °C et retirer le surnageant.
- Resuspendez les cellules non magnétiques dans 500 μL de PBS-2% NBCS stérile.
- Procédez au comptage des cellules.
REMARQUE: Une souris donne généralement 3,9 ± 1,1 x 106 cellules. Les colorations typiques de la lignée avant et après l’épuisement sont présentées à la figure 2B.
3. Tri cellulaire des progéniteurs mégacarytaires par cytométrie en flux
- Prenez les tubes étiquetés Total Bone Marrow, Lin-Pos Fraction et Lin-Neg Fraction.
- Procéder à la division égale du contenu du tube Total Bone Marrow en six tubes stériles en polystyrène de 5 mL. Étiquetez les tubes avec les chiffres 1 à 6.
- Procédez à l’étiquetage du tube Lin-Pos Fraction avec le chiffre 7.
- Procédez à la division du contenu du tube Lin-Neg Fraction comme suit.
- Transférer 50 μL dans un tube stérile en polystyrène de 5 mL contenant 250 μL de PBS-2% NBCS stérile. Ensuite, divisez son contenu également en 3 tubes stériles en polystyrène de 5 mL. Étiquetez ces tubes avec les chiffres 8-10.
- Les 450 μL restants de suspension de cellules de fraction Lin-Neg correspondent au tube portant le numéro 11.
- Ajouter les anticorps aux tubes comme décrit dans le tableau 2.
| Tube | Étiquette | Cocktail d’anticorps |
| Moelle osseuse totale | ||
| 1 | Contrôle non taché | |
| 2 | Contrôle à simple taché | CD45-FITC (1/200) |
| 3 | Contrôle à simple taché | CD45-PE (1/200) |
| 4 | Contrôle à simple taché | TER119-APC (1/200) |
| 5 | Contrôle à simple taché | CD45-PECy7 (1/200) |
| 6 | Contrôle à simple taché | BIOTINE CD45-APC-Cy7 (1/200) |
| Lin-Pos Fraction | ||
| 7 | Contrôle à simple taché | Contrôle à simple taché. Streptavidine-APC-Cy7 (1/500) |
| Lin-Neg Fraction | ||
| 8 | Contrôle FMO FITC | c-kit-APC (1/200) + Sca-1-PE (1/200) + CD16/32-PE (1/200) + CD150-PECy7 (1/200) + Streptavidin-APC-Cy7 (1/500) |
| 9 | Contrôle FMO PE | CD9-FITC (1/200) + c-kit-APC (1/200) + CD150-PECy7 (1/200) + Streptavidin-APC-Cy7 (1/500) |
| 10 | Contrôle FMO PECy7 | CD9-FITC (1/200) + c-kit-APC (1/200) + Sca-1-PE (1/200) + CD16/32-PE (1/200) + Streptavidine-APC-Cy7 (1/500) |
| 11 | Tube positif pour le tri | CD9-FITC (1/200) + c-kit-APC (1/200) + Sca-1-PE (1/200) + CD16/32-PE (1/200) + CD150-PECy7 (1/200) + Streptavidin-APC-Cy7 (1/500) |
Tableau 2.
- Incuber sur de la glace pendant 30-45 min dans l’obscurité.
- Laver les cellules avec du PBS-2% NBCS stérile par centrifugation à 400 x g pendant 5 min à 4 °C.
- Resuspendez les pastilles de cellules comme suit.
- Pour les tubes 1 à 10, resuspendez la pastille dans 300 μL de PBS-2% NBCS stérile complété par 7AAD (2,5 μg/mL final) (PBS-7AAD).
ATTENTION : Le 7AAD est un intercalant ADN et doit donc être manipulé avec un EPI (gants) approprié. - Pour le tube 11, resuspendez la pastille dans du PBS-7AAD stérile à une concentration maximale de 5 x 106 cellules par mL et un volume minimum de 1 mL.
- Pour les tubes 1 à 10, resuspendez la pastille dans 300 μL de PBS-2% NBCS stérile complété par 7AAD (2,5 μg/mL final) (PBS-7AAD).
- Préparer deux tubes de collecte de polypropylène étiquetés MEP et MKp contenant 2 mL de PBS-2% NBCS.
REMARQUE: Alternativement, les cellules peuvent être collectées dans un milieu de culture ou un tampon de lyse cellulaire en fonction de l’application ultérieure pour les cellules triées. L’utilisation de tubes en polystyrène n’est pas recommandée en raison d’interférences possibles avec les gouttelettes chargées contenant les cellules d’intérêt. - Gardez tous les tubes sur la glace dans l’obscurité.
- Passez à la configuration du trieur de cellules.
- Utilisez les tubes 1 à 7 pour régler la tension et la compensation, les tubes 7 à 10 pour déterminer les portes de tri pour les populations cellulaires d’intérêt et le tube 11 pour le tri cellulaire.
- Les premières étapes de la stratégie de gating visent à exclure les doublets et les cellules mortes de l’analyse, comme décrit à la figure 3. Identifiez des cellules viables uniques et affichez le diagramme à points SSC-vs Lin-APC-Cy7 pour confirmer l’efficacité de l’épuisement de la lignée. À partir des cellulesLin- une porte est définie pour sélectionner les cellules positives pour c-kit et négatives ou dim pour Sca-1 et CD16/32. Un diagramme à points d’expression CD9 vs CD150 pour les cellules sélectionnées permet d’identifier quatre populations.
REMARQUE: Les cellules MEP et MKp sont toutes deux positives pour CD150. Trois niveaux d’expression pour CD9 peuvent être définis (neg, dim et high). MKp exprime un niveau élevé de CD9 et MEP exprime CD9 à un niveau d’intensité de fluorescence intermédiaire. La population MEP correspond à Lin- c-Kit+ Sca-1-CD16/32-/dim CD150+ CD9dim et la population MKp correspond à Lin- c-Kit+ Sca-1-CD16/32-/dim CD150+ CD9lumineux. La discrimination entre les populations CD9 élevées et CD9 faibles pour les cellules CD150 positives est établie en fonction du niveau maximal d’expression de CD9 dans la population CD150 négative. Une souris donne généralement 5,3 ± 0,6 x 103 MKp et 27,2 ± 2,4 x 103 MEP.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
L’analyse phénotypique des cellules identifiées comme MEP et MKp a été réalisée par cytométrie en flux. Les cellules ont été marquées avec des anticorps conjugués par fluorescence contre CD41a et CD42c, marqueurs classiques des lignées mégacaryocytaires et plaquettaires. Les deux marqueurs ont été exprimés par les cellules de la population MKp alors que ces marqueurs ne sont pas encore détectés à la surface des cellules de la population MEP (Figure 4Ai, 4Aii). La polyploïdie est une caractéristique des mégacaryocytes. La teneur en ADN des populations triées a également été analysée et a démontré que les cellules sont principalement 2N pour la population MEP et qu’une petite proportion des cellules MKp sont 4N, mais une ploïdie plus élevée n’est pas détectée de manière significative dans ces populations (Figure 4Aiii).
Afin de confirmer l’identité des populations cellulaires triées, plusieurs tests de différenciation ont été effectués pour évaluer leur capacité à se différencier vers les lignées mégacaryocytaires et érythroïdes. Tout d’abord, des tests clonogènes semi-solides ont été effectués pour quantifier les progéniteurs mégacaryocytaires (CFU-MK) et érythroïdes (BFU-E). Les UFC-MK ont été détectés dans les populations meP et MKp, mais pas dans les autres populations testées(figure 4B). Les BFU-E n’ont pas été détectés dans la population MKp mais ont été détectés dans la population MEP et la population de cellulesDIM CD150-CD9(Figure 4C).
La différenciation des cellules triées a également été suivie en culture liquide en présence d’une faible concentration de cytokines hématopoïétiques. Des images représentatives de l’observation microscopique au3ème jour de différenciation montrent que MEP et MKp ont produit principalement des mégacaryocytes qui sont identifiés comme de grandes cellules (Figure 5Aiii,5Aiv). Les mégacaryocytes ont été identifiés à l’aide de l’expression de CD41 et CD42c et représentent respectivement 53,9 ± 10,4 % et 82,0 ± 2,0 % des cellules produites à partir de populations de cellules MEP et MKp(figure 5B). Remarquablement, la ploïdie des mégacaryocytes produits analysés à l’aide du marqueur ADN Hoescht 33242, était plus grande pour le mégacaryocyte dérivé de la population MKp par rapport à la population MEP suggérant un état plus mature(Figure 5C). Enfin, les cellules produites à partir de chaque population le3ème jour ont été soumises à un test de formation de proplaquettaires9. Il a été observé que seules les cellules dérivées de la population MKp étaient capables d’émission de proplaquettaires dans cet état(figure 5D). Cela suggère un stade de maturation plus avancé pour la population de MKp. De plus, lorsque la durée de la culture est prolongée jusqu’à 4-5 jours, les mégacaryocytes générés à partir de MEP prolongeront également les proplaquettaires.

Figure 2: Épuisement magnétique des cellules de lignée engagées (Lin). (A) Représentation schématique du protocole d’épuisement magnétique. Tout d’abord, les cellules non triées de la moelle osseuse sont étiquetées avec le cocktail d’anticorps anti-souris de rat conjugué à la biotine. Les cellules sont ensuite incubées avec des billes magnétiques recouvertes d’Ig anti-rat et ensuite soumises à l’épuisement magnétique à l’aide d’un aimant puissant. L’aimant retiendra la fraction Magnétique Lin+ étiquetée contre les parois du tube, tandis que la fraction Lin- négative non magnétique non étiquetée sera collectée dans un nouveau tube. (B) Les cellules engagées dans la lignée peuvent être identifiées à l’aide de la streptavidine conjuguée fluorescente. Analyse typique de l’expression de la lignée dans les cellules avant l’épuisement magnétique (moelle osseuse totale) et après l’épuisement magnétique (fraction Lin) N = 21. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3: Stratégie de contrôle du tri cellulaire. (A) Sélection des événements correspondant à des cellules individuelles viables. (B) Sélection de la population des députés européens et des députés. (i) La population lin neg est sélectionnée parmi les événements viables d’une seule cellule. (ii) Les progéniteurs exprimant le c-kit et avec une expression faible ou nulle de l’antigène Sca-1 ou CD16/32 sont ensuite sélectionnés. (iii) Les niveaux d’expression de CD9 et CD150 définissent quatre populations cellulaires. MKp sont définis comme CD9brightCD150+ cellules, MEP sont définis comme CD9dimCD150+. La limite supérieure pour l’expression de CD9 pour la population cd9dimCD150+ est basée sur le niveau d’expression maximal de CD9 pour lescellules CD150-. Aux fins de l’analyse, cd9dimCD150- cellules (progéniteurs) et CD9-CD150- (double négatif: DN) ont également été triés. (C) Les portes de tri des cellules sont basées sur des contrôles de fluorescence moins un (FMO). (i) Contrôle FMO pour les portes CD9 (ii) Contrôle FMO pour portes CD150. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4: Caractérisation des populations de cellules MEP et MKp. (A) Analyse par cytométrie en flux de ( i )l’expressionde CD41, (ii) l’expression de CD42c et (iii) la teneur en ADN (Hoechst33342) dans les populations cellulaires CD9+CD150dim (MEP) et CD9+CD150bright (MKp). (B) Quantification de l’UFC-MK à partir des populations cellulaires triées. Les populations de cellules CD9-CD150- (DN), CD9+CD150- (Prog), CD9+CD150dim (MEP) et CD9+CD150bright (MKp) ont été triées et plaquées dans du gel de collagène conformément aux instructions du fabricant. (C) Quantification de la BFU-E à partir des populations cellulaires triées. Les populations de cellules CD9-CD150- (DN), CD9+CD150- (Prog), CD9+CD150dim (MEP) et CD9+CD150bright (MKp) ont été triées et plaquées dans du gel de méthylcellulose conformément aux instructions du fabricant. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5: Potentiel de différenciation des députés européens et des députés. Cd9-CD150-(DN), CD9+CD150-(Prog), CD9+CD150dim(MEP) et CD9+CD150bright(MKp) ont été cultivées pendant trois jours dans un milieu StemSpan complété par SCF (7,5 ng / mL), Flt-3 (5 ng / mL), IL-6 (1 ng / mL) et TPO (10 ng / mL). (A) Des images représentatives ont été prises par microscopie à contraste de phase. (B) Le pourcentage de mégacaryocytes CD41+CD42c+ a ensuite été évalué par cytométrie en flux. N = 3. (C) Le taux de ploïdie des mégacaryocytes CD41+CD42c+ a ensuite été évalué avec Hoechst par cytométrie en flux. N = 3. (D) Les cellules produites au jour 3 à partir de populations de cellules CD9-CD150-(DN), CD9+CD150-(Prog), CD9+CD150dim(MEP) et CD9+CD150bright(MKp) ont été récoltées et cultivées dans un milieu DMEM complété par 50 ng / mL TPO, 10 % de sérum de veau fœtal et 100 U / mL d’hirudine. (i) La proportion de mégacaryocytes formant des proplaquettaires dans la culture a été déterminée par observation microscopique. Les mégacaryocytes ont été identifiés en fonction de leur taille et/ou de la présence de proplaquettaires. N = 2. ii)Photographie représentative d’un mégacaryocyte porteur de proplaquettaires par microscopie à contraste de phase. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
La méthode décrite dans cet article permet l’extraction et la purification de MEP et MKp de souris. Un paramètre important dans l’optimisation du protocole était d’obtenir un nombre suffisant de cellules qui seraient compatibles avec la plupart des essais moléculaires et cellulaires. La pratique générale de la collecte d’os de souris pour l’extraction de cellules hématopoïétiques consiste généralement à prélever à la fois les fémurs et les tibias de chaque souris. L’os pelvien, autre source de matériel hématopoïétique, est donc souvent négligé. Les raisons de ne pas collecter la crête iliaque sont la mauvaise connaissance de l’anatomie interne du squelette de la souris et le fait que les utilisateurs collectent classiquement les membres postérieurs en coupant à travers ou juste au-dessus de la tête du fémur. En outre, on suppose souvent que les cellules de la moelle ne seraient pas évacuées efficacement de l’os de la crête iliaque en raison de la présence de trabécules, qui sont absentes dans la partie centrale du tibia et du fémur. Dans ce protocole, ces deux préoccupations sont abordées et une méthode normalisée, fiable et efficace en temps utile est présentée qui permet un rinçage approprié de chaque os des membres postérieurs, y compris l’os pelvien. En particulier, l’utilisation de l’os iliaque donne 105 ± 7 x 106 cellules par souris alors que la méthode classique donne généralement 42 ± 5 x 106 cellules. Un avantage majeur de cette méthode est la réduction du nombre d’animaux nécessaires pour obtenir un nombre donné de cellules cibles, offrant ainsi des conditions expérimentales plus éthiques et rentables. Cette procédure est donc également applicable à toute étude nécessitant une suspension de cellules de moelle osseuse telle que l’isolement de cellules souches hématopoïétiques10 ou l’analyse du comportement progéniteur hématopoïétique dans des conditions semi-solides11.
Le tri cellulaire par cytométrie en flux est une technique puissante avec un avantage majeur en termes de pureté par rapport aux techniques d’enrichissement magnétique, mais le rendement du tri cellulaire pour les populations rares peut être inférieur à celui des populations plus abondantes. L’épuisement magnétique des cellules indésirables au préalable est donc une méthode utile pour augmenter la fréquence des cellules d’intérêt. Ici, la procédure d’épuisement magnétique diffère de la recommandation du fabricant et prend en considération l’hétérogénéité dans l’expression des marqueurs de surface utilisés pour éliminer les cellules positives de lignée indésirables. Avec les protocoles typiques en une étape, les cellules positives de lignée avec la plus forte expression de marqueurs de surface satureront rapidement les perles magnétiques. Ils empêcheront la capture ultérieure des cellules étiquetées restantes par la concurrence et l’obstacle stérique, réduisant ainsi considérablement l’efficacité de l’épuisement. Pour résoudre ce problème, une déplétion magnétique en deux étapes a été conçue pour permettre l’élimination séquentielle de toutes les cellules positives à la lignée, permettant ainsi des épuisements rigoureux adaptés au tri cellulaire.
Un autre paramètre critique pour obtenir un épuisement efficace est les conditions d’étiquetage appropriées des cellules indésirables de lignée positive. Le titrage des anticorps a donc été spécifiquement optimisé. L’utilisation de concentrations plus élevées d’anticorps entraînera un rosetage excessif des billes magnétiques et l’épuisement non spécifique des cellules d’intérêt de lignée négative. L’utilisation de populations cellulaires MEP et MKp hautement purifiées est un outil important dans l’étude de la mégacaryopoïèse. Afin d’élucider les mécanismes contrôlant ce processus, l’étude a étudié le rôle du microenvironnement cellulaire et a montré qu’une population de cellules stromales du foie fœtal soutiendrait la différenciation de MKp10. La population triée pourrait également être utilisée pour des analyses moléculaires ou unicellulaires. Cela sera particulièrement pertinent compte tenu de la notion émergente de HSC12 , 13,14biaisé par les mégacaryocytes. La production de mégacaryocytes directement à partir de la population de CSH sans la génération d’un progéniteur bipotent serait une voie d’urgence en réponse au stress13.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Les auteurs ne déclarent aucun intérêt financier concurrent.
Acknowledgments
Les auteurs tiennent à remercier Monique Freund, Catherine Ziessel et Ketty pour leur assistance technique. Ce travail a été soutenu par l’ARMESA (Association de Recherche et Développement en Médecine et Santé Publique), et par Grant ANR-17-CE14-0001-01 à Henri.de la. Salle.
Materials
| Name | Company | Catalog Number | Comments |
| 21-gauge needles | BD Microlance | 301155 | |
| 7AAD | Sigma-Aldrich | A9400 | |
| Antibody Gr-1-biotin | eBioscience | 13-5931-85 | Magnetic depletion |
| Antibody B220-biotin | eBioscience | 13-0452-85 | Magnetic depletion |
| Antibody Mac-1-biotin | eBioscience | 13-0112-85 | Magnetic depletion |
| Antibody CD3e-biotin | eBioscience | 13-0031-85 | Magnetic depletion |
| Antibody CD4-biotin | eBioscience | 13-9766-82 | Magnetic depletion |
| Antibody CD5-biotin | eBioscience | 13-0051-85 | Magnetic depletion |
| Antibody CD8a-biotin | eBioscience | 13-0081-85 | Magnetic depletion |
| Antibody TER119-biotin | eBioscience | 13-5921-85 | Magnetic depletion |
| Antibody CD127-biotin | eBioscience | 13-1271-85 | Magnetic depletion |
| Antibody CD45-FITC | eBioscience | 11-0451-85 | Cell sorting |
| Antibody CD45-PE | eBioscience | 12-0451-83 | Cell sorting |
| Antibody TER119-APC | eBioscience | 17-5921-83 | Cell sorting |
| Antibody CD45-PECy7 | eBioscience | 25-0451-82 | Cell sorting |
| Antibody CD45-biotin | eBioscience | 13-0451-85 | Cell sorting |
| Antibody CD9-FITC | eBioscience | 11-0091-82 | Cell sorting |
| Antibody c-kit-APC | eBioscience | 17-1171-83 | Cell sorting |
| Antibody Sca-1-PE | eBioscience | 12-5981-83 | Cell sorting |
| Antibody CD16/32-PE | eBioscience | 12-0161-83 | Cell sorting |
| Antibody CD150-PECy7 | eBioscience | 25-1502-82 | Cell sorting |
| Culture medium StemSpan-SFEM | Stemcell technologies | #09650 | |
| Dissection pad | Fisher Scientific | 10452395 | |
| DPBS | Life Technologies | 14190-094 | |
| Ethanol | vWR Chemicals | 83813.360 | |
| Forceps | Euronexia | P-120-AS | |
| Glass pasteur pipette | Dutscher | 42011 | |
| Magnet : DynaMag-5 | Thermo Fisher Scientific | 12303D | |
| Magnetic beads: Dynabeads Sheep Anti-Rat IgG | Thermo Fisher Scientific | 11035 | |
| Megacult | Stemcell technologies | #04970 | |
| MethoCult SF M3436 | Stemcell technologies | #03436 | |
| Newborn Calf Serum | Dutscher | 50750-500 | |
| Red Cell Lysis solution | BD Bioscience | 555899 | |
| Scalpels | Fisher Scientific | 12308009 | |
| Scissors | Euronexia | C-165-ASB | |
| Sterile 1 mL syringes | BD Bioscience | 303172 | |
| Sterile 15mL tubes | Sarstedt | 62.554.502 | |
| Sterile 5mL polypropylene tubes | Falcon | 352063 | |
| Sterile 5mL polystyrene tubes | Falcon | 352054 | |
| Sterile tubes with 70µm cell strainer cap | Falcon | 352235 | |
| Sterile petri dish | Falcon | 353003 | |
| Streptavidin-APC-Cy7 | BD Biosciences | 554063 | Cell sorting |
| Tube roller | Benchmark Scientific | R3005 |
References
- Kaushansky, K. Historical review: megakaryopoiesis and thrombopoiesis. Blood. 111 (3), 981-986 (2008).
- Akashi, K., Traver, D., Miyamoto, T., Weissman, I. L. A clonogenic common myeloid progenitor that gives rise to all myeloid lineages. Nature. 404 (6774), 193-197 (2000).
- Debili, N., et al. Characterization of a bipotent erythro-megakaryocytic progenitor in human bone marrow. Blood. 88 (4), 1284-1296 (1996).
- Forsberg, E. C., Serwold, T., Kogan, S., Weissman, I. L., Passegué, E. New evidence supporting megakaryocyte-erythrocyte potential of flk2/flt3+ multipotent hematopoietic progenitors. Cell. 126 (2), 415-426 (2006).
- Vannucchi, A. M., et al. Identification and characterization of a bipotent (erythroid and megakaryocytic) cell precursor from the spleen of phenylhydrazine-treated mice. Blood. 95 (8), 2559-2568 (2000).
- Pronk, C. J., et al. Elucidation of the phenotypic, functional, and molecular topography of a myeloerythroid progenitor cell hierarchy. Cell Stem Cell. 1 (4), 428-442 (2007).
- Nakorn, T. N., Miyamoto, T., Weissman, I. L. Characterization of mouse clonogenic megakaryocyte progenitors. Proceedings of the National Academy of Sciences of the United States of America. 100 (1), 205-210 (2003).
- Ng, A. P., et al. Characterization of thrombopoietin (TPO)-responsive progenitor cells in adult mouse bone marrow with in vivo megakaryocyte and erythroid potential. Proceedings of the National Academy of Sciences of the United States of America. 109 (7), 2364-2369 (2012).
- Strassel, C., et al. Hirudin and heparin enable efficient megakaryocyte differentiation of mouse bone marrow progenitors. Experimental Cell Research. 318 (1), 25-32 (2012).
- Brouard, N., et al. A unique microenvironment in the developing liver supports the expansion of megakaryocyte progenitors. Blood Advances. 1 (21), 1854-1866 (2017).
- Boscher, J., Gachet, C., Lanza, F., Léon, C. Megakaryocyte culture in 3D methylcellulose-based hydrogel to improve cell maturation and study the impact of stiffness and confinement. Journal of Visualized Experiments:JOVE. , (2021).
- Sanjuan-Pla, A., et al. Platelet-biased stem cells reside at the apex of the haematopoietic stem-cell hierarchy. Nature. 502 (7470), 232-236 (2013).
- Haas, S., et al. Inflammation-driven fast-track differentiation of HSCs into the megakaryocytic lineage. Experimental Hematology. 42 (8), 14 (2014).
- Shin, J. Y., Hu, W., Naramura, M., Park, C. Y. High c-Kit expression identifies hematopoietic stem cells with impaired self-renewal and megakaryocytic bias. The Journal of Experimental Medicine. 211 (2), 217-231 (2014).
Tags
Biologie numéro 171Erratum
Formal Correction: Erratum: Isolation of Mouse Megakaryocyte Progenitors
Posted by JoVE Editors on 07/28/2021.
Citeable Link.
An erratum was issued for: Isolation of Mouse Megakaryocyte Progenitors. A figure was updated.
Figure 2 was updated from:
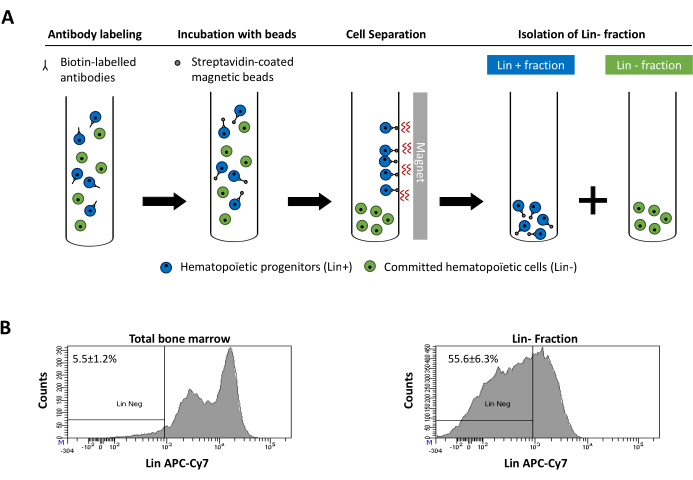
Figure 2: Magnetic depletion of lineage committed (Lin) cells. (A) Schematic representation of the magnetic depletion protocol. First, unsorted bone marrow cells are labeled with the biotin-conjugated rat anti-mouse antibody cocktail. Cells are then incubated with anti-rat Ig coated magnetic beads and subsequently subjected to the magnetic depletion using a strong magnet. The magnet will retain the labeled magnetic Lin+ fraction against the tube walls, while the unlabeled non-magnetic Lin- negative fraction will be collected in a new tube. (B) Lineage committed cells can be identified using fluorescent conjugated streptavidin. Typical analysis of the lineage expression in cells prior to magnetic depletion (total bone marrow) and after magnetic depletion (Lin- Fraction) N = 21. Please click here to view a larger version of this figure.
to:
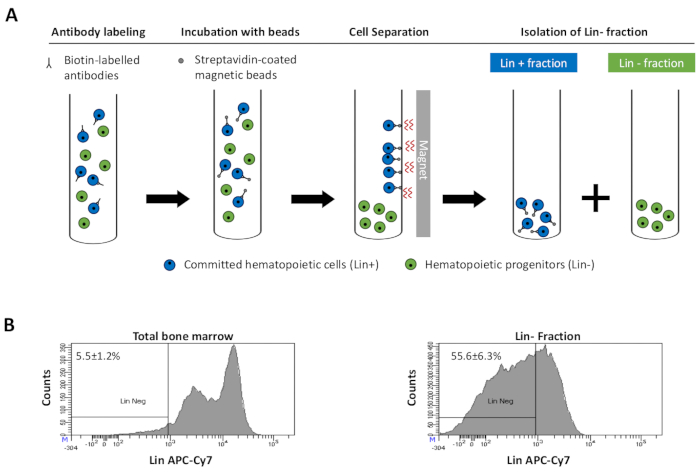
Figure 2: Magnetic depletion of lineage committed (Lin) cells. (A) Schematic representation of the magnetic depletion protocol. First, unsorted bone marrow cells are labeled with the biotin-conjugated rat anti-mouse antibody cocktail. Cells are then incubated with anti-rat Ig coated magnetic beads and subsequently subjected to the magnetic depletion using a strong magnet. The magnet will retain the labeled magnetic Lin+ fraction against the tube walls, while the unlabeled non-magnetic Lin- negative fraction will be collected in a new tube. (B) Lineage committed cells can be identified using fluorescent conjugated streptavidin. Typical analysis of the lineage expression in cells prior to magnetic depletion (total bone marrow) and after magnetic depletion (Lin- Fraction) N = 21. Please click here to view a larger version of this figure.



