商業顕微鏡でのライブ細胞膜上の分子拡散法への高速蛍光イメージングから
Summary
Spatial distribution and temporal dynamics of plasma membrane proteins and lipids is a hot topic in biology. Here this issue is addressed by a spatio-temporal image fluctuation analysis that provides conceptually the same physical quantities of single particle tracking, but it uses small molecular labels and standard microscopy setups.
Abstract
それは、空間分布および脂質やタンパク質のような膜成分の動きが多くの細胞機能の調節に重要な因子であることがますます明らかになった。ただし、高速ダイナミクスと関係する小さな構造に起因し、非常に高い時空間的分解能は、分子の実際の行動をキャッチするために必要とされている。ここでは、高時空間分解能で生きた細胞内の蛍光標識形質膜タンパク質や脂質のダイナミクスを研究するための実験プロトコルを提示する。注目すべきことに、このアプローチは、各分子を追跡する必要がないが、それは、膜の所定の領域内のすべての分子を用いて母集団の挙動を計算する。出発点は、膜上の所定の領域の高速撮像である。その後、完全な空間時間的自己相関関数は、例えば、それぞれ2個、3個、nは繰り返しの時間遅延を増加させるで取得された画像を相関計算される。それは、幅があることを証明することができます空間的自己相関関数のピークの拡散による粒子移動の関数として増加する時間遅延で増加する。そのため、自己相関関数の一連のフィッティング(iMSD)を画像化するから、正方形の変位を意味し、実際のタンパク質を抽出することができ、ここでは平均変位対見掛け拡散係数の形で提示。これは、ナノメートルの精度で単一分子の平均ダイナミクスの定量的な見解が得られます。標識されたトランスフェリン受容体(TfRは)及びATTO488のGFPタグ付き変異体を用いて、1 -パルミトイル-2 -ヒドロキシ- のsnグリセロ-3 -ホスホエタノールアミン(PPE)は、上のタンパク質および脂質拡散の時空間レギュレーションを観察することが可能であるマイクロ対ミリ秒の時間範囲の程度の大きさの膜領域を含む。
Introduction
シンガーとニコルソンによるオリジナル「流体モザイク」モデルから始めて、携帯形質膜の画像を連続的に細胞骨格と脂質ドメイン1,2の新たな役割を含めるために、過去数十年の間、更新されています。
最初の観察は、膜タンパク質のかなりの割合が不動3-5であることを発表(FRAP)の光退色後蛍光回復により得た。これらの先駆的な研究は、非常に有益なものの、FRAPのセットアップの比較的乏しい空間での分解能(ミクロン)と時間(秒)に苦しんだ。また、アンサンブル平均測定値である、FRAP、単一分子の挙動についての情報を与えることに欠けている。
この文脈において、特に非常に明るいタグ(一度に拡散工程つの分子の研究を可能にする)を有する単一の分子を標識する可能性は非常に成功している。特に、押してマイクロ秒の時間スケールへの単一粒子追跡(SPT)アプローチの時間分解能、楠見ら大幅に膜生理学6におけるアクチンベースの膜骨格の役割の認識に貢献した脂質とタンパク質ダイナミクスの未知の機能へのアクセスを獲得7。これらの知見は、脂質およびタンパク質の拡散がアクチン系骨格によって調節される、いわゆる「ピケットフェンス」モデルが、生成された。しかし、SPT多くの実験の問題によって提供される情報の膨大な量にアクセスするためにアドレス指定されなければならない。特に、標識操作は、一般的に、生産、精製、およびシステムへの標識種の導入のような多くのステップによって構成されています。さらに、大きなラベルは、量子ドット、または金属ナノ粒子のような、多くの場合、ミリ秒以下の時間スケールに達するために必要とされ、ラベルによる標的分子の架橋は、多くの場合において回避することができなかった。最後に、多くの軌跡統計的基準に適合するために記録されなければならないし、同時にラベルの低密度は、追跡を可能にするために必要とされている。
SPT、蛍光相関分光法(FCS)と比較して、これらの欠点の多くを克服し、分子動力学を研究するための非常に有望なアプローチを表す。実際には、FCSが一過性にトランスフェクションされた細胞に蛍光タンパク質タグ化分子のダイナミクスを研究するために有効に、薄暗い、緻密なラベルでもうまく動作します。また、限られた時間の中で高い統計を達することができます。最後に、ラベルの「高」密度にもかかわらず、FCSが単一分子情報を提供します。おかげで、これらすべての特性のために、FCSは非常に簡単なアプローチを表し、広範囲にモデル膜において、生細胞8-10に両方の脂質およびタンパク質のダイナミクスを研究するために適用されている。多くの異なるアプローチは、分子拡散の詳細を明らかにするためにFCSの能力を増加させるために提案されている。例えば、それはshにあっ異なるサイズの観察領域にFCSを実行することによって、一つは分子運動11,12の「FCS拡散法」啓発隠された機能を定義できることを自分自身。サイズが多様であることに加え、焦点領域も13重複した行14-20または高速カメラ21,22共役一緒に空間内を移動。これらの「時空間」相関アプローチを用いて、いくつかの膜成分の関連する生物学的パラメータを定量的にこのようにして、膜の空間的組織への洞察をもたらす、モデル膜と実際の生物学的なものの両方に記載されている。
しかし、すべてのFRAPとFCS用途でこれまでに説明した焦点領域の大きさを克服することができない空間分解能の限界を表している。いくつかの超解像画像化方法は、最近、この制限を回避するために開発されている。いくつかは、そのような確率論的光学再構築顕微鏡(STORM)として、ローカライズの精度に基づいています<SUP> 23,24、光活性化局在化顕微鏡(PALM)25、蛍光PALM(FPALM)26、及び単一粒子追跡PALM(sptPALM)27:各スナップショットに必要な光子の比較的大きな量が、しかしながら、時間分解能を制限する少なくとも数ミリ秒、これらの方法は、従って、インビボでのそれらの適用を妨げる。
対照的に、超解像イメージングのための有望な代替手段は、空間的に誘導放出の枯渇法(STEDまたは可逆的飽和性光学蛍光遷移(RESOLFT))28,29で蛍光発光を変調することによって開かれている。これらのアプローチは、高速走査型顕微鏡および検出システムを使用する可能性を有するだけでなく、回折限界以下の観察体積の整形を組み合わせる。蛍光変動解析と組み合わせて、STED顕微鏡を直接脂質およびpのナノスケール時空間的動態を探査することができ生細胞膜30,31中roteins。
STED顕微鏡ベースの同一の物理量は、生細胞中の蛍光標識膜タンパク質及び/又は脂質のダイナミクスを研究するのに適している改変された時空間画像相関分光法(STICS 32,33)の方法によって得ることができるや商業顕微鏡による。ここで紹介する実験プロトコルは、いくつかの手順によって構成されています。最初のものは膜上の関心領域の高速撮像を必要とします。そして、画像の得られる積層体は、平均空間 – 時間相関関数を計算するために使用される。 -平均変位プロット対 -相関関数のシリーズを嵌合することにより、分子の「拡散法は、「見かけの拡散係数(D アプリ )の形態の撮像から直接得ることができる。このプロットは、批判的に分子によって探求、環境に依存しており、直接、実際の拡散モードを認識することができます興味のある脂質/タンパク質の。
より詳細には、先に示した34ように、取得した画像シリーズの時空間的自己相関関数は、批判的に収集された画像シリーズ内を移動する分子のダイナミクスに依存する(同じ論法がラインの取得に適用することができることに注意してください空間内のただ一つの次元が考慮されている)。特に、本発明者らは、のように、相関関数を定義する。
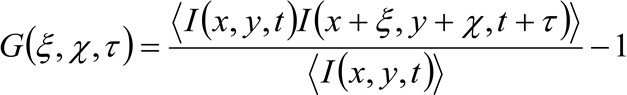 (1)
(1)
どこ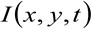 位置xで測定された蛍光強度を表し、yおよび時間tにおける、
位置xで測定された蛍光強度を表し、yおよび時間tにおける、 ![]() そして
そして![]() x方向の距離を表し、それぞれy方向、
x方向の距離を表し、それぞれy方向、 ![]() タイムラグを表し、そして
タイムラグを表し、そして![]() 平均を表す。この関数は、のように表すことができる。
平均を表す。この関数は、のように表すことができる。
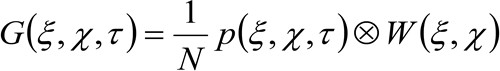 (2)
(2)
'N'は、観察領域内の分子の平均数を表す![]() 空間における畳み込み演算を表し、そして
空間における畳み込み演算を表し、そして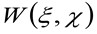 楽器の腰の自己相関を表しています。この後者は、光学/録画設定に単一の発光体の光子が空間に広がっているかの尺度として解釈することができる(いわゆる点広がり関数、PSF、遺伝子よくガウス関数で近似ラリー)。最後に、
楽器の腰の自己相関を表しています。この後者は、光学/録画設定に単一の発光体の光子が空間に広がっているかの尺度として解釈することができる(いわゆる点広がり関数、PSF、遺伝子よくガウス関数で近似ラリー)。最後に、 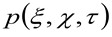 距離で粒子を見つけるために確率を表す
距離で粒子を見つけるために確率を表す![]() そして
そして![]() 時間遅延の後に
時間遅延の後に![]() 。私たちは、粒子が全ての方向にランダムに移動し、正味フラックスは存在せず、この関数はまた、よく分散が移動する粒子の平均二乗変位(MSD)として識別することができ、ガウス関数で近似される拡散動力学を考慮した場合。従って、相関関数(の腰とも呼ば
。私たちは、粒子が全ての方向にランダムに移動し、正味フラックスは存在せず、この関数はまた、よく分散が移動する粒子の平均二乗変位(MSD)として識別することができ、ガウス関数で近似される拡散動力学を考慮した場合。従って、相関関数(の腰とも呼ば![]() )、粒子のMSDの和楽器の腰のように定義することができ、ガウスフィットによって測定することができる各時間遅延のための相関関数のティン。測定された私 MSD移動分子の見かけの拡散係数を計算するために使用することができ
)、粒子のMSDの和楽器の腰のように定義することができ、ガウスフィットによって測定することができる各時間遅延のための相関関数のティン。測定された私 MSD移動分子の見かけの拡散係数を計算するために使用することができ![]() と平均変位
と平均変位![]() として:
として:
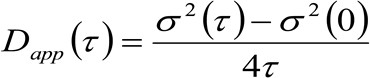 (3)
(3)
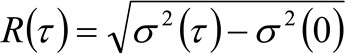 (4)
(4)
使用された実験のセットアップには、いくつかの考慮事項は、次のセクションを通して読者を導くことができる。選択的に生細胞の基底膜上のフルオロフォアを励起するために、私たちは、商業のTIR蛍光(TIRF)顕微鏡(詳細は材料セクションを参照のこと)を使用して、全反射(TIR)照明を使用します。また、順番に目を収集する電子蛍光当社は高倍率の対物レンズ(100X NA 1.47、高開口数を全反射照明に必要とされる)と、EMCCDカメラ(内蔵さ16μmの画素の物理サイズ)を使用します。 100nmのピクセルサイズに到達するために、私たちは、1.6Xのさらなる拡大レンズを適用する。以下に説明するように、1ミリ秒以下の時間分解能を適切に100nm未満の速い膜脂質の動力学を記述するために必要とされるであろう。この時間分解能を達成するために、私たちは、カメラ(512×512)のチップ全体よりも小さい関心領域(ROI)を選択する必要があります。このように、カメラは、時間分解能を増加線の数の減少を読み取る。しかし、この読み出し領域でフレーム時間は、カメラの読み出しチップへの暴露からの電荷をシフトするために必要な時間によって制限されると、512×512ピクセルEMCCDミリ秒のオーダーで通常である。この制限を破ったために、新しい技術は、wは、ROIラインのみの代わりに、フレーム全体をシフトできます(私たちのEMCCDにクロップドセンサーモードと呼ばれる)が露出し、チップサイズの実用的な効果的な減少番目。この設定を有効にするには、ROIの外側のチップは、光路に取り付けられたスリットのカップルによってカバーされなければならない。時間分解能ダウン10 -4秒にこのセットアップのおかげで達成することができる。 「議論」のセクションで説明したように、このアプローチは、多くの異なった実験装置と結合することができること、しかし、注意してください。
この方法の実証は両方ATTO488を使用することにより、生きた細胞内で提供される1 -パルミトイル-2-ヒドロキシのsnグリセロ-3 -ホスホエタノールアミン(ATTO488-PPE)及びトランスフェリン受容体のGFP標識された変異体(GFP-標識されたのTfR)。以前に30,35を報告したようにATTO488-PPEの場合、このアプローチは成功し、大部分は自由拡散を示す平均変位の関数としてほぼ一定のD アプリを回復することができます。対照的に、TfRは-GFPは減少Dを示している<su部分的に閉じ込められた拡散6を示唆して平均変位の関数としてb>のアプリ。また、後者の場合には、ローカル拡散定数と膜面上の多数のミクロンを超える平均閉じ込め領域を定量化することが可能である。
Protocol
Representative Results
Discussion
単一粒子追跡(SPT)は、分子動力学を研究するための最も一般的な戦略の1つを表し、それは粒子軌道を測定するという大きな利点を有する。これは、複雑なシステムでさえ、いくつかの標識された粒子の挙動をプロービングを可能にする。しかし、SPTは、典型的には、プローブと、非常に明るいラベルの低密度を必要とするこの利点に到達する。特に、高い時間分解能(マイクロ秒の範囲)…
Disclosures
The authors have nothing to disclose.
Acknowledgements
This work is supported in part by NIH-P41 P41-RRO3155 and NIH P50-GM076516 (grant to EG), and Fondazione Monte dei Paschi di Siena (grant to FB).
Materials
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| iXon Ultra 897 | Andor | DU-897U-CS0 | |
| Solis | Andor | ||
| CHO-K1 | ATCC | CCL-61 | |
| ATTO 488 labeled PPE | ATTO-TEC GmbH | AD 488-151 | |
| DOPE | Avanti Polar Lipids, Inc. | 850725 | |
| DOTAP | Avanti Polar Lipids, Inc. | 890890 | |
| 100X Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | |
| DMEM/F-12 | Gibco | 21331 | |
| FBS | Gibco | 10082147 | |
| HEPES | Gibco | 15630-106 | |
| PBS | Gibco | 10010-023 | |
| SimFCS 3.0 | Globals Software | the software can be downloaded here: http://www.lfd.uci.edu/globals/ | |
| DMI6000 with TIRF modulus | Leica | ||
| LAS AF | Leica | ||
| Lipofectamine 2000 | Lipofectamine | 11668019 | |
| Matlab | MathWork | ||
| Imagej | NIH | ||
| C-terminal GFP tagged Tranferrin Receptor | OriGene | RG200980 | |
| Agar | Sigma Aldrich | A5306 | |
| Chloroform | Sigma Aldrich | 528730 | |
| Latex beads, fluorescent yellow-green, 30 nm | Sigma Aldrich | L5155 | |
| SONICA Ultrasonic Cleaners | SOLTEC | ETH S3 | |
| Petri Dishes | Willco | GWSt-3522 | |
| Bio-Format importer for Matlab | http://www.openmicroscopy.org/site/support/bio-formats5/users/matlab/ | ||
| ICS-MatLab Tools | https://www.cellmigration.org/resource/imaging/software/ICSMATLAB_28-02-06.zip | ||
| Simulation by Matlab Tutorial | https://www.cellmigration.org/resource/imaging/icsmatlab/ICSTutorial.html | ||
| Simulation by SimFCS Tutorial | https://www.cellmigration.org/resource/imaging/ppt-pdf/RICS%20Simulations.ppt |
References
- Engelman, D. M. Membranes are more mosaic than fluid. Nature. 438 (7068), 578-580 (2005).
- Vereb, G., et al. yet structured: The cell membrane three decades after the Singer-Nicolson model. Proc. Natl. Acad. Sci. U. S. A. 100 (14), 8053-8058 (1073).
- Ishihara, A., Hou, Y., Jacobson, K. The Thy-1 antigen exhibits rapid lateral diffusion in the plasma membrane of rodent lymphoid cells and fibroblasts. 84 (5), 1290-1293 (1987).
- Axelrod, D., et al. Lateral motion of fluorescently labeled acetylcholine receptors in membranes of developing muscle fibers. Proc. Natl. Acad. Sci. U. S. A. 73 (12), 4594-4598 (1976).
- Jacobson, K., Derzko, Z., Wu, E. S., Hou, Y., Poste, G. Measurement of the lateral mobility of cell surface components in single, living cells by fluorescence recovery after photobleaching. J. Supramol. Struct. 5 (4), 10-1002 (1976).
- Kusumi, A., et al. Paradigm shift of the plasma membrane concept from the two-dimensional continuum fluid to the partitioned fluid: high-speed single-molecule tracking of membrane molecules. Annu. Rev. Biophys. Biomol. Struct. 34, 351-378 (2005).
- Kusumi, A., Ike, H., Nakada, C., Murase, K., Fujiwara, T. Single-molecule tracking of membrane molecules: plasma membrane compartmentalization and dynamic assembly of raft-philic signaling molecules. Semin. Immunol. 17 (1), 3-21 (2005).
- Schwille, P., Korlach, J., Webb, W. W. Fluorescence correlation spectroscopy with single-molecule sensitivity on cell and model membranes. Cytometry. 36, 176-182 (1999).
- Gielen, E., et al. Diffusion of sphingomyelin and myelin oligodendrocyte glycoprotein in the membrane of OLN-93 oligodendroglial cells studied by fluorescence correlation spectroscopy. C. R. Biol. 328 (12), 1057-1064 (2005).
- Weiss, M., Hashimoto, H., Nilsson, T. Anomalous protein diffusion in living cells as seen by fluorescence correlation spectroscopy. Biophys. J. 84, 4043-4052 (2003).
- Wawrezinieck, L., Rigneault, H., Marguet, D., Lenne, P. F. Fluorescence correlation spectroscopy diffusion laws to probe the submicron cell membrane organization. Biophys. J. 89 (6), 4029-4042 (2005).
- Lenne, P. F., et al. Dynamic molecular confinement in the plasma membrane by microdomains and the cytoskeleton meshwork. EMBO J. 25 (14), 3245-3256 (2006).
- Ries, J., Schwille, P. Studying slow membrane dynamics with continuous wave scanning fluorescence correlation spectroscopy. Biophys. J. 91 (5), 1915-1924 (2006).
- Ruan, Q., Cheng, M. A., Levi, M., Gratton, E., Mantulin, W. W. Spatial-temporal studies of membrane dynamics: scanning fluorescence correlation spectroscopy (SFCS). Biophys. J. 87 (2), 1260-1267 (2004).
- Berland, K. M., So, P. T., Chen, Y., Mantulin, W. W., Gratton, E. Scanning two-photon fluctuation correlation spectroscopy: particle counting measurements for detection of molecular aggregation. Biophys. J. 71, 410-420 (1996).
- Heinemann, F., Betaneli, V., Thomas, F. A., Schwille, P. Quantifying lipid diffusion by fluorescence correlation spectroscopy: a critical treatise. Langmuir. 28 (37), 13395-13404 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Capturing directed molecular motion in the nuclear pore complex of live cells. Proc. Natl. Acad. Sci. U. S. A. 109 (25), 9863-9868 (2012).
- Sanchez, S. A., Tricerri, M. A., Gratton, E. Laurdan generalized polarization fluctuations measures membrane packing micro-heterogeneity in vivo. Proc. Natl. Acad. Sci. U. S. A. 109 (19), 7314-7319 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Fluorescence correlation spectroscopy of intact nuclear pore complexes. Biophys. J. 101 (4), 27-29 (2012).
- Di Rienzo, C., et al. Unveiling LOX-1 receptor interplay with nanotopography: mechanotransduction and atherosclerosis onset. Sci. Rep. 3, 10-1038 (2013).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophys. J. 95 (11), 5385-5398 (2008).
- Kannan, B., et al. Electron multiplying charge-coupled device camera based fluorescence correlation spectroscopy. Anal. Chem. 78 (10), 3444-3451 (2006).
- Jones, S. A., Shim, S. H., He, J., Fast Zhuang, X. three-dimensional super-resolution imaging of live cells. Nat. Methods. 8 (6), 499-508 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy. 3 (10), 793-795 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91 (11), 4258-4272 (2006).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Klar, T. A., Hell, S. W. Subdiffraction resolution in far-field fluorescence microscopy. Opt. Lett. 24 (14), 954-956 (1999).
- Eggeling, C., et al. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457 (7233), 1159-1162 (2009).
- Hedde, P. N., et al. Stimulated emission depletion-based raster image correlation spectroscopy reveals biomolecular dynamics in live cells. Nat. Commun. 4, .
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88 (5), 3601-3614 (2005).
- Brown, C. M., et al. Probing the integrin-actin linkage using high-resolution protein velocity mapping. J. Cell Sci. 119, 5204-5214 (2006).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proc. Natl. Acad. Sci. U. S. A. 110 (30), 12307-12312 (2013).
- Mueller, V., et al. STED nanoscopy reveals molecular details of cholesterol- and cytoskeleton-modulated lipid interactions in living cells. Biophys. J. 101 (7), 1651-1660 (2011).
- Kleusch, C., Hersch, N., Hoffmann, B., Merkel, R., Csiszar, A. Fluorescent lipids: functional parts of fusogenic liposomes and tools for cell membrane labeling and visualization. Molecules. 17 (1), 1055-1073 (2012).
- Ries, J., Chiantia, S., Schwille, P. Accurate determination of membrane dynamics with line-scan FCS. Biophys. J. 96 (5), 1999-2008 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49 (3), 141-164 (2007).
- Digman, M. A., et al. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89 (2), 1317-1327 (2005).
- Ritchie, K., et al. Detection of non-Brownian diffusion in the cell membrane in single molecule tracking. Biophys. J. 88 (3), 2266-2277 (2005).
- Voie, A. H., Burns, D. H., Spelman, F. A. Orthogonal-plane fluorescence optical sectioning: three-dimensional imaging of macroscopic biological specimens. J. Microsc. 170, 229-236 (1993).
- Huisken, J., Swoger, J., Del Bene, ., Wittbrodt, F., J, E. H., Stelzer, Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Wohland, T., Shi, X., Sankaran, J., Stelzer, E. H. Single plane illumination fluorescence correlation spectroscopy (SPIM-FCS) probes inhomogeneous three-dimensional environments. Opt. Express. 18 (10), 10627-10641 (2010).

