Edição do genoma em linhas de células de mamíferos usando CRISPR-Cas
Summary
CRISPR-Cas é uma tecnologia poderosa para projetar os genomas complexos de plantas e animais. Aqui, detalhamos um protocolo eficiente editar o genoma humano usando diferentes endonucleases de Cas. Destacamos importantes considerações e parâmetros de projeto para otimizar a eficiência de edição.
Abstract
O sistema regularmente intercaladas curta palíndromo repetições (CRISPR) em cluster funciona naturalmente na imunidade adaptativa bacteriana, mas tem com êxito foi realocado para engenharia do genoma em muitos organismos vivos diferentes. Mais comumente, a sua CRISPR associado 9 (Cas9) ou Cas12a endonuclease é usado para decompor a locais específicos no genoma, após o qual o intervalo de dupla-hélice do DNA é reparado através de fim não-homóloga aderir a caminho (NHEJ) ou o reparo de homologia-dirigido ( Caminho HDR) dependendo se é um modelo de doador ausentes ou apresentam respectivamente. Até à data, sistemas CRISPR de diferentes espécies bacterianas foram mostrados para ser capaz de realizar a edição de genoma em células de mamíferos. No entanto, apesar da aparente simplicidade da tecnologia, vários parâmetros de projeto precisam ser considerados, que muitas vezes deixam perplexos sobre como melhor realizar seu genoma edição de experiências de usuários. Aqui, descrevemos um fluxo de trabalho completo de delineamento experimental para identificação de clones de células que levam a modificações de DNA desejadas, com o objetivo de facilitar a execução bem-sucedida do genoma edição experimentos em linhas de células de mamíferos. Destacamos as considerações-chave para que os usuários anotem, incluindo a escolha do sistema CRISPR, o comprimento do espaçador e o design de um modelo de doador single-stranded oligodeoxynucleotide (ssODN). Vislumbramos que este fluxo de trabalho será útil para estudos de gene nocaute, doença modelagem esforços, ou linhas de célula a geração de repórter.
Introduction
A capacidade para projetar o genoma de qualquer organismo vivo tem muitas aplicações biomédicas e biotecnológicas, como a correção da doença-causando mutações, construção de modelos de celulares precisos para estudos de doença, ou geração de produtos agrícolas culturas com características desejáveis. Desde a virada do século, diversas tecnologias foram desenvolvidas para a engenharia do genoma em células de mamíferos, incluindo meganucleases1,2,3, zinco dedo nucleases4,5, ou transcrição como ativador effector nucleases (TALENs)6,7,8,9. No entanto, estas tecnologias anteriores são difíceis de programa ou tedioso para montar, prejudicando, assim, sua adoção generalizada na investigação e a indústria.
Nos últimos anos, o cluster regularmente intercaladas curtas palíndromos repetições (CRISPR) – sistema CRISPR-associado (Cas) tem emergido como um poderoso novo genoma engenharia tecnologia10,11. Originalmente um sistema imune adaptativo em bactérias, tem sido com sucesso implantado para a modificação do genoma em plantas e animais, incluindo seres humanos. Uma razão principal por que CRISPR-Cas ganhou tanta popularidade em tão pouco tempo é que o elemento que traz a chave endonuclease de Cas, tais como Cas9 ou Cas12a (também conhecido como Cpf1), para o local correto no genoma é simplesmente um pedaço curto de quimérico único guia RN Um (sgRNA), que é simples para o projeto e barato para sintetizar. Depois de ser recrutado para o site de destino, a enzima Cas funciona como um par de tesouras moleculares e cliva o DNA acoplado com sua RuvC, HNH ou Nuc domínios12,13,14. A resultante quebra encalhada dobro (DSB) é posteriormente reparada pelas células através de fim não-homóloga (NHEJ) juntar-se ou no caminho de reparação homologia-dirigido (HDR). Na ausência de um modelo de reparação, o ORL é reparado por via NHEJ propenso a erro, que pode dar origem a pseudo-aleatório inserção ou supressão de nucleotídeos (puntuais) no local do corte, potencialmente causando frameshift mutações nos genes codificantes de proteínas. No entanto, na presença de um modelo de doador que contém as alterações de DNA desejadas, o ORL é reparado por via HDR de alta fidelidade. Tipos comuns de modelos de doadores incluem oligonucleotides single-stranded (ssODNs) e plasmídeos. O primeiro costuma ser usado se as alterações de DNA pretendidas são pequenas (por exemplo, a alteração de um único par de base), enquanto o último é geralmente usado se deseja inserir uma sequência relativamente longa (por exemplo, a sequência de codificação de uma proteína verde fluorescente ou GFP) para o local de destino.
A atividade de endonuclease da proteína Cas requer a presença de um motivo protospacer adjacentes (PAM) para o local de destino15. O PAM de Cas9 é na extremidade 3′ da protospacer, enquanto o PAM de Cas12a (também chamado Cpf1) é, ao invés, na extremidade 5′16. O Cas-guia complexo RNA é incapaz de apresentar um ORL se o PAM estiver ausente17. Daí, o PAM coloca uma restrição na genômicas locais onde um determinado nuclease Cas é capaz de decompor. Felizmente, nucleases de Cas de diferentes espécies bacterianas geralmente apresentam exigências diferentes do PAM. Portanto, integrando vários sistemas CRISPR-Cas em nossa caixa de ferramentas de engenharia, podemos expandir a gama de sites que podem ser alvo de um genoma. Além disso, uma enzima natural do Cas pode ser projetada ou evoluiu para reconhecer sequências de PAM alternativas, ainda mais, alargar o âmbito da genômicos alvos acessíveis para manipulação de19,18,20.
Embora vários sistemas CRISPR-Cas estão disponíveis para fins de engenharia do genoma, a maioria dos usuários da tecnologia dependem principalmente da nuclease Cas9 de Streptococcus pyogenes (SpCas9) por várias razões. Em primeiro lugar, requer um PAM relativamente simplesmente NGG, ao contrário de muitas outras proteínas de Cas que podem cleave apenas na presença de PAMs mais complexas. Em segundo lugar, é a primeira endonuclease Cas seja implantado com sucesso em células humanas21,22,23,24. Em terceiro lugar, SpCas9 é, de longe, a melhor enzima caracterizada até à data. Se um pesquisador deseja usar outro nuclease Cas, ele ou ela muitas vezes seria incerto sobre qual a melhor forma de projetar o experimento e bem como outras enzimas irão realizar em diferentes contextos biológicos em relação ao SpCas9.
Para fornecer clareza para o desempenho relativo dos diferentes sistemas de CRISPR-Cas, recentemente Efetuamos uma comparação sistemática de cinco endonucleases de Cas-SpCas9, a enzima Cas9 de Staphylococcus aureus (SaCas9), a enzima Cas9 de Neisseria meningitidis (NmCas9), a enzima Cas12a de SP Acidaminococcus BV3L6 (AsCas12a) e a enzima Cas12a de bactéria Lachnospiraceae ND2006 (LbCas12a)25. Para uma comparação justa, avaliamos as nucleases de Cas diferentes usando o mesmo conjunto de sites de destino e outras condições experimentais. Os parâmetros de projeto de estudo também delineado para cada sistema CRISPR-Cas, que serviu como uma referência útil para os usuários da tecnologia. Aqui, para melhor permitir aos investigadores fazer uso das CRISPR-Cas sistema, nós fornecemos um protocolo passo a passo para engenharia de genoma ideal com diferentes enzimas Cas9 e Cas12a (ver Figura 1). O protocolo inclui não apenas detalhes experimentais, mas considerações de design também é importante para maximizar a probabilidade de um resultado de sucesso do genoma engenharia em células de mamíferos.
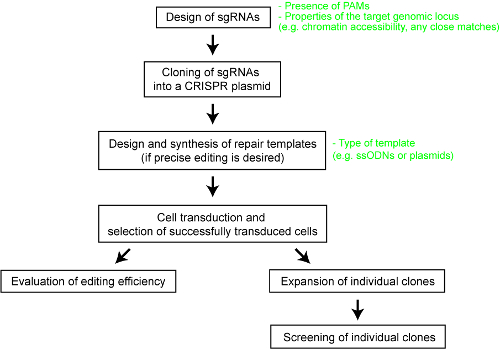
Figura 1 : Uma visão geral do fluxo de trabalho para gerar o genoma edição linha celular humana. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
O sistema CRISPR-Cas é uma poderosa, revolucionária tecnologia para projetar os genomas e transcriptomes de plantas e animais. Numerosas espécies bacterianas foram encontradas para conter sistemas CRISPR-Cas, que potencialmente podem ser adaptados para o genoma e transcriptoma fins44de engenharia. Embora a endonuclease Cas9 de Streptococcus pyogenes (SpCas9) foi a primeira enzima a ser implantado com sucesso em células humanas21,22<…
Disclosures
The authors have nothing to disclose.
Acknowledgements
M.H.T. é suportado por uma agência de tecnologia da ciência e da pesquisa concessão de escritório comum do Conselho (1431AFG103), um Conselho Nacional de pesquisa médica grant (OFIRG/0017/2016), Fundação Nacional de pesquisa concede (NRF2013-THE001-046 e NRF2013-THE001-093), um Concessão do Ministério da educação Tier 1 (RG50/17 (S)), uma startup grant da Universidade Tecnológica de Nanyang e fundos para a competição internacional geneticamente engenharia máquina (Medren) da Universidade Tecnológica de Nanyang.
Materials
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | – | – | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
References
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. 遗传学. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).

