نمذجة الغشاء الواقعية باستخدام مخاليط الدهون المعقدة في دراسات المحاكاة
Summary
يعد تنوع الدهون الغشائية في التركيب والتكوين مساهما مهما في العمليات الخلوية ويمكن أن يكون علامة على المرض. تسمح لنا محاكاة الديناميات الجزيئية بدراسة الأغشية وتفاعلاتها مع الجزيئات الحيوية بدقة ذرية. هنا ، نقدم بروتوكولا لبناء وتشغيل وتحليل أنظمة الأغشية المعقدة.
Abstract
الدهون هي اللبنات الهيكلية لأغشية الخلايا. تختلف أنواع الليبيدات عبر عضيات الخلايا وعبر الكائنات الحية. ينتج عن هذا التنوع خصائص ميكانيكية وهيكلية مختلفة في الغشاء تؤثر بشكل مباشر على الجزيئات والعمليات التي تحدث في هذه الواجهة. تكوين الدهون ديناميكي ويمكن أن يعمل على تعديل عمليات إشارات الخلية. تستخدم الأساليب الحسابية بشكل متزايد للتنبؤ بالتفاعلات بين الجزيئات الحيوية وتوفير رؤى جزيئية للملاحظات التجريبية. الديناميات الجزيئية (MD) هي تقنية تعتمد على الميكانيكا الإحصائية التي تتنبأ بحركة الذرات بناء على القوى التي تعمل عليها. يمكن استخدام محاكاة MD لتوصيف تفاعل الجزيئات الحيوية. هنا ، نقدم بإيجاز التقنية ، ونحدد الخطوات العملية للمبتدئين المهتمين بمحاكاة طبقات الدهون المزدوجة ، ونوضح البروتوكول باستخدام برامج صديقة للمبتدئين ، ونناقش البدائل والتحديات والاعتبارات المهمة للعملية. على وجه الخصوص ، نؤكد على أهمية استخدام مخاليط الدهون المعقدة لنمذجة غشاء الخلية المهم لالتقاط البيئات الميكانيكية والكارهة للماء المناسبة في المحاكاة. نناقش أيضا بعض الأمثلة حيث يعدل تكوين الغشاء وخصائصه تفاعلات الطبقات الثنائية مع الجزيئات الحيوية الأخرى.
Introduction
الدهون هي المكونات الرئيسية للأغشية ، والتي توفر حدودا للخلايا وتمكن من التقسيم داخل الخلايا1،2،3. الدهون هي البرمائيات ، مع مجموعة رأس قطبية واثنين من ذيول الأحماض الدهنية الكارهة للماء. تتجمع هذه ذاتيا في طبقة ثنائية لتقليل ملامسة السلاسل الكارهة للماءبالماء 3,4. ينتج عن مجموعات مختلفة من مجموعات الرأس المحبة للماء والذيول الكارهة للماء فئات مختلفة من الليبيدات في الأغشية البيولوجية ، مثل الجليسيروفوسفوليبيدات ، والشحميات السفينغوبية ، والستيرول (الشكل 1) 1،5،6. الغليسيروفوسفوليبيدات هي اللبنات الأساسية لأغشية الخلايا حقيقية النواة المكونة من الجليسيروفوسفات، والأحماض الدهنية طويلة السلسلة، ومجموعات الرأس ذات الوزن الجزيئيالمنخفض 7. تعتمد تسميات الدهون على الاختلافات في مجموعات الرأس. ومن الأمثلة على ذلك فوسفاتيديل كولين (PC) ، فوسفاتيديل إيثانولامين (PE) ، فوسفاتيديل سيرين (PS) ، فوسفاتيديل جليسرول (PG) ، فوسفاتيديل إينوسيتول (PI) ، أو حمض الفوسفاتيديك غير المعدل (PA) 5,6. أما بالنسبة للذيول الكارهة للماء ، فإن طول ودرجة التشبع تختلف ، جنبا إلى جنب مع بنية العمود الفقري. التوليفات المحتملة عديدة ، مما يؤدي إلى الآلاف من أنواع الدهون في خلايا الثدييات6. تؤدي التغييرات في تكوين الدهون الغشائية إلى خصائص غشاء ميكانيكية وهيكلية مختلفة تؤثر على نشاط كل من بروتينات الغشاء المتكامل والبروتينات المحيطية 2,6.
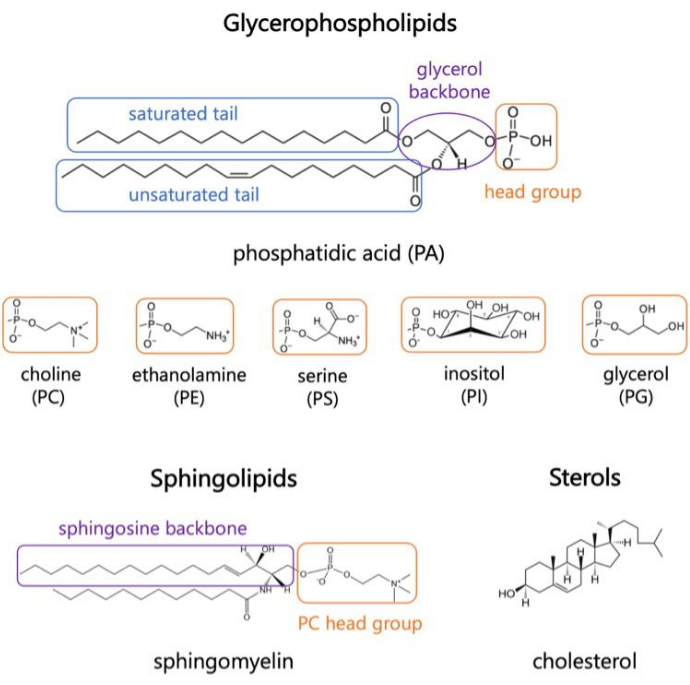
الشكل 1. هياكل الدهون التمثيلية. تظهر ذيول الأحماض الدهنية في مربعات زرقاء، ومجموعات رؤوس الليبيدات الشائعة باللون البرتقالي، والعمود الفقري للعينة باللون الأرجواني. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
الدهون هي لاعبين نشطين في العمليات الخلوية ، وتنشيط البروتين في شلالات الإشارات ، وتوازن الخلايا السليمة 8,9. ديناميات الدهون المتغيرة هي نتيجة للعدوى أو يمكن أن تكون علامات على التسبب في المرض10،11،12،13،14،15. كحواجز للخلية ، فإن دراسة الدهون الغشائية ودورها في تغلغل الجزيئات الصغيرة ذات صلة بأنظمة توصيل الأدوية وآليات تعطيل الغشاء16,17. يؤدي التنوع الكيميائي والنسب المختلفة لأنواع الدهون عبر العضيات والأنسجة والكائنات الحية إلى ديناميكيات غشاء معقدة2. لذلك من المهم الاحتفاظ بهذه الخصائص في دراسات النمذجة للطبقات المزدوجة الدهنية ، خاصة عندما يكون الهدف من الدراسة هو فحص تفاعلات الجزيئات الحيوية الأخرى مع الغشاء. تعتمد الأنواع الدهنية التي يجب مراعاتها في النموذج على الكائن الحي والمقصورة الخلوية محل الاهتمام. على سبيل المثال ، تعتبر دهون PG مهمة لنقل الإلكترون في bateria18 الضوئي ، في حين أن دهون الإينوزيتول المفسفرة (PIPs) هي لاعبين رئيسيين في ديناميكيات غشاء البلازما (PM) وشلالات الإشارات في خلايا الثدييات 19,20. داخل الخلية ، تحتوي أغشية PM والشبكة الإندوبلازمية (ER) و Golgi وأغشية الميتوكوندريا على وفرة فريدة من الدهون تؤثر على وظيفتها. على سبيل المثال ، ER هو محور التكوين الحيوي للدهون وينقل الكوليسترول إلى PM و Golgi. يحتوي على تنوع عالي الدهون مع وفرة من PC و PE ، ولكن محتوى ستيرول منخفض ، مما يعزز سيولة الغشاء21،22،23،24. في المقابل ، يشتمل PM على مئات وحتى آلاف الأنواع الدهنية اعتمادا على الكائن الحي25 ، ويحتوي على مستويات عالية من الدهون السفينغولية والكوليسترول التي تمنحه صلابة مميزة مقارنة بالأغشية الأخرى في الخلية24. يجب مراعاة عدم تناسق الوريقات للأغشية مثل PM ، التي تحتوي على نشرة خارجية غنية بالسفينغوميلين ، و PC ، والكوليسترول ، ونشرة داخلية غنية ب PE و PI و PS التي تعتبر مهمة للإشارة إلى الشلالات24. أخيرا ، يدفع تنوع الدهون أيضا إلى تكوين مجالات دقيقة تختلف في التعبئة والنظام الداخلي ، والمعروفة باسم طوافات الدهون24,26 ؛ تظهر هذه عدم التماثل الجانبي ، ويفترض أنها تلعب أدوارا مهمة في الإشارات الخلوية26 ، ويصعب دراستها بسبب طبيعتها العابرة.
تم استخدام التقنيات التجريبية مثل التنظير الفلوري والتحليل الطيفي وأنظمة الأغشية النموذجية مثل الحويصلات أحادية الصفيحة العملاقة (GUVs) للتحقيق في تفاعلات الجزيئات الحيوية مع الأغشية. ومع ذلك ، من الصعب التقاط الطبيعة المعقدة والديناميكية للمكونات المعنية بالطرق التجريبية وحدها. على سبيل المثال ، هناك قيود على تصوير المجالات عبر الغشاء للبروتينات ، وتعقيد الأغشية المستخدمة في مثل هذه الدراسات ، وتحديد الحالات الوسيطة أو العابرة أثناء عملية الاهتمام27،28،29. منذ ظهور المحاكاة الجزيئية للطبقات الأحادية الدهنية والطبقات الثنائية في ثمانينيات القرن العشرين29 ، يمكن الآن تحديد أنظمة البروتين الدهني وتفاعلاتها على المستوى الجزيئي. محاكاة الديناميات الجزيئية (MD) هي تقنية حسابية شائعة تتنبأ بحركة الجسيمات بناء على قواها بين الجزيئات. يصف جهد التفاعل الإضافي التفاعلات المرتبطة وغير المرتبطة بين جسيمات النظام30. تسمى مجموعة المعلمات المستخدمة لنمذجة هذه التفاعلات حقل قوة المحاكاة (FF). يتم الحصول على هذه المعلمات من حسابات ab initio ، والحسابات شبه التجريبية ، وميكانيكا الكم ، والأمثل للبيانات المستنسخة من تجارب الأشعة السينية وحيود الإلكترون ، والرنين المغناطيسي النووي ، والأشعة تحت الحمراء ، والتحليل الطيفي للنيوترونات ، من بين طرق أخرى31.
يمكن استخدام محاكاة MD لدراسة الأنظمة على مستويات مختلفة من الدقة32،33،34. تتم دراسة الأنظمة التي تهدف إلى توصيف تفاعلات جزيئية حيوية محددة ، وروابط هيدروجينية ، وتفاصيل أخرى عالية الدقة باستخدام محاكاة جميع الذرات (AA). في المقابل ، تقوم عمليات المحاكاة الخشنة (CG) بتجميع الذرات في مجموعات وظيفية أكبر لتقليل التكلفة الحسابية وفحص ديناميكيات النطاقالأكبر 33. تقع بين هذين المحاكاة للذرة المتحدة (UA) ، حيث يتم دمج ذرات الهيدروجين مع ذراتها الثقيلة لتسريع الحساب33,35. تعد محاكاة MD أداة قوية لاستكشاف ديناميكيات الأغشية الدهنية وتفاعلاتها مع الجزيئات الأخرى ويمكن أن تعمل على توفير آليات المستوى الجزيئي للعمليات ذات الأهمية في واجهة الغشاء. بالإضافة إلى ذلك ، يمكن أن تعمل محاكاة MD على تضييق نطاق الأهداف التجريبية والتنبؤ بالخصائص الجزيئية الكبيرة لنظام معين بناء على التفاعلات المجهرية.
باختصار ، بالنظر إلى مجموعة من الإحداثيات الأولية والسرعات ومجموعة من الشروط مثل درجة الحرارة والضغط الثابتين ، يتم حساب مواضع وسرعات كل جسيم من خلال التكامل العددي لجهد التفاعل وقانون نيوتن للحركة. يتكرر هذا بشكل متكرر ، وبالتالي توليد مسار محاكاة30. يتم إجراء هذه الحسابات باستخدام محرك MD ؛ من بين العديد من الحزم مفتوحة المصدر ، يعد GROMACS36 أحد أكثر المحركات استخداما والذي وصفناه هنا. ويشمل أيضا أدوات لتحليل وبناء الإحداثيات الأولية للنظم المراد محاكاتها37. تشمل محركات MD الأخرى NAMD38. CHARMM39 و AMBER40 ، والتي يمكن للمستخدم اختيارها وفقا لتقديره الخاص بناء على الأداء الحسابي لنظام معين. من الأهمية بمكان تصور المسارات أثناء المحاكاة وكذلك لتحليل النتائج وتفسيرها. تتوفر مجموعة متنوعة من الأدوات. نناقش هنا الديناميات الجزيئية المرئية (VMD) التي تقدم مجموعة واسعة من الميزات ، بما في ذلك التصور ثلاثي الأبعاد (3-D) مع طرق الرسم والتلوين الموسعة ، وتصور البيانات الحجمي ، وبناء وإعداد وتحليل مسارات أنظمة محاكاة MD ، وصنع فيلم المسار بدون حدود لحجم النظام ، إذا كانت الذاكرة متوفرة41،42،43.
تتأثر دقة الديناميكيات المتوقعة بين مكونات النظام بشكل مباشر ب FF المختار لنشر المسار. يتم متابعة جهود معلمات FF التجريبية من قبل عدد قليل من مجموعات البحث. تشمل FF الأكثر رسوخا وشيوعا ل MD CHARMM39 و AMBER 40 و Martini44 و OPLS 45 و SIRAH 46. يستخدم مجال القوة المضافة لجميع الذرات CHARMM36 (C36)47 على نطاق واسع ل AA MD لأنظمة الأغشية لأنه يعيد إنتاج البيانات الهيكلية التجريبية بدقة. تم تطويره في الأصل من قبل مجتمع CHARMM ، وهو متوافق مع العديد من محركات MD مثل GROMACS و NAMD. على الرغم من التحسينات عبر FFs الشائعة ، هناك جهد مستمر لتحسين مجموعات المعلمات للسماح بالتنبؤات التي تعيد إنتاج الملاحظات التجريبية عن كثب ، مدفوعة بالمصالح في أنظمة معينة للدراسة48,49.
يتمثل التحدي عند محاكاة الأغشية الدهنية في تحديد طول مسار المحاكاة. يعتمد هذا إلى حد كبير على المقاييس التي سيتم تحليلها والعملية التي يهدف المرء إلى توصيفها. عادة ما تتطلب مخاليط الليبيدات المعقدة وقتا أطول للوصول إلى الاتزان؛ حيث يجب أن يكون لدى المزيد من الأنواع وقت كاف للانتشار على مستوى الغشاء والوصول إلى تنظيم جانبي مستقر. يقال إن المحاكاة تكون في حالة توازن عندما تصل خاصية الاهتمام إلى هضبة وتتقلب حول قيمة ثابتة. من الممارسات الشائعة الحصول على ما لا يقل عن 100-200 نانوثانية من المسار المتوازن لإجراء تحليل إحصائي مناسب على الخصائص والتفاعلات ذات الاهتمام. من الشائع إجراء عمليات محاكاة غشائية فقط بين 200-500 نانوثانية ، اعتمادا على مدى تعقيد خليط الدهون وسؤال البحث. تتطلب تفاعلات البروتين والدهون عادة أوقات محاكاة أطول ، بين 500-2000 نانوثانية. وفيما يلي بعض النهج لتسريع أخذ العينات والديناميات التي يمكن ملاحظتها باستخدام النظم الغشائية: ‘1’ نموذج المحاكاة الغشائية العالية الحركة، الذي يستعيض عن الكربونات الطرفية للدهون في الغشاء بالمذيب العضوي لتسريع أخذ العينات 50؛ و ‘2’ نموذج المحاكاة الغشائية العالية الحركة، الذي يستعيض عن الكربونات الطرفية للدهون في الغشاء بالمذيب العضوي لتسريع أخذ العينات 50؛ و ‘2’ نموذج المحاكاة الغشائية العالية الحركة، الذي يستعيض عن الكربونات الطرفية للدهون في الغشاء بالمذيب العضوي لتسريع أخذ العينات50؛ و ‘2’ نموذج المحاكاة الغشائية العالية الحركة، الذي يستعيض عن الكربونات الغشائية العالية الحركة (HMMM)، الذي يستعيض عن و (ii) إعادة تقسيم كتلة الهيدروجين (HMR) ، والذي يجمع جزءا صغيرا من كتل الذرات الثقيلة داخل النظام مع تلك الخاصة بذرات الهيدروجين للسماح باستخدام محاكاة أكبر للخطوةالزمنية 51.
يناقش البروتوكول التالي نهجا صديقا للمبتدئين لبناء وتشغيل وتحليل نماذج الأغشية الواقعية باستخدام AA MD. نظرا لطبيعة محاكاة MD ، يجب تشغيل مسارات متعددة لمراعاة قابلية التكرار والتحليل الإحصائي المناسب للنتائج. من الممارسات الحالية تشغيل ثلاث نسخ متماثلة على الأقل لكل نظام من أنظمة الاهتمام. بمجرد اختيار الأنواع الليبيدة للكائن الحي والعملية محل الاهتمام، يتم توضيح وتلخيص الخطوات الأساسية لبناء مسار محاكاة لنظام الغشاء فقط وتشغيله وتحليله.
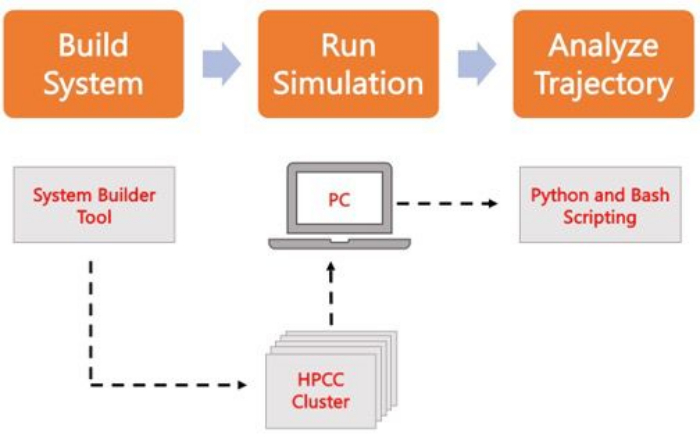
الشكل 2. تخطيطي لتشغيل محاكاة MD. تتوافق المربعات البرتقالية مع الخطوات الرئيسية الثلاث الموضحة في البروتوكول. يوجد تحتها سير عمل عملية المحاكاة. أثناء إعداد النظام ، يتم بناء النظام الذي يحتوي على الإحداثيات الأولية لنظام الغشاء المذاب باستخدام مولد إدخال النظام مثل CHARMM-GUI Membrane Builder. بعد نقل ملفات الإدخال إلى مجموعة حوسبة عالية الأداء ، يتم نشر مسار المحاكاة باستخدام محرك MD ، مثل GROMACS. يمكن إجراء تحليل المسار على مجموعة الكمبيوتر أو محطة عمل محلية جنبا إلى جنب مع التصور. ثم يتم إجراء التحليل ، باستخدام الحزم التي تحتوي على كود تحليل مدمج مثل GROMACS و VMD ، أو باستخدام نصوص Bash أو مكتبات Python المختلفة. يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
Protocol
Representative Results
Discussion
يمكن للتقنيات التجريبية تصور الجزيئات الحيوية بدقة عالية باستخدام المجهر الإلكتروني المبرد (cryo-EM)58 ، وتقنيات التألق ، ومجهر القوة الذرية (AFM)59. ومع ذلك ، من الصعب التقاط التفاعل وديناميكيات التفاعلات الجزيئية التي تكمن وراء المسارات البيولوجية ، والتسبب في المرض…
Disclosures
The authors have nothing to disclose.
Acknowledgements
يشكر المؤلفون جينهوي لي وريكاردو إكس راميريز على مسارات المحاكاة والمناقشات أثناء كتابة هذه المخطوطة. تم دعم O.C. من قبل زمالة الجامعة في بوفالو الرئاسية ومبادرة المعهد الوطني للصحة لتعظيم منحة التدريب على تطوير الطلاب 1T32GM144920-01 الممنوحة لمارغريتا إل دوبوكوفيتش (PI).
Materials
| Anaconda3 | Anaconda Inc (Python & related libraries) | N/A | |
| CHARMM-GUI.org | Im lab, Lehigh University | N/A | |
| GROMACS | GROMACS development team | N/A | |
| Linux HPC Cluster | UB CCR | N/A | |
| MATLAB | MathWorks | N/A | |
| VMD | Theoretical and Computational Biophysics Group | N/A |
References
- Vanni, S., Riccardi, L., Palermo, G., De Vivo, M. Structure and Dynamics of the Acyl Chains in the Membrane Trafficking and Enzymatic Processing of Lipids. Accounts of Chemical Research. 52 (11), 3087-3096 (2019).
- Harayama, T., Riezman, H. Understanding the diversity of membrane lipid composition. Nature Reviews Molecular Cell Biology. 19 (5), 281-296 (2018).
- Tanaka, M., Edward, H. .. . E. g. e. l. m. a. n. . Comprehensive Biophysics. , 261-272 (2012).
- Bruce Alberts, A. J., Julian Lewis, , Martin Raff, , Keith Roberts, , Peter Walter, . Molecular Biology of the Cell. , (2002).
- Watson, H. Biological membranes. Essays in Biochemistry. 59, 43-69 (2015).
- Coskun, &. #. 2. 2. 0. ;., Simons, K. Cell Membranes: The Lipid Perspective. Structure. 19 (11), 1543-1548 (2011).
- Douglas G, H. a. y. e. s., Daniel, K. Y., Solaiman, D., Richard, . Biobased Surfactants (Second Edition) eds. , 515-529 (2019).
- González-Rubio, P., Gautier, R., Etchebest, C., Fuchs, P. F. J. Amphipathic-Lipid-Packing-Sensor interactions with lipids assessed by atomistic molecular dynamics. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1808, 2119-2127 (2011).
- Halbleib, K., et al. Activation of the Unfolded Protein Response by Lipid Bilayer Stress. Molecular Cell. 67, 673-684 (2017).
- Andreasen, M., Lorenzen, N., Otzen, D. Interactions between misfolded protein oligomers and membranes: A central topic in neurodegenerative diseases. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1848 (9), 1897-1907 (2015).
- Calianese, D. C., Birge, R. B. Biology of phosphatidylserine (PS): basic physiology and implications in immunology, infectious disease, and cancer. Cell Commununication and Signaling. 18 (1), 41 (2020).
- Nieto-Garai, J. A., Contreras, F. X., Arboleya, A., Lorizate, M. Role of Protein-Lipid Interactions in Viral Entry. Advanced Biology. 6, 2101264 (2022).
- Mazzon, M., Mercer, J. Lipid interactions during virus entry and infection. Cell Microbiology. 16, 1493-1502 (2014).
- Colombelli, C., Aoun, M., Tiranti, V. Defective lipid metabolism in neurodegeneration with brain iron accumulation (NBIA) syndromes: not only a matter of iron. Journal of Inherited Metabolic Disease. 38 (1), 123-136 (2015).
- Saini-Chohan, H. K., Mitchell, R. W., Vaz, F. M., Zelinski, T., Hatch, G. M. Delineating the role of alterations in lipid metabolism to the pathogenesis of inherited skeletal and cardiac muscle disorders: Thematic Review Series: Genetics of Human Lipid Diseases. Journal of Lipid Research. 53 (1), 4-27 (2012).
- Martinotti, C., Ruiz-Perez, L., Deplazes, E., Mancera, R. L. Molecular Dynamics Simulation of Small Molecules Interacting with Biological Membranes. ChemPhysChem. 21 (14), 1486-1514 (2020).
- Li, J., Kalyanram, P., Rozati, S., Monje-Galvan, V., Gupta, A. Interaction of Cyanine-D112 with Binary Lipid Mixtures: Molecular Dynamics Simulation and Differential Scanning Calorimetry Study. ACS Omega. 7 (11), 9765-9774 (2022).
- Nagy, L., et al. Protein/Lipid Interaction in the Bacterial Photosynthetic Reaction Center: Phosphatidylcholine and Phosphatidylglycerol Modify the Free Energy Levels of the Quinones. 生物化学. 43 (40), 12913-12923 (2004).
- Ramirez, R. X., Campbell, O., Pradhan, A. J., Atilla-Gokcumen, G. E., Monje-Galvan, V. Modeling the molecular fingerprint of protein-lipid interactions of MLKL on complex bilayers. Frontiers in Chemistry. 10, (2023).
- Dondelinger, Y., et al. MLKL Compromises Plasma Membrane Integrity by Binding to Phosphatidylinositol Phosphates. Cell Reports. 7 (4), 971-981 (2014).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews Molecular Cell Biology. 9 (2), 112-124 (2008).
- van Meer, G., de Kroon, A. I. P. M. Lipid map of the mammalian cell. Journal of Cell Science. 124 (1), 5 (2011).
- Lee, H. R., Lee, G. Y., You, D. G., Kim, H. K., Young, D. Y. Hepatitis C virus p7 induces membrane permeabilization by interacting with phosphatidylserine. International Journal of Molecular Sciences. 21 (3), 897 (2020).
- Casares, D., Escribá, P. V., Rosselló, C. A. Membrane Lipid Composition: Effect on Membrane and Organelle Structure, Function and Compartmentalization and Therapeutic Avenues. International Journal of Molecular Sciences. 20 (9), 2167 (2019).
- Marrink, S. J., et al. Computational Modeling of Realistic Cell Membranes. Chemical Reviews. 119 (9), 6184-6226 (2019).
- Janmey, P. A., Kinnunen, P. K. J. Biophysical properties of lipids and dynamic membranes. Trends in Cell Biology. 16 (10), 538-546 (2006).
- Brémaud, E., Favard, C., Muriaux, D. Deciphering the Assembly of Enveloped Viruses Using Model Lipid Membranes. Membranes. 12, 441 (2022).
- Campbell, O., Monje-Galvan, V. Protein-driven membrane remodeling: Molecular perspectives from Flaviviridae infections. Biophysical Journal. 122 (11), 1890-1899 (2022).
- Loschwitz, J., Olubiyi, O. O., Hub, J. S., Strodel, B., Poojari, C. S. Computer simulations of protein-membrane systems. Progress in molecular biology and translational science. 170, 273-403 (2020).
- Shell, M. S., Scott Shell, M. . Thermodynamics and Statistical Mechanics: An Integrated ApproachCambridge Series in Chemical Engineering. , 21-49 (2015).
- Yang, J., et al. Molecular Dynamic Simulation of Ni-Al Alloy-H2O Reactions Using the ReaxFF Reactive Force Field. ACS Omega. 8 (11), 9807-9814 (2023).
- Ingólfsson, H. I., Arnarez, C., Periole, X., Marrink, S. J. Computational ‘microscopy’ of cellular membranes. Journal of Cell Science. 129 (2), 257-268 (2016).
- Klauda, J. B. Perspective: Computational modeling of accurate cellular membranes with molecular resolution. The Journal of Chemical Physics. 149 (22), 220901 (2018).
- Chavent, M., Duncan, A. L., Sansom, M. S. P. Molecular dynamics simulations of membrane proteins and their interactions: from nanoscale to mesoscale. Current Opinion in Structural Biology. 40, 8-16 (2016).
- Khakbaz, P., Monje-Galvan, V., Zhuang, X., Klauda, J. B., Otto Geiger, . Biogenesis of Fatty Acids, Lipids and Membranes. , 1-19 (2017).
- Abraham, M. J., et al. GROMACS: High performance molecular simulations through multi-level parallelism from laptops to supercomputers. SoftwareX. 1, 19-25 (2015).
- Lemkul, J. A. From Proteins to Perturbed Hamiltonians: A Suite of Tutorials for the GROMACS-2018 Molecular Simulation Package. Living Journal of Computational Molecular Science. 1 (1), 5068 (2018).
- Phillips, J. C., et al. Scalable molecular dynamics on CPU and GPU architectures with NAMD. The Journal of Chemical Physics. 153 (4), 044130 (2020).
- Klauda, J. B., et al. Update of the CHARMM All-Atom Additive Force Field for Lipids: Validation on Six Lipid Types. The Journal of Physical Chemistry B. 114 (23), 7830-7843 (2010).
- Wang, J., Wolf, R. M., Caldwell, J. W., Kollman, P. A., Case, D. A. Development and testing of a general amber force field. Journal of Computational Chemistry. 25 (9), 1157-1174 (2004).
- John Stone, A. A., et al. . Using VMD. , (2011).
- Humphrey, W., Dalke, A., Schulten, K. VMD: Visual molecular dynamics. Journal of Molecular Graphics. 14 (1), 33-38 (1996).
- Hsin, J., Arkhipov, A., Yin, Y., Stone, J. E., Schulten, K. Using VMD: An Introductory Tutorial. Current Protocols in Bioinformatics. 24 (1), 5.7.1-5.7.48 (2008).
- Souza, P. C. T., et al. Martini 3: a general purpose force field for coarse-grained molecular dynamics. Nature Methods. 18 (4), 382-388 (2021).
- Jorgensen, W. L., Maxwell, D. S., Tirado-Rives, J. Development and Testing of the OPLS All-Atom Force Field on Conformational Energetics and Properties of Organic Liquids. Journal of the American Chemical Society. 118 (45), 11225-11236 (1996).
- Machado, M. R., et al. The SIRAH 2.0 Force Field: Altius, Fortius, Citius. Journal of Chemical Theory and Computation. 15 (4), 2719-2733 (2019).
- Huang, J., et al. CHARMM36m: an improved force field for folded and intrinsically disordered proteins. Nature Methods. 14 (1), 71-73 (2017).
- Mu, J., Liu, H., Zhang, J., Luo, R., Chen, H. F. Recent Force Field Strategies for Intrinsically Disordered Proteins. Journal of Chemical Information and Modeling. 61 (3), 1037-1047 (2021).
- Inakollu, V. S. S., Geerke, D. P., Rowley, C. N., Yu, H. Polarisable force fields: what do they add in biomolecular simulations. Current Opinion in Structural Biology. 61, 182-190 (2020).
- Ohkubo, Y. Z., et al. Accelerating Membrane Insertion of Peripheral Proteins with a Novel Membrane Mimetic Model. Biophysical Journal. 102 (9), 2130-2139 (2012).
- Hopkins, C. W., Le Grand, S., Walker, R. C., Roitberg, A. E. Long-Time-Step Molecular Dynamics through Hydrogen Mass Repartitioning. Journal of Chemical Theory and Computation. 11 (4), 1864-1874 (2015).
- Park, S., Beaven, A. H., Klauda, J. B., Im, W. How Tolerant are Membrane Simulations with Mismatch in Area per Lipid between Leaflets. Journal of Chemical Theory and Computation. 11 (7), 3466-3477 (2015).
- Park, S., Im, W., Pastor, R. W. Developing initial conditions for simulations of asymmetric membranes: a practical recommendation. Biophysical Journal. 120 (22), 5041-5059 (2021).
- Wu, E. L., et al. CHARMM-GUI Membrane Builder toward realistic biological membrane simulations. Journal of Computational Chemistry. 35 (27), 1997-2004 (2014).
- Center for Computational Research, U.a.B.. . CCR Facility Description. , (2019).
- Piggot, T. J., Allison, J. R., Sessions, R. B., Essex, J. W. On the Calculation of Acyl Chain Order Parameters from Lipid Simulations. Journal of Chemical Theory and Computation. 13 (11), 5683-5696 (2017).
- Li, J., Monje-Galvan, V. Effect of Glycone Diversity on the Interaction of Triterpenoid Saponins and Lipid Bilayers. ACS Applied Bio Materials. , (2023).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews Drug Discovery. 17 (7), 471-492 (2018).
- Ando, T., Uchihashi, T., Kodera, N. High-Speed AFM and Applications to Biomolecular Systems. Annual Review of Biophysics. 42 (1), 393-414 (2013).
- Martínez, L., Andrade, R., Birgin, E. G., Martínez, J. M. PACKMOL: A package for building initial configurations for molecular dynamics simulations. Journal of Computational Chemistry. 30 (13), 2157-2164 (2009).
- Jewett, A. I., et al. Moltemplate: A Tool for Coarse-Grained Modeling of Complex Biological Matter and Soft Condensed Matter Physics. Journal of Molecular Biology. 433 (11), 166841 (2021).
- Jo, S., Kim, T., Iyer, V. G., Im, W. CHARMM-GUI: A web-based graphical user interface for CHARMM. Journal of Computational Chemistry. 29 (11), 1859-1865 (2008).
- Polêto, M. D., Lemkul, J. A. Integration of experimental data and use of automated fitting methods in developing protein force fields. Communications Chemistry. 5 (1), 38 (2022).
- Hynninen, A. P., Crowley, M. F. New faster CHARMM molecular dynamics engine. Journal of Computational Chemistry. 35 (5), 406-413 (2014).
- Kim, S. Issues on the Choice of a Proper Time Step in Molecular Dynamics. Physics Procedia. 53, 60-62 (2014).
- Grubmüller, H., Heller, H., Windemuth, A., Schulten, K. Generalized Verlet Algorithm for Efficient Molecular Dynamics Simulations with Long-range Interactions. Molecular Simulation. 6 (1-3), 121-142 (1991).
- Darden, T., York, D., Pedersen, L. Particle mesh Ewald: An N·log(N) method for Ewald sums in large systems. Journal of Chemical Physics. 98 (12), 10089-10092 (1993).
- . . Hepatitis C. , (2021).
- Braun, E., et al. Best Practices for Foundations in Molecular Simulations [Article v1.0]. Living Journal of Computational Molecular Science. 1 (1), 5957 (2018).
- Moradi, S., Nowroozi, A., Shahlaei, M. Shedding light on the structural properties of lipid bilayers using molecular dynamics simulation: a review study. RSC Advances. 9 (8), 4644-4658 (2019).
- Monje-Galvan, V., Klauda, J. B. Modeling Yeast Organelle Membranes and How Lipid Diversity Influences Bilayer Properties. 生物化学. 54 (45), 6852-6861 (2015).
- Michaud-Agrawal, N., Denning, E. J., Woolf, T. B., Beckstein, O. MDAnalysis: A toolkit for the analysis of molecular dynamics simulations. Journal of Computational Chemistry. 32 (10), 2319-2327 (2011).
- Gowers, R., et al. MDAnalysis: A Python Package for the Rapid Analysis of Molecular Dynamics Simulations. SciPy. , (2016).
- McGibbon, R. o. b. e. r. t. . T., et al. MDTraj: A Modern Open Library for the Analysis of Molecular Dynamics Trajectories. Biophysical Journal. 109 (8), 1528-1532 (2015).
- Fortunato, M. E., Colina, C. M. pysimm: A python package for simulation of molecular systems. SoftwareX. 6, 7-12 (2017).
- Scherer, M. K., et al. PyEMMA 2: A Software Package for Estimation, Validation, and Analysis of Markov Models. Journal of Chemical Theory and Computation. 11 (11), 5525-5542 (2015).
- Song, W., et al. PyLipID: A Python Package for Analysis of Protein-Lipid Interactions from Molecular Dynamics Simulations. Journal of Chemical Theory and Computation. 18 (2), 1188-1201 (2022).
- Monje-Galvan, V., Klauda, J. B. Peripheral membrane proteins: Tying the knot between experiment and computation. Biochimica et Biophysica Acta (BBA) – Biomembranes. 1858 (7, Part B), 1584-1593 (2016).
- Monje-Galvan, V., Voth, G. A. Binding mechanism of the matrix domain of HIV-1 gag on lipid membranes. eLife. 9, e58621 (2020).
- Wang, B., Guo, C. Concentration-Dependent Effects of Cholesterol on the Dimerization of Amyloid-β Peptides in Lipid Bilayers. ACS Chemical Neuroscience. 13 (18), 2709-2718 (2022).

